+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-5844 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Subtomogram average of a virus-associated pyramid | |||||||||
 マップデータ マップデータ | Reconstruction of a virus associated pyramid | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | VAP / archaeovirus / archaea / SIRV2 / Sulfolobus islandicus / viral egress | |||||||||
| 機能・相同性 | : / membrane / Uncharacterized protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Sulfolobus islandicus rod-shaped virus 2 (ウイルス) Sulfolobus islandicus rod-shaped virus 2 (ウイルス) | |||||||||
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 | |||||||||
 データ登録者 データ登録者 | Daum B / Kuehlbrandt W | |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2014 ジャーナル: Proc Natl Acad Sci U S A / 年: 2014タイトル: Self-assembly of the general membrane-remodeling protein PVAP into sevenfold virus-associated pyramids. 著者: Bertram Daum / Tessa E F Quax / Martin Sachse / Deryck J Mills / Julia Reimann / Özkan Yildiz / Sabine Häder / Cosmin Saveanu / Patrick Forterre / Sonja-Verena Albers / Werner Kühlbrandt / ...著者: Bertram Daum / Tessa E F Quax / Martin Sachse / Deryck J Mills / Julia Reimann / Özkan Yildiz / Sabine Häder / Cosmin Saveanu / Patrick Forterre / Sonja-Verena Albers / Werner Kühlbrandt / David Prangishvili /  要旨: Viruses have developed a wide range of strategies to escape from the host cells in which they replicate. For egress some archaeal viruses use a pyramidal structure with sevenfold rotational symmetry. ...Viruses have developed a wide range of strategies to escape from the host cells in which they replicate. For egress some archaeal viruses use a pyramidal structure with sevenfold rotational symmetry. Virus-associated pyramids (VAPs) assemble in the host cell membrane from the virus-encoded protein PVAP and open at the end of the infection cycle. We characterize this unusual supramolecular assembly using a combination of genetic, biochemical, and electron microscopic techniques. By whole-cell electron cryotomography, we monitored morphological changes in virus-infected host cells. Subtomogram averaging reveals the VAP structure. By heterologous expression of PVAP in cells from all three domains of life, we demonstrate that the protein integrates indiscriminately into virtually any biological membrane, where it forms sevenfold pyramids. We identify the protein domains essential for VAP formation in PVAP truncation mutants by their ability to remodel the cell membrane. Self-assembly of PVAP into pyramids requires at least two different, in-plane and out-of-plane, protein interactions. Our findings allow us to propose a model describing how PVAP arranges to form sevenfold pyramids and suggest how this small, robust protein may be used as a general membrane-remodeling system. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_5844.map.gz emd_5844.map.gz | 2.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-5844-v30.xml emd-5844-v30.xml emd-5844.xml emd-5844.xml | 10.3 KB 10.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_5844_1.jpg emd_5844_1.jpg emd_5844_2.tif emd_5844_2.tif | 124.9 KB 2.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-5844 http://ftp.pdbj.org/pub/emdb/structures/EMD-5844 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5844 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5844 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_5844_validation.pdf.gz emd_5844_validation.pdf.gz | 78.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_5844_full_validation.pdf.gz emd_5844_full_validation.pdf.gz | 77.5 KB | 表示 | |
| XML形式データ |  emd_5844_validation.xml.gz emd_5844_validation.xml.gz | 499 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5844 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5844 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5844 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5844 | HTTPS FTP |
-関連構造データ
| 類似構造データ |
|---|
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_5844.map.gz / 形式: CCP4 / 大きさ: 3.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_5844.map.gz / 形式: CCP4 / 大きさ: 3.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of a virus associated pyramid | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 8.663 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
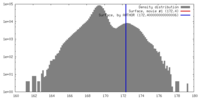
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Reconstruction of a virus-associated pyramid induced by overexpre...
| 全体 | 名称: Reconstruction of a virus-associated pyramid induced by overexpressing the viral protein PVAP in E. coli |
|---|---|
| 要素 |
|
-超分子 #1000: Reconstruction of a virus-associated pyramid induced by overexpre...
| 超分子 | 名称: Reconstruction of a virus-associated pyramid induced by overexpressing the viral protein PVAP in E. coli タイプ: sample / ID: 1000 / Number unique components: 1 |
|---|
-分子 #1: protein forming virus-associated pyramids
| 分子 | 名称: protein forming virus-associated pyramids / タイプ: protein_or_peptide / ID: 1 / Name.synonym: P98, PVAP 詳細: The gene encoding PVAP was heterologously overexpressed in E. coli to incorporate pyramid-shaped multimers of PVAP into the plasma membrane. 集合状態: Multimer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Sulfolobus islandicus rod-shaped virus 2 (ウイルス) Sulfolobus islandicus rod-shaped virus 2 (ウイルス)細胞中の位置: Plasma membrane |
| 分子量 | 実験値: 10 KDa / 理論値: 10 KDa |
| 組換発現 | 生物種:  |
| 配列 | UniProtKB: Uncharacterized protein / GO: GO: 0951408 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7 / 詳細: 50 mM Tris, 300 mM NaCl |
|---|---|
| グリッド | 詳細: Quantifoil 300 mesh R2/2, glow-discharged |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 10 % / 装置: HOMEMADE PLUNGER 手法: Blot for 3-5 seconds from the side where the sample was applied. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 温度 | 最低: 89 K / 最高: 96 K / 平均: 90 K |
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 40,000 times magnification |
| 特殊光学系 | エネルギーフィルター - 名称: Gatan Tridium GIF |
| 日付 | 2012年3月12日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN ULTRASCAN 1000 (2k x 2k) ビット/ピクセル: 32 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 16870 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.2 mm / 最大 デフォーカス(公称値): 10.0 µm / 最小 デフォーカス(公称値): 8.0 µm / 倍率(公称値): 27500 |
| 試料ステージ | 試料ホルダー: liquid nitrogen cooled / 試料ホルダーモデル: OTHER / Tilt series - Axis1 - Min angle: -60.73 ° / Tilt series - Axis1 - Max angle: 59.20 ° |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | ソフトウェア - 名称: PEET, IMOD / 使用したサブトモグラム数: 57 |
|---|
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)