+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-5802 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Electron crystallography of AQP0 with anionic phospholipids. | |||||||||
 マップデータ マップデータ | 3D reconstruction of AQP0_DMPG 2D crystal in p4212 form | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | aquaporin-0 / AQP0 / DMPG / phosphatidylglycerol / DMPS / phosphatidylserine / anionic phospholipids / electron crystallography | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報maintenance of lens transparency / cell adhesion mediator activity / homotypic cell-cell adhesion / gap junction-mediated intercellular transport / water transport / water channel activity / structural constituent of eye lens / lens development in camera-type eye / anchoring junction / visual perception ...maintenance of lens transparency / cell adhesion mediator activity / homotypic cell-cell adhesion / gap junction-mediated intercellular transport / water transport / water channel activity / structural constituent of eye lens / lens development in camera-type eye / anchoring junction / visual perception / calmodulin binding / apical plasma membrane / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 電子線結晶学 / 解像度: 7.0 Å | |||||||||
 データ登録者 データ登録者 | Hite RK / Chiu P-L / Schuller J / Walz T | |||||||||
 引用 引用 |  ジャーナル: PLoS One / 年: 2015 ジャーナル: PLoS One / 年: 2015タイトル: Effect of lipid head groups on double-layered two-dimensional crystals formed by aquaporin-0. 著者: Richard Kevin Hite / Po-Lin Chiu / Jan Michael Schuller / Thomas Walz /  要旨: Aquaporin-0 (AQP0) is a lens-specific water channel that also forms membrane junctions. Reconstitution of AQP0 with dimyristoyl phosphatidylcholine (DMPC) and E. coli polar lipids (EPL) yielded well- ...Aquaporin-0 (AQP0) is a lens-specific water channel that also forms membrane junctions. Reconstitution of AQP0 with dimyristoyl phosphatidylcholine (DMPC) and E. coli polar lipids (EPL) yielded well-ordered, double-layered two-dimensional (2D) crystals that allowed electron crystallographic structure determination of the AQP0-mediated membrane junction. The interacting tetramers in the two crystalline layers are exactly in register, resulting in crystals with p422 symmetry. The high-resolution density maps also allowed modeling of the annular lipids surrounding the tetramers. Comparison of the DMPC and EPL bilayers suggested that the lipid head groups do not play an important role in the interaction of annular lipids with AQP0. We now reconstituted AQP0 with the anionic lipid dimyristoyl phosphatidylglycerol (DMPG), which yielded a mixture of 2D crystals with different symmetries. The different crystal symmetries result from shifts between the two crystalline layers, suggesting that the negatively charged PG head group destabilizes the interaction between the extracellular AQP0 surfaces. Reconstitution of AQP0 with dimyristoyl phosphatidylserine (DMPS), another anionic lipid, yielded crystals that had the usual p422 symmetry, but the crystals showed a pH-dependent tendency to stack through their cytoplasmic surfaces. Finally, AQP0 failed to reconstitute into membranes that were composed of more than 40% dimyristoyl phosphatidic acid (DMPA). Hence, although DMPG, DMPS, and DMPA are all negatively charged lipids, they have very different effects on AQP0 2D crystals, illustrating the importance of the specific lipid head group chemistry beyond its mere charge. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_5802.map.gz emd_5802.map.gz | 57.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-5802-v30.xml emd-5802-v30.xml emd-5802.xml emd-5802.xml | 10.6 KB 10.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  400_5802.gif 400_5802.gif 80_5802.gif 80_5802.gif | 117 KB 7.7 KB | ||
| Filedesc structureFactors |  emd_5802_sf.cif.gz emd_5802_sf.cif.gz | 65.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-5802 http://ftp.pdbj.org/pub/emdb/structures/EMD-5802 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5802 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5802 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  5803C C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_5802.map.gz / 形式: CCP4 / 大きさ: 120.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_5802.map.gz / 形式: CCP4 / 大きさ: 120.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3D reconstruction of AQP0_DMPG 2D crystal in p4212 form | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X: 0.3275 Å / Y: 0.3275 Å / Z: 1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
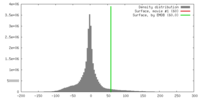
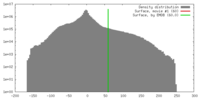
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 90 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : 2D crystal structure of AQP0 (Ovis aries) with DMPG lipid in p421...
| 全体 | 名称: 2D crystal structure of AQP0 (Ovis aries) with DMPG lipid in p4212 form |
|---|---|
| 要素 |
|
-超分子 #1000: 2D crystal structure of AQP0 (Ovis aries) with DMPG lipid in p421...
| 超分子 | 名称: 2D crystal structure of AQP0 (Ovis aries) with DMPG lipid in p4212 form タイプ: sample / ID: 1000 / 詳細: 2D crystals / 集合状態: Tetramer / Number unique components: 1 |
|---|---|
| 分子量 | 理論値: 113 KDa |
-分子 #1: Aquaporin-0
| 分子 | 名称: Aquaporin-0 / タイプ: protein_or_peptide / ID: 1 / Name.synonym: AQP0 詳細: Aquaporin-0 was crystallized with dimyristoylglycerol (DMPG) in p4212 symmetry. コピー数: 32 / 集合状態: Tetramer / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種:  |
| 配列 | UniProtKB: Lens fiber major intrinsic protein |
-実験情報
-構造解析
 解析 解析 | 電子線結晶学 |
|---|---|
| 試料の集合状態 | 2D array |
- 試料調製
試料調製
| 緩衝液 | pH: 6 / 詳細: 10 mM MES, 50 mM Mg2Cl2, 150 mM NaCl, 0.05% NaN3 |
|---|---|
| グリッド | 詳細: Carbon sandwich method combined with trehalose embedding |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
| 詳細 | Dialysis |
| 結晶化 | 詳細: Dialysis |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 温度 | 最低: 90 K / 最高: 100 K / 平均: 95 K |
| 日付 | 2012年3月31日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) デジタル化 - サンプリング間隔: 15 µm / 実像数: 227 / 平均電子線量: 20 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 80000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 1.6 mm / 最大 デフォーカス(公称値): 0.65 µm / 最小 デフォーカス(公称値): 0.35 µm / 倍率(公称値): 52000 |
| 試料ステージ | 試料ホルダー: liquid-nitrogen cooled / 試料ホルダーモデル: OTHER |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | Crystal images were unbent and reconstructed using 2DX and MRC software. |
|---|---|
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 7.0 Å / 解像度の算出法: DIFFRACTION PATTERN/LAYERLINES / ソフトウェア - 名称: 2DX, and, MRC / 詳細: The reconstruction map shows in 2x2 unit cells. |
| 結晶パラメータ | 単位格子 - A: 65.5 Å / 単位格子 - B: 65.5 Å / 単位格子 - C: 200 Å / 単位格子 - γ: 90.0 ° / 単位格子 - α: 90.0 ° / 単位格子 - β: 90.0 ° / 面群: P 4 21 2 |
| CTF補正 | 詳細: Phase flipping with each micrograph |
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: A |
|---|---|
| ソフトウェア | 名称:  UCSF Chimera UCSF Chimera |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)