+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-5711 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Novel Structural Labeling Method using Cryo-electron Tomography and Biotin-Streptavidin System | |||||||||
 マップデータ マップデータ | Averaged subtomogram of Chlamydomonas axoneme, labeled with Streptavidin | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Cryo-electron tomography / Chlamydomonas reinhardtii / biotin-streptavidin / cilia and flagella / dynein / structural labeling | |||||||||
| 生物種 |  | |||||||||
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 57.0 Å | |||||||||
 データ登録者 データ登録者 | Oda T / Kikkawa M | |||||||||
 引用 引用 |  ジャーナル: J Struct Biol / 年: 2013 ジャーナル: J Struct Biol / 年: 2013タイトル: Novel structural labeling method using cryo-electron tomography and biotin-streptavidin system. 著者: Toshiyuki Oda / Masahide Kikkawa /  要旨: There are a number of large macromolecular complexes that play important roles in the cell, and identifying the positions of their components is a key step to understanding their structure and ...There are a number of large macromolecular complexes that play important roles in the cell, and identifying the positions of their components is a key step to understanding their structure and function. Several structural labeling methods have been applied to electron microscopy in order to locate a specific component within a macromolecular complex, but each method is associated with problems in specificity, occupancy, signal intensity or precision. Here, we report a novel method for identifying the 3D locations of proteins using biotin-streptavidin labeling and cryo-electron tomography. We labeled a biotinylation-tagged intermediate chain of an axonemal dynein by streptavidin within the Chlamydomonas axoneme and visualized the 3D positions of the labels using subtomogram averaging. Increase of the density attributed to the bound streptavidin was validated by Student's t-test. In conclusion, the combination of the biotin-streptavidin system and cryo-electron tomography is a powerful method to investigate the structure of large macromolecular complexes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_5711.map.gz emd_5711.map.gz | 42.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-5711-v30.xml emd-5711-v30.xml emd-5711.xml emd-5711.xml | 8.4 KB 8.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_5711_1.tif emd_5711_1.tif | 222.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-5711 http://ftp.pdbj.org/pub/emdb/structures/EMD-5711 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5711 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5711 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_5711_validation.pdf.gz emd_5711_validation.pdf.gz | 77.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_5711_full_validation.pdf.gz emd_5711_full_validation.pdf.gz | 76.9 KB | 表示 | |
| XML形式データ |  emd_5711_validation.xml.gz emd_5711_validation.xml.gz | 493 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5711 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5711 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5711 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5711 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_5711.map.gz / 形式: CCP4 / 大きさ: 51.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_5711.map.gz / 形式: CCP4 / 大きさ: 51.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Averaged subtomogram of Chlamydomonas axoneme, labeled with Streptavidin | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 6.07 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
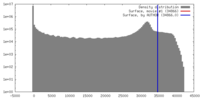
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Chlamydomonas axoneme, strain oda6-ic2-n-bccp, labeled with strep...
| 全体 | 名称: Chlamydomonas axoneme, strain oda6-ic2-n-bccp, labeled with streptavidin |
|---|---|
| 要素 |
|
-超分子 #1000: Chlamydomonas axoneme, strain oda6-ic2-n-bccp, labeled with strep...
| 超分子 | 名称: Chlamydomonas axoneme, strain oda6-ic2-n-bccp, labeled with streptavidin タイプ: sample / ID: 1000 / Number unique components: 2 |
|---|
-超分子 #1: axoneme
| 超分子 | 名称: axoneme / タイプ: organelle_or_cellular_component / ID: 1 / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種:  株: oda6-ic2-n-bccp / Organelle: flagella |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 濃度 | 0.04 mg/mL |
|---|---|
| 緩衝液 | pH: 7.2 詳細: 30 mM Hepes-NaOH pH 7.2, 5 mM MgCl2, 1 mM dithiothreitol, 1 mM EGTA, 50 mM CH3COOK, and 1 mM phenylmethylsulfonyl fluoride |
| グリッド | 詳細: 300 mesh copper grid with home-made holey carbon support |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 80 % / チャンバー内温度: 93 K / 装置: LEICA EM GP / 手法: Blot for 5 seconds before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 3100FFC |
|---|---|
| 温度 | 最低: 90 K / 最高: 95 K |
| 日付 | 2013年2月20日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: TVIPS TEMCAM-F416 (4k x 4k) 平均電子線量: 90 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 25700 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 倍率(公称値): 15000 |
| 試料ステージ | 試料ホルダーモデル: GATAN LIQUID NITROGEN / Tilt series - Axis1 - Min angle: -60.5 ° / Tilt series - Axis1 - Max angle: 60.5 ° |
- 画像解析
画像解析
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 57.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: PEET, Ruby-Helix / 使用したサブトモグラム数: 77 |
|---|
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)