+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-5369 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structural transitions in RCNMV revealing potential mechanism of RNA release (native map) | |||||||||
 マップデータ マップデータ | this is EM map of native RCNMV | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | cryo-electron microscopy / virus / red clover necrotic mosaic virus | |||||||||
| 生物種 |  Red clover necrotic mosaic virus (ウイルス) Red clover necrotic mosaic virus (ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 8.5 Å | |||||||||
 データ登録者 データ登録者 | Sherman MB / Guenther RH / Tama F / Sit TL / Brooks CL / Mikhailov AM / Orlova EV / Baker TS / Lommel SA | |||||||||
 引用 引用 |  ジャーナル: J Virol / 年: 2006 ジャーナル: J Virol / 年: 2006タイトル: Removal of divalent cations induces structural transitions in red clover necrotic mosaic virus, revealing a potential mechanism for RNA release. 著者: Michael B Sherman / Richard H Guenther / Florence Tama / Tim L Sit / Charles L Brooks / Albert M Mikhailov / Elena V Orlova / Timothy S Baker / Steven A Lommel /  要旨: The structure of Red clover necrotic mosaic virus (RCNMV), an icosahedral plant virus, was resolved to 8.5 A by cryoelectron microscopy. The virion capsid has prominent surface protrusions and ...The structure of Red clover necrotic mosaic virus (RCNMV), an icosahedral plant virus, was resolved to 8.5 A by cryoelectron microscopy. The virion capsid has prominent surface protrusions and subunits with a clearly defined shell and protruding domains. The structures of both the individual capsid protein (CP) subunits and the entire virion capsid are consistent with other species in the Tombusviridae family. Within the RCNMV capsid, there is a clearly defined inner cage formed by complexes of genomic RNA and the amino termini of CP subunits. An RCNMV virion has approximately 390 +/- 30 Ca2+ ions bound to the capsid and 420 +/- 25 Mg2+ ions thought to be in the interior of the capsid. Depletion of both Ca2+ and Mg2+ ions from RCNMV leads to significant structural changes, including (i) formation of 11- to 13-A-diameter channels that extend through the capsid and (ii) significant reorganization within the interior of the capsid. Genomic RNA within native capsids containing both Ca2+ and Mg2+ ions is extremely resistant to nucleases, but depletion of both of these cations results in nuclease sensitivity, as measured by a significant reduction in RCNMV infectivity. These results indicate that divalent cations play a central role in capsid dynamics and suggest a mechanism for the release of viral RNA in low-divalent-cation environments such as those found within the cytoplasm of a cell. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_5369.map.gz emd_5369.map.gz | 30.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-5369-v30.xml emd-5369-v30.xml emd-5369.xml emd-5369.xml | 10.9 KB 10.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_5369_1.jpg emd_5369_1.jpg | 39.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-5369 http://ftp.pdbj.org/pub/emdb/structures/EMD-5369 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5369 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5369 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_5369_validation.pdf.gz emd_5369_validation.pdf.gz | 78.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_5369_full_validation.pdf.gz emd_5369_full_validation.pdf.gz | 77.4 KB | 表示 | |
| XML形式データ |  emd_5369_validation.xml.gz emd_5369_validation.xml.gz | 493 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5369 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5369 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5369 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5369 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_5369.map.gz / 形式: CCP4 / 大きさ: 62.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_5369.map.gz / 形式: CCP4 / 大きさ: 62.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | this is EM map of native RCNMV | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.5 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
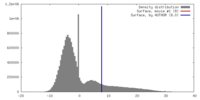
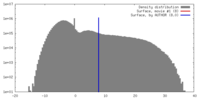
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : RCNMV
| 全体 | 名称: RCNMV |
|---|---|
| 要素 |
|
-超分子 #1000: RCNMV
| 超分子 | 名称: RCNMV / タイプ: sample / ID: 1000 / 詳細: monodisperse sample / Number unique components: 1 |
|---|---|
| 分子量 | 理論値: 7 MDa |
-超分子 #1: Red clover necrotic mosaic virus
| 超分子 | 名称: Red clover necrotic mosaic virus / タイプ: virus / ID: 1 / Name.synonym: RCNMV / NCBI-ID: 12267 / 生物種: Red clover necrotic mosaic virus / データベース: NCBI / ウイルスタイプ: VIRION / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: No / ウイルス・中空状態: No / Syn species name: RCNMV |
|---|---|
| 宿主 | 生物種:  Dianthus (植物) / 別称: PLANTAE(HIGHER PLANTS) Dianthus (植物) / 別称: PLANTAE(HIGHER PLANTS) |
| 分子量 | 実験値: 7 MDa / 理論値: 7 MDa |
| ウイルス殻 | Shell ID: 1 / 直径: 366 Å / T番号(三角分割数): 3 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 6.5 / 詳細: 10 mM Tris-HCl |
| グリッド | 詳細: 200 mesh Quantifoil |
| 凍結 | 凍結剤: ETHANE / チャンバー内温度: 90 K / 装置: HOMEMADE PLUNGER / 詳細: Vitrification instrument: cryo-plunger |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI/PHILIPS CM300FEG/T |
|---|---|
| 温度 | 最低: 95 K / 最高: 123 K / 平均: 95 K |
| アライメント法 | Legacy - 非点収差: manual / Legacy - Electron beam tilt params: 0 |
| 日付 | 2003年10月15日 |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: ZEISS SCAI / デジタル化 - サンプリング間隔: 7 µm / 実像数: 2500 / 平均電子線量: 20 e/Å2 / Od range: 1.5 / ビット/ピクセル: 16 |
| Tilt angle min | 0 |
| Tilt angle max | 0 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 49523 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 2.8 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 47000 |
| 試料ステージ | 試料ホルダー: GATAN 626 / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
- 画像解析
画像解析
| CTF補正 | 詳細: micrograph |
|---|---|
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 8.5 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: PFT / 使用した粒子像数: 4000 |
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| ソフトウェア | 名称: SITUS |
| 詳細 | Protocol: NNM |
| 精密化 | 空間: REAL |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)