+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-5367 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
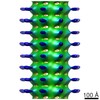
| タイトル | Structure of endophilin bound to a membrane tubule with a diameter of 28nm | |||||||||
 マップデータ マップデータ | Structure of Endophilin bound to bilayer tubule with a diameter of 28nm | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  | |||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 15.0 Å | |||||||||
 データ登録者 データ登録者 | Mim C / Cui H / Gawronski-Salerno JA / Frost A / Lyman E / Voth GA / Unger VM | |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2012 ジャーナル: Cell / 年: 2012タイトル: Structural basis of membrane bending by the N-BAR protein endophilin. 著者: Carsten Mim / Haosheng Cui / Joseph A Gawronski-Salerno / Adam Frost / Edward Lyman / Gregory A Voth / Vinzenz M Unger /  要旨: Functioning as key players in cellular regulation of membrane curvature, BAR domain proteins bend bilayers and recruit interaction partners through poorly understood mechanisms. Using electron ...Functioning as key players in cellular regulation of membrane curvature, BAR domain proteins bend bilayers and recruit interaction partners through poorly understood mechanisms. Using electron cryomicroscopy, we present reconstructions of full-length endophilin and its N-terminal N-BAR domain in their membrane-bound state. Endophilin lattices expose large areas of membrane surface and are held together by promiscuous interactions between endophilin's amphipathic N-terminal helices. Coarse-grained molecular dynamics simulations reveal that endophilin lattices are highly dynamic and that the N-terminal helices are required for formation of a stable and regular scaffold. Furthermore, endophilin accommodates different curvatures through a quantized addition or removal of endophilin dimers, which in some cases causes dimerization of endophilin's SH3 domains, suggesting that the spatial presentation of SH3 domains, rather than affinity, governs the recruitment of downstream interaction partners. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_5367.map.gz emd_5367.map.gz | 7.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-5367-v30.xml emd-5367-v30.xml emd-5367.xml emd-5367.xml | 9.2 KB 9.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_5367_1.jpg emd_5367_1.jpg | 48.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-5367 http://ftp.pdbj.org/pub/emdb/structures/EMD-5367 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5367 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5367 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_5367_validation.pdf.gz emd_5367_validation.pdf.gz | 78.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_5367_full_validation.pdf.gz emd_5367_full_validation.pdf.gz | 78 KB | 表示 | |
| XML形式データ |  emd_5367_validation.xml.gz emd_5367_validation.xml.gz | 494 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5367 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5367 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5367 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5367 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_5367.map.gz / 形式: CCP4 / 大きさ: 62.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_5367.map.gz / 形式: CCP4 / 大きさ: 62.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Structure of Endophilin bound to bilayer tubule with a diameter of 28nm | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.73 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Structure of Endophilin bound to the bilayer
| 全体 | 名称: Structure of Endophilin bound to the bilayer |
|---|---|
| 要素 |
|
-超分子 #1000: Structure of Endophilin bound to the bilayer
| 超分子 | 名称: Structure of Endophilin bound to the bilayer / タイプ: sample / ID: 1000 / 集合状態: Dimer of endophilin bound to the bilayer / Number unique components: 1 |
|---|---|
| 分子量 | 実験値: 37 MDa / 手法: SDS-PAGE |
-分子 #1: endophilin-A1
| 分子 | 名称: endophilin-A1 / タイプ: protein_or_peptide / ID: 1 / Name.synonym: endophilin / コピー数: 2 / 集合状態: Dimer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 37 MDa |
| 組換発現 | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | helical array |
- 試料調製
試料調製
| 濃度 | 0.25 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 / 詳細: 50mM potassium aspartate, 10mM Tris/HCl, 1mM EGTA |
| グリッド | 詳細: C-Flat, 2-2, 400nm mesh |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 105 K / 装置: OTHER / 詳細: Vitrification instrument: FEI Vitrobot |
| 詳細 | protein and liposomes added in solution before freezing |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 日付 | 2009年6月17日 |
| 撮影 | 平均電子線量: 10 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.4 µm / 倍率(公称値): 30000 |
| 試料ステージ | 試料ホルダー: eucentric, single tilt / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - らせんパラメータ - Δz: 19.82 Å 想定した対称性 - らせんパラメータ - ΔΦ: 66.83 ° 想定した対称性 - らせんパラメータ - 軸対称性: C2 (2回回転対称) アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 15.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: SPIDER,XMIPP,IHRSR |
|---|---|
| CTF補正 | 詳細: ACE,MATLAB |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)