+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Lymphostatin at pH 8 in Phosphate buffer - Consensus Map | |||||||||||||||||||||
 マップデータ マップデータ | ||||||||||||||||||||||
 試料 試料 |
| |||||||||||||||||||||
 キーワード キーワード | Lymphostatin virulence factor A/E bacteria Toxin-like / TOXIN | |||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||||||||||||||
| 生物種 |  | |||||||||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.2 Å | |||||||||||||||||||||
 データ登録者 データ登録者 | Griessmann M / Schneider R / Rasmussen T / Bottcher B | |||||||||||||||||||||
| 資金援助 |  ドイツ, ドイツ,  英国, 6件 英国, 6件
| |||||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2025 ジャーナル: Nat Commun / 年: 2025タイトル: Structure of lymphostatin, a large multi-functional virulence factor of pathogenic Escherichia coli. 著者: Matthias Griessmann / Tim Rasmussen / Vanessa J Flegler / Christian Kraft / Ronja Schneider / Max Hateley / Lukas Spantzel / Mark P Stevens / Michael Börsch / Bettina Böttcher /   要旨: Lymphostatin is a key virulence factor of enteropathogenic and enterohaemorrhagic Escherichia coli, playing roles in bacterial colonisation of the gut and in the inhibition of lymphocyte ...Lymphostatin is a key virulence factor of enteropathogenic and enterohaemorrhagic Escherichia coli, playing roles in bacterial colonisation of the gut and in the inhibition of lymphocyte proliferation and proinflammatory responses. The protein's glycosyltransferase and cysteine protease motifs are required for activity against lymphocytes, but high-resolution structural information has proven elusive. Here, we describe the structure of lymphostatin from enteropathogenic E. coli O127:H6, determined by electron cryo-microscopy at different pH values. We observe three conformations of a highly complex molecule with two glycosyltransferase domains, one PaToxP-like protease domain, an ADP-ribosyltransferase domain, a vertex domain and a delivery domain. Long linkers hold these domains together and occlude the catalytic sites of the N-terminal glycosyltransferase and protease domains. Lymphostatin binds to bovine T-lymphocytes and HEK-293T cells, forming clusters at the plasma membrane that are internalized. With six distinct domains, lymphostatin can be regarded as a multitool of pathogenic Escherichia coli, enabling complex interactions with host cells. | |||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_53168.map.gz emd_53168.map.gz | 226.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-53168-v30.xml emd-53168-v30.xml emd-53168.xml emd-53168.xml | 19.9 KB 19.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_53168_fsc.xml emd_53168_fsc.xml | 14.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_53168.png emd_53168.png | 125.8 KB | ||
| マスクデータ |  emd_53168_msk_1.map emd_53168_msk_1.map | 244.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-53168.cif.gz emd-53168.cif.gz | 7.1 KB | ||
| その他 |  emd_53168_half_map_1.map.gz emd_53168_half_map_1.map.gz emd_53168_half_map_2.map.gz emd_53168_half_map_2.map.gz | 195.4 MB 195.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-53168 http://ftp.pdbj.org/pub/emdb/structures/EMD-53168 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-53168 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-53168 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_53168_validation.pdf.gz emd_53168_validation.pdf.gz | 859 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_53168_full_validation.pdf.gz emd_53168_full_validation.pdf.gz | 858.6 KB | 表示 | |
| XML形式データ |  emd_53168_validation.xml.gz emd_53168_validation.xml.gz | 21.5 KB | 表示 | |
| CIF形式データ |  emd_53168_validation.cif.gz emd_53168_validation.cif.gz | 28.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-53168 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-53168 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-53168 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-53168 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_53168.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_53168.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.946 Å | ||||||||||||||||||||||||||||||||||||
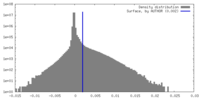
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_53168_msk_1.map emd_53168_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
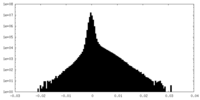
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_53168_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_53168_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Lymphostatin
| 全体 | 名称: Lymphostatin |
|---|---|
| 要素 |
|
-超分子 #1: Lymphostatin
| 超分子 | 名称: Lymphostatin / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 366 KDa |
-分子 #1: Lymphostatin
| 分子 | 名称: Lymphostatin / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MRLPEKVLFP PVTSGLSGQE KQKKPKSITG FQENYQRNIR PIKTASEARL RFFDKMVSKE NSLEDVVSLG EMIQKEIYGH EQRTFSPVHH TGNWKSSLLH NALLGLANVY NGLRETEYPN TFNRDGIKST NSFRDNLLTK TRTPRDNFEE GIKHPEHATI PYDNDNESNK ...文字列: MRLPEKVLFP PVTSGLSGQE KQKKPKSITG FQENYQRNIR PIKTASEARL RFFDKMVSKE NSLEDVVSLG EMIQKEIYGH EQRTFSPVHH TGNWKSSLLH NALLGLANVY NGLRETEYPN TFNRDGIKST NSFRDNLLTK TRTPRDNFEE GIKHPEHATI PYDNDNESNK LLKAGKIAGN NNELLMEIKK ESQSDHQIPL SDKFLKRKKR SPVAEDKVQN SLTPENFVQK ISLSDELKTK YANEIIEIKR IMGEYNLLPD KNSRNGLKLL QKQADLLKII MEDTSVTENT FKNIEIAITD IKREYYSHTV DIEKNIHAIW VAGSPPESIS DYIKTFLKTY KEFTYYLWVD EKAFGAAKFT SVLKQIAFDL ACRTIQQNTP QKNIDFINLY NEIRKKYNNN PSGQQEYLNK LRELYATYQK ISTPLKHMFN SFFLENMIKL QDNFFNYCIV KGVTEINDEL RINYLKNVIK LSDDDIGNYQ KTINDNKDRV KKLILDLQKQ FGENRISIKD VNSLTSLSKS ENNHNYQTEM LLRWNYPAAS DLLRMYILKE HGGIYTDTDM MPAYSKQVIF KIMMQTNGDN RFLEDLKLRR AISDGVLRYV NNQNIDEVNY NEISDADKNI IKKILTEISK MPEDSIFTKI NTRIPRDTMP ILRRYHLWPD GWNIRGLNGF MLSHKGSEVI DAVIAGQNQA YRELRRIRDN IHSEIYFKQT DELSSLPDTD KIGGILVKKY LSGSLFSKFR QDTIIPEALS TLQISGPDLI QRKMLQFFRS RGVLGEEFIN ERKLSDKAYI GVYKTTGTGK YDWLTPESIG VNDVTPADES TWCIGKGRCV DDFLFKDVST LKTENLPELF LTKIDTDTFF SQWSTKTKKD LQKKIQDLTV RYNELIDSST IDFKNLYEID QMLHMIMLEM NDDIAKRSLF SLQVQISEKI RRMTIPVDNI INIYPDLHKK NDNDLSMSIK GFLASNPHTK INILYSNKTE HNIFIKDLFS FAVMENELRD IINNMSKDKT PENWEGRVML QRYLELKMKD HLSLQSSQEA NEFLEISTFI YENDFLREKI EAVKNKMNSH ELYFEKIKKE QNTWQDLSTK EQKLQLIKAL KEISGNTEKD SHYDRLLDAF FKKHNENIHN KIQRIKDEFK EYSRVAIHNI DKVIFKGQTL DRLYHEGYVF SDINTLSRYT LHGLGITGVH TEENLLPAPS SSLINILKEH YNEDEISAKL PLAYDYILNK KESSSIPVEI LNKLSELPPH ELLTPVLGQS VNPLGMGYSS DNGKITEQVI VSGADGFDNP ISGLIYTYLE DLYNIHVRMR EGTLNSQNLR QLLENSVSSC FLTEQSINKL LSEAEKRPYQ SLTEIHQHLT GLPTIADATL SLLSVGLPGT GKLLRREQDY GRPPVTAIQD STFVLPYNFK GIGFNDNIIS SAPVASSLHF IAEHAKYTLL SWPEFYRHHA QRWFEMAKGY GSQNIDFHPQ SLLVTQEGRC MGLALLYLQT EDTAHYSILQ ENLMTVSALH QTSNRDKLPL SKDDNSLMTR TYSLIEMLQY QGNKYITNES LLHKTAWNQE RITLLFNEKG VKRALISTPN HTLVLQQLED IYRLTDPNFG HADFLSPIDA LKFIEAMIQL TPTLQEYYGL LNKDINKHIQ VHYAESDMVW NKLLPENDAG LSTRIQHTTT DRLANLAEPV AVAGISLPVK TLYDIGATLD GRRITSPPTS EQIPSLRLNG DVLNDYLSRT VLTPEQADNI RKILHTQGIR SGTRPIDPEM IRGTQDDLVS SQTRLQRQAT RVKQQLAGVL DTLQQHFQNI PRSSGRHLSV ENIELADIGS GRFNLQIRDG ETLHTTSVEV PEVVSRFQKL STMLSALPAS GIMDFDLGMS VVGVVQYARL LQQGHEDSTL AKINLAMDIK QLSEATLGSM IQIAGNKFLN TEGIQGFRLE SAVAEGMRSV ATRTGGTMGK ALSASARVLE LPVLETVLGT WNLYNSVIQL QQATSYSETM AARVQIAFDS ISLGLTAASV AFPPLIIATG PIAAIGMGAS SIARNVARKE ERHTQWLEYK KFLTDGSKHI VVASPERGLL DFSGNKVFGK MVLDLRQSPP LLHGESSFNA DRKIGHRPDL GDWQIREKVG YANSISPYSS LAHGYANSKW PRTIPKIPSG EYDTIILGYG HQYQANTEIE YLSNWIVWRE AVPDSTSRHK RPPLEVLNSQ CTVIAGERKT TVLPLRVLSD LTPECTEQAI SLKDYKFILR GGSGGLAVQV GGAGYYDIDA NLVAKENTLS FRGLPEEFPL TFDLSKQTQS VMLKTPDDEV PVMTITQKGI NTLVGTAAGK DRLIGNDKDN TFHTSSGGGT VISGGGNNRY IIPRDLKTPL TLTLSSNSVS HEIFLPETTL AELKPVAFEL SLIYWAGNNI NVQPEDEAKL NHFAGNFRVH TRDGMTLEAV SRENGIQLAI SLCDVQRWQA VYPEENNRPD AILDRLHDMG WSLTPEVRFQ GGETQVSYDP LTRQLVYQLQ ARYSEFQLAG SRHHTTAVTG TPGSRYIIMK PVTTQILPTQ IILAGDNDHP ETIDLLEASP VLVEGKKDKN SVILTIATIQ YSLQLTISGI EESLPETTRV AIQPQDTRLL GDVLRILPDN GNWVGIFRSG HTPTVNRLEN LMALNQVMTF LPRVSGSAEQ VLCLENLGGV RKKVEGELLS GKLKGAWKAE GEPTVPVNIS DLSIPPYSRL YLIFEGKNNV LLRSKVHAAP LKITSAGEMQ LSERQWQQQE HIIVKPDNEA PSLILSEFRR FTISSDKTFS LKLMCHQGMV RIDRRSLSVR LFYLREQPGI GSLRLTFRDF FTEVMDTTDR EILEKELRPI LIGDTHRFIN AAYKNHLNIQ LGDGVLNLAD IVAEYARIQK EETSKILYQY QGAMKKKTDG PSVVEDAIMT TTVTTDSGEL FPTFHPWYTD DLSGRYKSVP MARKADTLYH LTPKGDLQII YQVATKMVNQ AMIVSLPNYR HEWEKYNLSI LSEIPQNNNT VVHSILRVNG PTMQVRTIDY RGTDENNPIV SFSDTTFING EQMLSYDSHS SGRVYSREEY MMWELQQRVS EASSARTQDY WLMDAAVRNG EWKITPELLR HTPGYIRSTV SKWSRGWLKT GTILQTPEDR NTDVYLTTIQ NNVFSRQGGG YQVYYRIDGM AGADIADNAP GETRCTLRPG TCFEVTSVDE RHYEWNIIYV TLKTCGWSRN GQSKTPNGDN LFN UniProtKB: Lymphostatin |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 詳細: 25 mM sodium phosphate pH 8, 100 mM NaCl, 0.075 mM MnCl2, 1.5 mM TCEP |
|---|---|
| グリッド | モデル: Quantifoil |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 70.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.2 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: RIGID BODY FIT |
|---|
 ムービー
ムービー コントローラー
コントローラー




































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)