+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-electron microscopy structure of glucose/xylose isomerase from Streptomyces rubiginosus with magnesium ions in the active site | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | magnesium / isomerase / sugars / glucose / xylose / metal ion / Cryo-EM / METAL BINDING PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報xylose isomerase / xylose isomerase activity / D-xylose metabolic process / magnesium ion binding / identical protein binding / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Streptomyces rubiginosus (バクテリア) Streptomyces rubiginosus (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.0 Å | |||||||||
 データ登録者 データ登録者 | Slawek J / Klonecka A / Rawski M / Kozak M | |||||||||
| 資金援助 |  ポーランド, 1件 ポーランド, 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Cryo-electron microscopy structure of glucose/xylose isomerase from Streptomyces rubiginosus with magnesium ions in the active site 著者: Slawek J / Klonecka A / Rawski M / Kozak M | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_51522.map.gz emd_51522.map.gz | 32.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-51522-v30.xml emd-51522-v30.xml emd-51522.xml emd-51522.xml | 16.1 KB 16.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_51522_fsc.xml emd_51522_fsc.xml | 11.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_51522.png emd_51522.png | 117.1 KB | ||
| Filedesc metadata |  emd-51522.cif.gz emd-51522.cif.gz | 5.6 KB | ||
| その他 |  emd_51522_half_map_1.map.gz emd_51522_half_map_1.map.gz emd_51522_half_map_2.map.gz emd_51522_half_map_2.map.gz | 59.3 MB 59.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-51522 http://ftp.pdbj.org/pub/emdb/structures/EMD-51522 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-51522 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-51522 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  9greMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_51522.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_51522.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.84 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_51522_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
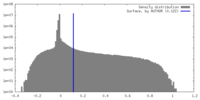
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_51522_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : glucose/xylose isomerase with cobalt ions
| 全体 | 名称: glucose/xylose isomerase with cobalt ions |
|---|---|
| 要素 |
|
-超分子 #1: glucose/xylose isomerase with cobalt ions
| 超分子 | 名称: glucose/xylose isomerase with cobalt ions / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Streptomyces rubiginosus (バクテリア) Streptomyces rubiginosus (バクテリア) |
| 分子量 | 理論値: 43 KDa |
-分子 #1: Xylose isomerase
| 分子 | 名称: Xylose isomerase / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO / EC番号: xylose isomerase |
|---|---|
| 由来(天然) | 生物種:  Streptomyces rubiginosus (バクテリア) Streptomyces rubiginosus (バクテリア) |
| 分子量 | 理論値: 43.283297 KDa |
| 配列 | 文字列: MNYQPTPEDR FTFGLWTVGW QGRDPFGDAT RRALDPVESV RRLAELGAHG VTFHDDDLIP FGSSDSEREE HVKRFRQALD DTGMKVPMA TTNLFTHPVF KDGGFTANDR DVRRYALRKT IRNIDLAVEL GAETYVAWGG REGAESGGAK DVRDALDRMK E AFDLLGEY ...文字列: MNYQPTPEDR FTFGLWTVGW QGRDPFGDAT RRALDPVESV RRLAELGAHG VTFHDDDLIP FGSSDSEREE HVKRFRQALD DTGMKVPMA TTNLFTHPVF KDGGFTANDR DVRRYALRKT IRNIDLAVEL GAETYVAWGG REGAESGGAK DVRDALDRMK E AFDLLGEY VTSQGYDIRF AIEPKPNEPR GDILLPTVGH ALAFIERLER PELYGVNPEV GHEQMAGLNF PHGIAQALWA GK LFHIDLN GQNGIKYDQD LRFGAGDLRA AFWLVDLLES AGYSGPRHFD FKPPRTEDFD GVWASAAGCM RNYLILKERA AAF RADPEV QEALRASRLD ELARPTAADG LQALLDDRSA FEEFDVDAAA ARGMAFERLD QLAMDHLLGA RG UniProtKB: Xylose isomerase |
-分子 #2: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 2 / コピー数: 8 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #3: water
| 分子 | 名称: water / タイプ: ligand / ID: 3 / コピー数: 4 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.9 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 構成要素 - 濃度: 50.0 mM / 構成要素 - 式: PBS / 構成要素 - 名称: Phosphate-buffered saline / 詳細: 50 mM PBS pH 7.4 |
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 200 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 5 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 0.9 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)