+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-5110 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Feline panleukopenia virus in complex with FAb from neutralizing antibody MAb 16 | |||||||||
 マップデータ マップデータ | This is a map of the FAb from neutralizing antibody MAb 16 interacting with FPV | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | parvovirus / antigenic epitope / antibody / Fab / neutralizing | |||||||||
| 生物種 |  Feline panleukopenia virus (ネコ汎白血球減少症ウイルス) Feline panleukopenia virus (ネコ汎白血球減少症ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 18.0 Å | |||||||||
 データ登録者 データ登録者 | Hafenstein S / Bowman VD / Sun T / Nelson CDS / Palermo LM / Chipman PR / Battisti AJ / Parrish CR / Rossmann MG | |||||||||
 引用 引用 |  ジャーナル: J Virol / 年: 2009 ジャーナル: J Virol / 年: 2009タイトル: Structural comparison of different antibodies interacting with parvovirus capsids. 著者: Susan Hafenstein / Valorie D Bowman / Tao Sun / Christian D S Nelson / Laura M Palermo / Paul R Chipman / Anthony J Battisti / Colin R Parrish / Michael G Rossmann /  要旨: The structures of canine parvovirus (CPV) and feline parvovirus (FPV) complexed with antibody fragments from eight different neutralizing monoclonal antibodies were determined by cryo-electron ...The structures of canine parvovirus (CPV) and feline parvovirus (FPV) complexed with antibody fragments from eight different neutralizing monoclonal antibodies were determined by cryo-electron microscopy (cryoEM) reconstruction to resolutions varying from 8.5 to 18 A. The crystal structure of one of the Fab molecules and the sequence of the variable domain for each of the Fab molecules have been determined. The structures of Fab fragments not determined crystallographically were predicted by homology modeling according to the amino acid sequence. Fitting of the Fab and virus structures into the cryoEM densities identified the footprints of each antibody on the viral surface. As anticipated from earlier analyses, the Fab binding sites are directed to two epitopes, A and B. The A site is on an exposed part of the surface near an icosahedral threefold axis, whereas the B site is about equidistant from the surrounding five-, three-, and twofold axes. One antibody directed to the A site binds CPV but not FPV. Two of the antibodies directed to the B site neutralize the virus as Fab fragments. The differences in antibody properties have been linked to the amino acids within the antibody footprints, the position of the binding site relative to the icosahedral symmetry elements, and the orientation of the Fab structure relative to the surface of the virus. Most of the exposed surface area was antigenic, although each of the antibodies had a common area of overlap that coincided with the positions of the previously mapped escape mutations. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_5110.map.gz emd_5110.map.gz | 3.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-5110-v30.xml emd-5110-v30.xml emd-5110.xml emd-5110.xml | 10.3 KB 10.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_5110_1.png emd_5110_1.png | 239.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-5110 http://ftp.pdbj.org/pub/emdb/structures/EMD-5110 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5110 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5110 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_5110_validation.pdf.gz emd_5110_validation.pdf.gz | 314.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_5110_full_validation.pdf.gz emd_5110_full_validation.pdf.gz | 314.1 KB | 表示 | |
| XML形式データ |  emd_5110_validation.xml.gz emd_5110_validation.xml.gz | 5.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5110 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5110 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5110 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5110 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  3iy5MC  5105C  5106C  5107C  5108C  5109C  5111C  5112C  3gk8C  3iy0C  3iy1C  3iy2C  3iy3C  3iy4C  3iy6C  3iy7C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_5110.map.gz / 形式: CCP4 / 大きさ: 23.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_5110.map.gz / 形式: CCP4 / 大きさ: 23.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | This is a map of the FAb from neutralizing antibody MAb 16 interacting with FPV | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.88 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
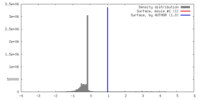
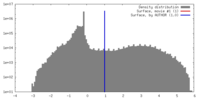
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Fab fragment from MAb 15 interacting with feline panleukopenia vi...
| 全体 | 名称: Fab fragment from MAb 15 interacting with feline panleukopenia virus (FPV) |
|---|---|
| 要素 |
|
-超分子 #1000: Fab fragment from MAb 15 interacting with feline panleukopenia vi...
| 超分子 | 名称: Fab fragment from MAb 15 interacting with feline panleukopenia virus (FPV) タイプ: sample / ID: 1000 / Number unique components: 2 |
|---|
-超分子 #1: Feline panleukopenia virus
| 超分子 | 名称: Feline panleukopenia virus / タイプ: virus / ID: 1 / Name.synonym: FPV / NCBI-ID: 10786 / 生物種: Feline panleukopenia virus / データベース: NCBI / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No / Syn species name: FPV |
|---|---|
| 宿主 | 生物種:  |
| ウイルス殻 | Shell ID: 1 / 直径: 280 Å / T番号(三角分割数): 1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 / 詳細: 10mM Tris-HCL |
| グリッド | 詳細: quantifoils |
| 凍結 | 凍結剤: ETHANE / チャンバー内温度: 120 K / 装置: HOMEMADE PLUNGER / 詳細: Vitrification instrument: plunger / 手法: blot before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI/PHILIPS CM300FEG/T |
|---|---|
| 温度 | 最低: 83 K / 最高: 83 K / 平均: 93 K |
| アライメント法 | Legacy - 非点収差: astigmatism was corrected at 100,000 times magnification |
| 日付 | 2004年6月3日 |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: ZEISS SCAI / デジタル化 - サンプリング間隔: 7 µm / 実像数: 48 / 平均電子線量: 37.70 e/Å2 / Od range: 0.9 / ビット/ピクセル: 8 |
| 電子線 | 加速電圧: 300 kV / 電子線源: TUNGSTEN HAIRPIN |
| 電子光学系 | 倍率(補正後): 47190 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 4.8 µm / 最小 デフォーカス(公称値): 0.4 µm / 倍率(公称値): 45000 |
| 試料ステージ | 試料ホルダー: side mounted nitrogen cooled / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
- 画像解析
画像解析
| CTF補正 | 詳細: robem |
|---|---|
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 18.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: EMPFT EM3DR / 使用した粒子像数: 2084 |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera



 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)