[English] 日本語
 Yorodumi
Yorodumi- EMDB-50979: Cryo-EM structure of IrtAB in outward-occluded state in LMNG in c... -
+ Open data
Open data
- Basic information
Basic information
| Entry |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Title | Cryo-EM structure of IrtAB in outward-occluded state in LMNG in complex with ADP-vanadate | |||||||||||||||
 Map data Map data | ||||||||||||||||
 Sample Sample |
| |||||||||||||||
 Keywords Keywords | ABC transporter / type IV ABC importer siderophore / mycobactin / heterodimeric ABC transporter / MEMBRANE PROTEIN | |||||||||||||||
| Function / homology |  Function and homology information Function and homology informationTranslocases; Catalysing the translocation of inorganic cations; Linked to the hydrolysis of a nucleoside triphosphate / ATPase-coupled lipid transmembrane transporter activity / ABC-type transporter activity / oxidoreductase activity / ATP hydrolysis activity / ATP binding / plasma membrane Similarity search - Function | |||||||||||||||
| Biological species |  Mycolicibacterium thermoresistibile ATCC 19527 (bacteria) Mycolicibacterium thermoresistibile ATCC 19527 (bacteria) | |||||||||||||||
| Method | single particle reconstruction / cryo EM / Resolution: 3.23 Å | |||||||||||||||
 Authors Authors | Gonda I / Seeger MA | |||||||||||||||
| Funding support |  Switzerland, European Union, 4 items Switzerland, European Union, 4 items
| |||||||||||||||
 Citation Citation |  Journal: Nat Commun / Year: 2025 Journal: Nat Commun / Year: 2025Title: The mycobacterial ABC transporter IrtAB employs a membrane-facing crevice for siderophore-mediated iron uptake. Authors: Imre Gonda / Simona Sorrentino / Laura Galazzo / Nicolas P Lichti / Fabian M Arnold / Ahmad R Mehdipour / Enrica Bordignon / Markus A Seeger /   Abstract: The mycobacterial ABC transporter IrtAB features an ABC exporter fold, yet it imports iron-charged siderophores called mycobactins. Here, we present extensive cryo-EM analyses and DEER measurements, ...The mycobacterial ABC transporter IrtAB features an ABC exporter fold, yet it imports iron-charged siderophores called mycobactins. Here, we present extensive cryo-EM analyses and DEER measurements, revealing that IrtAB alternates between an inward-facing and an outward-occluded conformation, but does not sample an outward-facing conformation. When IrtAB is locked in its outward-occluded conformation in nanodiscs, mycobactin is bound in the middle of the lipid bilayer at a membrane-facing crevice opening at the heterodimeric interface. Mutations introduced at the crevice abrogate mycobactin import and in corresponding structures, the crevice is collapsed. A conserved triple histidine motif coordinating a zinc ion is present below the mycobactin binding site. Substitution of these histidine residues with alanine results in a decoupled transporter, which hydrolyzes ATP, but lost its capacity to import mycobactins. Our data suggest that IrtAB imports mycobactin via a credit-card mechanism in a transport cycle that is coupled to the presence of zinc. | |||||||||||||||
| History |
|
- Structure visualization
Structure visualization
| Supplemental images |
|---|
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_50979.map.gz emd_50979.map.gz | 28.8 MB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-50979-v30.xml emd-50979-v30.xml emd-50979.xml emd-50979.xml | 24 KB 24 KB | Display Display |  EMDB header EMDB header |
| FSC (resolution estimation) |  emd_50979_fsc.xml emd_50979_fsc.xml | 9.2 KB | Display |  FSC data file FSC data file |
| Images |  emd_50979.png emd_50979.png | 77.2 KB | ||
| Masks |  emd_50979_msk_1.map emd_50979_msk_1.map | 30.5 MB |  Mask map Mask map | |
| Filedesc metadata |  emd-50979.cif.gz emd-50979.cif.gz | 8.1 KB | ||
| Others |  emd_50979_half_map_1.map.gz emd_50979_half_map_1.map.gz emd_50979_half_map_2.map.gz emd_50979_half_map_2.map.gz | 28.4 MB 28.4 MB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-50979 http://ftp.pdbj.org/pub/emdb/structures/EMD-50979 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-50979 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-50979 | HTTPS FTP |
-Related structure data
| Related structure data |  9g2mMC  9fw3C  9fxcC  9g2kC  9g2lC  9g2pC  9g2sC  9g2tC  9g2vC  9g2xC  9g2yC  9g2zC  9g36C  9g37C  9gl3C M: atomic model generated by this map C: citing same article ( |
|---|---|
| Similar structure data | Similarity search - Function & homology  F&H Search F&H Search |
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| Related items in Molecule of the Month |
- Map
Map
| File |  Download / File: emd_50979.map.gz / Format: CCP4 / Size: 30.5 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_50979.map.gz / Format: CCP4 / Size: 30.5 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & slices | Image control
Images are generated by Spider. | ||||||||||||||||||||||||||||||||||||
| Voxel size | X=Y=Z: 1.3 Å | ||||||||||||||||||||||||||||||||||||
| Density |
| ||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
|
-Supplemental data
-Mask #1
| File |  emd_50979_msk_1.map emd_50979_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
| Density Histograms |
-Half map: #2
| File | emd_50979_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
| Density Histograms |
-Half map: #1
| File | emd_50979_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
| Density Histograms |
- Sample components
Sample components
-Entire : IrtAB
| Entire | Name: IrtAB |
|---|---|
| Components |
|
-Supramolecule #1: IrtAB
| Supramolecule | Name: IrtAB / type: complex / ID: 1 / Parent: 0 / Macromolecule list: #1-#2 Details: wild-type IrtAB in LMNG in complex with ADP-vanadate in presence of 100uM MBT (unobserved) |
|---|---|
| Source (natural) | Organism:  Mycolicibacterium thermoresistibile ATCC 19527 (bacteria) Mycolicibacterium thermoresistibile ATCC 19527 (bacteria) |
| Molecular weight | Theoretical: 158.9 KDa |
-Macromolecule #1: Mycobactin import ATP-binding/permease protein IrtA
| Macromolecule | Name: Mycobactin import ATP-binding/permease protein IrtA / type: protein_or_peptide / ID: 1 / Number of copies: 1 / Enantiomer: LEVO EC number: Translocases; Catalysing the translocation of inorganic cations; Linked to the hydrolysis of a nucleoside triphosphate |
|---|---|
| Source (natural) | Organism:  Mycolicibacterium thermoresistibile ATCC 19527 (bacteria) Mycolicibacterium thermoresistibile ATCC 19527 (bacteria) |
| Molecular weight | Theoretical: 97.365344 KDa |
| Recombinant expression | Organism:  |
| Sequence | String: SLRGLGARDH QATVVDKEYI APHFVRVRLV SPTLFDEVIV EPTSWLRFWF PDPDGSDTEF QRAYTITESD PETGRFAVDM VLHEPAGPA STWARTVEPG ATIAVMSMGS RGFSVPEDPE DRPVGYLLIG DSASTPAING IIEVVPHDIP IELYLEQHHD D DVLIPLAE ...String: SLRGLGARDH QATVVDKEYI APHFVRVRLV SPTLFDEVIV EPTSWLRFWF PDPDGSDTEF QRAYTITESD PETGRFAVDM VLHEPAGPA STWARTVEPG ATIAVMSMGS RGFSVPEDPE DRPVGYLLIG DSASTPAING IIEVVPHDIP IELYLEQHHD D DVLIPLAE HPRLRVHRVS RDDASSLAAA LELRDWSNWY CWAGPEAGAL KQVRTRLRDE FGFPKREVYA QAYWTEGRAM GS SRGETST PAKPAAKTAP AKAAAKPAAA SGAGTPEHAA APAAATTGAP QAAPAPGAAQ PRTPVRGRWR AEAGSRLLAP LKK PLIVSG VLQALITLIE LAPFVLLVEL ARLLLGGAEA ERLWTLGLTA VSLIGLGAVL AAAMTLWLHR VDARFAHELR GRLL TKLSR LPLGWFTRRG SASTKQLVQD DTLALHYLIT HAIPDAVAAV VAPVAVLVYL FVADWRVALV LFIPVLVYLV LMSVM TIQS GSKIAQAPRW AERMGGEAGA FLEGQPVIRI FGGAAASRFR RRLDDYIDFL VSWQRPFVGK KTLMDLVTRP ATFLWI ILV AGVPLVVTGR MDPVNLLPFL LLGTTFGARL LGIGYGLSGI QTGMLAARRI QTVLDEPELV VRDRTGQAGT DHASGDQ AR PGTVELDRVS FEYRPGVPVI RDVTLTLRPG TVTALVGPSG SGKSTLAALV ARFHDVTQGA IRVDGRDIRT LTADELYR R VGFVLQDAQL VHGSVAENIA LAEPDAGLER IRTAARDAQI HDRITRMPDG YDSVLGAGSA LSGGERQRVT IARAILADT PVLVLDEATA FADPESEYLV QQAINRLTRD RTVLVIAHRL HTITHADQIV VLDDGRIVEV GTHDELLAAG GRYRGLWDSG RYSSPDAGR PVSADAVEVG R UniProtKB: Mycobactin import ATP-binding/permease protein IrtA |
-Macromolecule #2: Mycobactin import ATP-binding/permease protein IrtB
| Macromolecule | Name: Mycobactin import ATP-binding/permease protein IrtB / type: protein_or_peptide / ID: 2 Details: contains cleaved, C-terminal 3C enzyme recognition site Number of copies: 1 / Enantiomer: LEVO EC number: Translocases; Catalysing the translocation of inorganic cations; Linked to the hydrolysis of a nucleoside triphosphate |
|---|---|
| Source (natural) | Organism:  Mycolicibacterium thermoresistibile ATCC 19527 (bacteria) Mycolicibacterium thermoresistibile ATCC 19527 (bacteria) |
| Molecular weight | Theoretical: 61.559539 KDa |
| Recombinant expression | Organism:  |
| Sequence | String: MIRTLLRLVP AEKRGAVAGY AVLTLLSVLL RAVGAVLLIP LLAALFSDTP SDAWLWLGWL TAVTLAGWVT DTNTARLGFD LGFAVLSRT QHDMADRLPN VAMSWFTPDN TATARQAIAA TGPELAGLVV NLLTPLIGAA LLPAAIGVAL LFVSVPLGLA A LAGVAVLF ...String: MIRTLLRLVP AEKRGAVAGY AVLTLLSVLL RAVGAVLLIP LLAALFSDTP SDAWLWLGWL TAVTLAGWVT DTNTARLGFD LGFAVLSRT QHDMADRLPN VAMSWFTPDN TATARQAIAA TGPELAGLVV NLLTPLIGAA LLPAAIGVAL LFVSVPLGLA A LAGVAVLF GALALSGRLS RAADKVAGET NSAFTERIIE FARTQQALRA ARRVEPARSQ VGSALAAQHG AGLRLLTMQI PG QVLFSLA GQVALIGFAG MAVWLTVRGQ LGVPEAIALI VVLVRYLEPF AAIADLAPAL ETTRATLNRI QAVLDAPTLP AGR RRLDRT GAAPSIEFDD VRFSYGDEVV LDGVSFTLRP GNTTAIVGPS GSGKTTILSL IAGLQQPASG RVLLDGVDVT TLDP EARRA AVSVVFQHPY LFDGTLRDNV LVGDPEADPD DVTAAMRLAR VDELLDRLPD GDATVVGEGG TALSGGERQR VSIAR ALLK PAPVLLVDEA TSALDNANEA AVVDALTADP RPRTRVIVAH RLASIRHADR VLFVEAGRVV EDGAIDELLA AGGRFA QFW AQQQAASEWA IGSTARALEV LFQ UniProtKB: Mycobactin import ATP-binding/permease protein IrtB |
-Macromolecule #3: ADP ORTHOVANADATE
| Macromolecule | Name: ADP ORTHOVANADATE / type: ligand / ID: 3 / Number of copies: 2 / Formula: AOV |
|---|---|
| Molecular weight | Theoretical: 544.156 Da |
| Chemical component information | 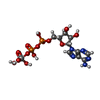 ChemComp-AOV: |
-Macromolecule #4: MAGNESIUM ION
| Macromolecule | Name: MAGNESIUM ION / type: ligand / ID: 4 / Number of copies: 2 / Formula: MG |
|---|---|
| Molecular weight | Theoretical: 24.305 Da |
-Macromolecule #5: ZINC ION
| Macromolecule | Name: ZINC ION / type: ligand / ID: 5 / Number of copies: 1 / Formula: ZN |
|---|---|
| Molecular weight | Theoretical: 65.409 Da |
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | single particle reconstruction |
| Aggregation state | particle |
- Sample preparation
Sample preparation
| Concentration | 3.2 mg/mL | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Buffer | pH: 7.5 Component:
Details: 20mM Tris-HCl pH 7.5, 150mM NaCl, 0.01%LMNG | |||||||||||||||||||||
| Grid | Model: Quantifoil R1.2/1.3 / Material: GOLD / Mesh: 200 / Support film - Material: CARBON / Support film - topology: HOLEY / Pretreatment - Type: GLOW DISCHARGE / Pretreatment - Time: 60 sec. / Pretreatment - Atmosphere: AIR | |||||||||||||||||||||
| Vitrification | Cryogen name: ETHANE-PROPANE / Chamber humidity: 90 % / Chamber temperature: 283 K / Instrument: LEICA EM GP | |||||||||||||||||||||
| Details | Sample was pre-incubated with 2.5mM ATP, 2.5mM Mg2+ ion, 5mM orthovanadate and 100uM mycobactin prior plunge-freezing. |
- Electron microscopy
Electron microscopy
| Microscope | TFS KRIOS |
|---|---|
| Specialist optics | Energy filter - Name: GIF Bioquantum / Energy filter - Slit width: 20 eV |
| Image recording | Film or detector model: GATAN K3 (6k x 4k) / Digitization - Dimensions - Width: 11520 pixel / Digitization - Dimensions - Height: 8184 pixel / Number grids imaged: 1 / Number real images: 7906 / Average exposure time: 0.93 sec. / Average electron dose: 59.9 e/Å2 Details: micrographs were collected in super-resolution mode, 34 frames per movie |
| Electron beam | Acceleration voltage: 300 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | C2 aperture diameter: 50.0 µm / Calibrated defocus max: 2.5 µm / Calibrated defocus min: 1.0 µm / Illumination mode: FLOOD BEAM / Imaging mode: BRIGHT FIELD / Cs: 2.7 mm / Nominal defocus max: 2.5 µm / Nominal defocus min: 1.0 µm / Nominal magnification: 130000 |
| Sample stage | Specimen holder model: FEI TITAN KRIOS AUTOGRID HOLDER / Cooling holder cryogen: NITROGEN |
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
 Movie
Movie Controller
Controller




















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)














































