登録情報 データベース : EMDB / ID : EMD-50514タイトル Cryo-EM structure of Mycobacterium tuberculosis sigma-B RNA polymerase bound to -10 promoter element ssDNA oligo - unswiveled conformation Primary map sharpened 複合体 : RNA polymerase holoenzyme bound to -10 promoter element ssDNA oligoタンパク質・ペプチド : DNA-directed RNA polymerase subunit alphaタンパク質・ペプチド : DNA-directed RNA polymerase subunit betaタンパク質・ペプチド : DNA-directed RNA polymerase subunit beta'タンパク質・ペプチド : DNA-directed RNA polymerase subunit omegaタンパク質・ペプチド : RNA polymerase sigma factor SigBDNA : DNA (17-MER) / / / / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
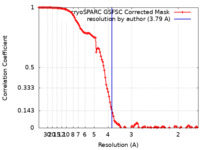
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Mycobacterium tuberculosis H37Rv (結核菌) / Escherichia coli (大腸菌)手法 / / 解像度 : 3.79 Å Brodolin K 資金援助 Organization Grant number 国 Agence Nationale de la Recherche (ANR) ANR-20-CE44-0020-01 Agence Nationale de la Recherche (ANR) ANR-16-CE11-0025 Instruct-ERIC Center (Strasbourg Centre) 19348
ジャーナル : Nucleic Acids Res / 年 : 2025タイトル : Single-stranded DNA drives σ subunit loading onto mycobacterial RNA polymerase to unlock initiation-competent conformations.著者 : Rishi Kishore Vishwakarma / Nils Marechal / Zakia Morichaud / Mickaël Blaise / Emmanuel Margeat / Konstantin Brodolin / 要旨 : Initiation of transcription requires the formation of the "open" promoter complex (RPo). For this, the σ subunit of bacterial RNA polymerase (RNAP) binds to the nontemplate strand of the -10 element ... Initiation of transcription requires the formation of the "open" promoter complex (RPo). For this, the σ subunit of bacterial RNA polymerase (RNAP) binds to the nontemplate strand of the -10 element sequence of promoters and nucleates DNA unwinding. This is accompanied by a cascade of conformational changes on RNAP, the exact mechanics of which remains elusive. Here, using single-molecule Förster resonance energy transfer and cryo-electron microscopy, we explored the conformational landscape of RNAP from the human pathogen Mycobacterium tuberculosis upon binding to a single-stranded DNA (ssDNA) fragment that includes the -10 element sequence (-10 ssDNA). We found that like the transcription activator RNAP-binding protein A, -10 ssDNA induced σ subunit loading onto the DNA/RNA channels of RNAP. This triggered RNAP clamp closure and unswiveling that are required for RPo formation and RNA synthesis initiation. Our results reveal a mechanism of ssDNA-guided RNAP maturation and identify the σ subunit as a regulator of RNAP conformational dynamics. 履歴 登録 2024年5月31日 - ヘッダ(付随情報) 公開 2025年4月2日 - マップ公開 2025年4月2日 - 更新 2025年5月7日 - 現状 2025年5月7日 処理サイト : PDBe / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報
 マップデータ
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Mycobacterium tuberculosis H37Rv (結核菌) /
Mycobacterium tuberculosis H37Rv (結核菌) / 
 データ登録者
データ登録者 フランス, 3件
フランス, 3件  引用
引用 ジャーナル: Nucleic Acids Res / 年: 2025
ジャーナル: Nucleic Acids Res / 年: 2025
 構造の表示
構造の表示 ダウンロードとリンク
ダウンロードとリンク emd_50514.map.gz
emd_50514.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-50514-v30.xml
emd-50514-v30.xml emd-50514.xml
emd-50514.xml EMDBヘッダ
EMDBヘッダ emd_50514_fsc.xml
emd_50514_fsc.xml FSCデータファイル
FSCデータファイル emd_50514.png
emd_50514.png emd-50514.cif.gz
emd-50514.cif.gz emd_50514_additional_1.map.gz
emd_50514_additional_1.map.gz emd_50514_half_map_1.map.gz
emd_50514_half_map_1.map.gz emd_50514_half_map_2.map.gz
emd_50514_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-50514
http://ftp.pdbj.org/pub/emdb/structures/EMD-50514 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-50514
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-50514 emd_50514_validation.pdf.gz
emd_50514_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_50514_full_validation.pdf.gz
emd_50514_full_validation.pdf.gz emd_50514_validation.xml.gz
emd_50514_validation.xml.gz emd_50514_validation.cif.gz
emd_50514_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-50514
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-50514 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-50514
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-50514 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_50514.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_50514.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素 Mycobacterium tuberculosis H37Rv (結核菌)
Mycobacterium tuberculosis H37Rv (結核菌) Mycobacterium tuberculosis H37Rv (結核菌)
Mycobacterium tuberculosis H37Rv (結核菌)
 Mycobacterium tuberculosis H37Rv (結核菌)
Mycobacterium tuberculosis H37Rv (結核菌)
 Mycobacterium tuberculosis H37Rv (結核菌)
Mycobacterium tuberculosis H37Rv (結核菌)
 Mycobacterium tuberculosis H37Rv (結核菌)
Mycobacterium tuberculosis H37Rv (結核菌)
 Mycobacterium tuberculosis H37Rv (結核菌)
Mycobacterium tuberculosis H37Rv (結核菌)

 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)