[日本語] English
 万見
万見- EMDB-47148: CRYO-EM STRUCTURE OF SACCHAROMYCES CEREVISIAE RAT1-RAI1-RTT103 COMPLEX -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CRYO-EM STRUCTURE OF SACCHAROMYCES CEREVISIAE RAT1-RAI1-RTT103 COMPLEX | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | Nuclease / Complex / Transcription termination / TRANSCRIPTION | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報RNA polymerase II termination complex / sno(s)RNA processing / positive regulation of termination of RNA polymerase II transcription / RNA NAD+-cap (NAD+-forming) hydrolase activity / termination of RNA polymerase II transcription, poly(A)-coupled / Las1 complex / termination of RNA polymerase II transcription, exosome-dependent / Butyrate Response Factor 1 (BRF1) binds and destabilizes mRNA / Tristetraprolin (TTP, ZFP36) binds and destabilizes mRNA / phosphodiesterase decapping endonuclease activity ...RNA polymerase II termination complex / sno(s)RNA processing / positive regulation of termination of RNA polymerase II transcription / RNA NAD+-cap (NAD+-forming) hydrolase activity / termination of RNA polymerase II transcription, poly(A)-coupled / Las1 complex / termination of RNA polymerase II transcription, exosome-dependent / Butyrate Response Factor 1 (BRF1) binds and destabilizes mRNA / Tristetraprolin (TTP, ZFP36) binds and destabilizes mRNA / phosphodiesterase decapping endonuclease activity / deadenylation-independent decapping of nuclear-transcribed mRNA / mRNA 5'-diphosphatase activity / RNA polymerase II C-terminal domain phosphoserine binding / NAD-cap decapping / nuclear polyadenylation-dependent rRNA catabolic process / 5'-3' RNA exonuclease activity / nuclear mRNA surveillance / transposable element silencing / maturation of 5.8S rRNA from tricistronic rRNA transcript (SSU-rRNA, 5.8S rRNA, LSU-rRNA) / mRNA 3'-end processing / RNA polymerase II transcribes snRNA genes / cleavage in ITS2 between 5.8S rRNA and LSU-rRNA of tricistronic rRNA transcript (SSU-rRNA, 5.8S rRNA, LSU-rRNA) / RNA polymerase II complex binding / nuclear-transcribed mRNA catabolic process / negative regulation of transcription elongation by RNA polymerase II / 加水分解酵素; 酸無水物に作用; リン含有酸無水物に作用 / enzyme regulator activity / maturation of LSU-rRNA from tricistronic rRNA transcript (SSU-rRNA, 5.8S rRNA, LSU-rRNA) / mRNA splicing, via spliceosome / mRNA processing / rRNA processing / site of double-strand break / 加水分解酵素; エステル加水分解酵素; 5'-リン酸モノエステル産生エキソリボヌクレアーゼ / rRNA binding / nucleotide binding / mRNA binding / chromatin / mitochondrion / DNA binding / RNA binding / metal ion binding / nucleus / cytosol 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  | ||||||||||||
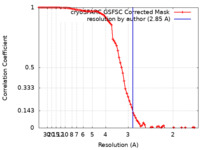
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.85 Å | ||||||||||||
 データ登録者 データ登録者 | Chu HF / Tong L | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2025 ジャーナル: Nat Commun / 年: 2025タイトル: Molecular basis for the interaction between Saccharomyces cerevisiae Rtt103 and the Rat1-Rai1 complex. 著者: Hsu-Feng Chu / Liang Tong /  要旨: The Rat1 5'-3' exoribonuclease together with its partner Rai1 have important roles in Saccharomyces cerevisiae RNA polymerase II transcription termination. Rtt103 copurifies with Rat1-Rai1 in S. ...The Rat1 5'-3' exoribonuclease together with its partner Rai1 have important roles in Saccharomyces cerevisiae RNA polymerase II transcription termination. Rtt103 copurifies with Rat1-Rai1 in S. cerevisiae, but its mechanism of interaction with them is not known. We report here the cryo-EM structure of the S. cerevisiae Rat1-Rai1-Rtt103 ternary complex at 2.9 Å resolution. We found that a short segment of Rtt103 is in close contact with Rai1, while the rest of Rtt103, including its RNA polymerase II C-terminal domain interaction domain, shows no interactions with Rai1 or Rat1. This is in contrast to the observations on the Komagataella phaffii Rat1-Rai1-Rtt103 complex, where only the RNA polymerase II C-terminal domain interaction domain of Rtt103 has contacts with Rai1. Our structure reveals that S. cerevisiae Rtt103 Pro261 and Tyr263 have important contacts with Rai1, and we show that the P261G/Y263A mutation of Rtt103 blocks the interaction with Rat1-Rai1. Our structure suggests that, in yeast, this segment of Rtt103, which we have named the Rai1 interaction segment, likely helps the recruitment of Rat1-Rai1 to RNA polymerase II for termination. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_47148.map.gz emd_47148.map.gz | 59.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-47148-v30.xml emd-47148-v30.xml emd-47148.xml emd-47148.xml | 20.2 KB 20.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_47148_fsc.xml emd_47148_fsc.xml | 8.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_47148.png emd_47148.png | 104.4 KB | ||
| Filedesc metadata |  emd-47148.cif.gz emd-47148.cif.gz | 7.4 KB | ||
| その他 |  emd_47148_half_map_1.map.gz emd_47148_half_map_1.map.gz emd_47148_half_map_2.map.gz emd_47148_half_map_2.map.gz | 59.5 MB 59.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-47148 http://ftp.pdbj.org/pub/emdb/structures/EMD-47148 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-47148 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-47148 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_47148_validation.pdf.gz emd_47148_validation.pdf.gz | 824.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_47148_full_validation.pdf.gz emd_47148_full_validation.pdf.gz | 823.7 KB | 表示 | |
| XML形式データ |  emd_47148_validation.xml.gz emd_47148_validation.xml.gz | 16.4 KB | 表示 | |
| CIF形式データ |  emd_47148_validation.cif.gz emd_47148_validation.cif.gz | 21.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-47148 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-47148 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-47148 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-47148 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  9dsoMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_47148.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_47148.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.826 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_47148_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_47148_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Rat1-Rai1-Rtt103 complex
| 全体 | 名称: Rat1-Rai1-Rtt103 complex |
|---|---|
| 要素 |
|
-超分子 #1: Rat1-Rai1-Rtt103 complex
| 超分子 | 名称: Rat1-Rai1-Rtt103 complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: 5'-3' exoribonuclease 2
| 分子 | 名称: 5'-3' exoribonuclease 2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO EC番号: 加水分解酵素; エステル加水分解酵素; 5'-リン酸モノエステル産生エキソリボヌクレアーゼ |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 114.204031 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MGVPSFFRWL SRKYPKIISP VLEEQPQIVD GVILPLDYSA SNPNGELDNL YLDMNGIVHP CSHPENKPPP ETEDEMLLAV FEYTNRVLN MARPRKVLVM AVDGVAPRAK MNQQRARRFR SARDAQIENE AREEIMRQRE EVGEIIDDAV RNKKTWDSNA I TPGTPFMD ...文字列: MGVPSFFRWL SRKYPKIISP VLEEQPQIVD GVILPLDYSA SNPNGELDNL YLDMNGIVHP CSHPENKPPP ETEDEMLLAV FEYTNRVLN MARPRKVLVM AVDGVAPRAK MNQQRARRFR SARDAQIENE AREEIMRQRE EVGEIIDDAV RNKKTWDSNA I TPGTPFMD KLAAALRYWT AFKLATDPGW KNLQVIISDA TVPGEGEHKI MNFIRSQRAD PEYNPNTTHC IYGLDADLIF LG LATHEPH FKILREDVFA QDNRKRNNLK DTINMTEEEK QFLQKQNSEQ PFLWLHINVL REYLSAELWV PGLPFTFDLE RAI DDWVFM CFFCGNDFLP HLPCLDVREN SIDILLDIWK VVLPKLKTYM TCDGVLNLPS VETLLQHLGS REGDIFKTRH IQEA RKKEA FERRKAQKNM SKGQDRHPTV ATEQLQMYDT QGNLAKGSWN LTTSDMVRLK KELMLANEGN EEAIAKVKQQ SDKNN ELMK DISKEEIDDA VSKANKTNFN LAEVMKQKII NKKHRLEKDN EEEEIAKDSK KVKTEKAESE CDLDAEIKDE IVADVN DRE NSETTEVSRD SPVHSTVNVS EGPKNGVFDT DEFVKLFEPG YHERYYTAKF HVTPQDIEQL RKDMVKCYIE GVAWVLM YY YQGCASWNWF YPYHYAPLAT DFHGFSHLEI KFEEGTPFLP YEQLMSVLPA ASGHALPKIF RSLMSEPDSE IIDFYPEE F PIDMNGKKMS WQGIALLPFI DQDRLLTAVR AQYPLLSDAE RARNIRGEPV LLISNKNANY ERFSKKLYSK ENNNNNVVV KFQHFKSGLS GIVSKDVEGF ELNGKIVCPI QGGSLPNLST TLILKMSYRL IPLPSRNKSI ILNGFIPSEP VLTAYDLDSI MYKYNNQNY SRRWNFGNDL KQNIVPVGPK GITQYKPRTG GYRAFFYFAE LSRNNVQPAH NYGRNSYNSQ PGFNNSRYDG G NNNYRQNS NYRNNKYSGN RNSLEHHHHH H UniProtKB: 5'-3' exoribonuclease 2 |
-分子 #2: Decapping nuclease RAI1
| 分子 | 名称: Decapping nuclease RAI1 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO EC番号: 加水分解酵素; 酸無水物に作用; リン含有酸無水物に作用 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 44.571445 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MGVSANLFVK QRGSTTALKQ PKEIGFYSRT KDEEYLISDD TNLNYYYLPD AELDRKLDLS SGFQKFKDYY KDFEDRCSLR GLLETIESS ERHKGKKINA DIITFRGIAR KLISCAFDSP SFNTVDLRIV SFNGQLFIKE VPEAVNAAKA SSATEAGRNI N QDLNVFTG ...文字列: MGVSANLFVK QRGSTTALKQ PKEIGFYSRT KDEEYLISDD TNLNYYYLPD AELDRKLDLS SGFQKFKDYY KDFEDRCSLR GLLETIESS ERHKGKKINA DIITFRGIAR KLISCAFDSP SFNTVDLRIV SFNGQLFIKE VPEAVNAAKA SSATEAGRNI N QDLNVFTG YKFETLATLS NPLQYTPREV IEKRTKRIVS HGDEYISVVR TGVGNCKLIL GAEVDCIFDF KENGRDNLKH YA ELKCTQQ VANISDTHKF ERKLFRTWLQ CFLVGIPRII YGFKDDHYVL KTVEEFSTEE VPVLLKNNNP QVGSACLEAI KWY GLLTEW LLKMIPRDED PHSQIRAFKL VFENNHLRLS EIEESDEEYS GLIDGEHILS NGFKEWRKSL K UniProtKB: Decapping nuclease RAI1 |
-分子 #3: Regulator of Ty1 transposition protein 103
| 分子 | 名称: Regulator of Ty1 transposition protein 103 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 48.725473 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MGSSHHHHHH SSGLVPRGSH MPFSSEQFTT KLNTLEDSQE SISSASKWLL LQYRDAPKVA EMWKEYMLRP SVNTRRKLLG LYLMNHVVQ QAKGQKIIQF QDSFGKVAAE VLGRINQEFP RDLKKKLSRV VNILKERNIF SKQVVNDIER SLKTESSPVE A LVLPQKLK ...文字列: MGSSHHHHHH SSGLVPRGSH MPFSSEQFTT KLNTLEDSQE SISSASKWLL LQYRDAPKVA EMWKEYMLRP SVNTRRKLLG LYLMNHVVQ QAKGQKIIQF QDSFGKVAAE VLGRINQEFP RDLKKKLSRV VNILKERNIF SKQVVNDIER SLKTESSPVE A LVLPQKLK DFAKDYEKLV KMHHNVCAMK MRFDKSSDEL DPSSSVYEEN FKTISKIGNM AKDIINESIL KRESGIHKLQ ST LDDEKRH LDEEQNMLSE IEFVLSAKDP SRLNKNVDED NIIPTYEVGD GDDDDDDGDN DDDDDDDDDD KNYDDRSNDS NYG VTNIST TDKKNEVVEK TDSEHKNSTH NPSDNQFGMK RTHDMIGHDD ANDIPEKKVH LDSKTSEDGT FNSEDGHYEL DIEG HVGAQ TDEGVENSGG VSSSIQDLLS KLAN UniProtKB: Regulator of Ty1 transposition protein 103 |
-分子 #4: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 4 / コピー数: 1 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.2 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
| |||||||||
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 400 / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 14 sec. / 前処理 - 雰囲気: OTHER | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 293 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 49.27 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.6 µm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: AlphaFold / Chain - Initial model type: in silico model |
|---|---|
| 精密化 | プロトコル: FLEXIBLE FIT |
| 得られたモデル |  PDB-9dso: |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)