+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4616 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structural basis of cohesin ring opening | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Chromatin / genome segregation / cohesin / CELL CYCLE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Establishment of Sister Chromatid Cohesion / Resolution of Sister Chromatid Cohesion / meiotic cohesin complex / establishment of meiotic sister chromatid cohesion / cohesin complex / mitotic cohesin complex / synaptonemal complex assembly / SUMOylation of DNA damage response and repair proteins / meiotic sister chromatid cohesion / replication-born double-strand break repair via sister chromatid exchange ...Establishment of Sister Chromatid Cohesion / Resolution of Sister Chromatid Cohesion / meiotic cohesin complex / establishment of meiotic sister chromatid cohesion / cohesin complex / mitotic cohesin complex / synaptonemal complex assembly / SUMOylation of DNA damage response and repair proteins / meiotic sister chromatid cohesion / replication-born double-strand break repair via sister chromatid exchange / establishment of mitotic sister chromatid cohesion / mitotic chromosome condensation / reciprocal meiotic recombination / sister chromatid cohesion / mitotic sister chromatid cohesion / protein acetylation / chromosome, centromeric region / condensed nuclear chromosome / G2/M transition of mitotic cell cycle / double-strand break repair / double-stranded DNA binding / cell division / apoptotic process / DNA damage response / chromatin binding / protein kinase binding / ATP hydrolysis activity / mitochondrion / DNA binding / ATP binding / identical protein binding / nucleus 類似検索 - 分子機能 | |||||||||
| 生物種 |  Chaetomium thermophilum (strain DSM 1495 / CBS 144.50 / IMI 039719) (菌類) / Chaetomium thermophilum (strain DSM 1495 / CBS 144.50 / IMI 039719) (菌類) /   Chaetomium thermophilum var. thermophilum DSM 1495 (菌類) / Chaetomium thermophilum var. thermophilum DSM 1495 (菌類) /  | |||||||||
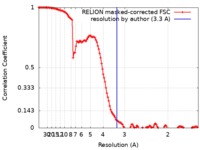
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Panne D / Muir KW | |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2020 ジャーナル: Nat Struct Mol Biol / 年: 2020タイトル: The structure of the cohesin ATPase elucidates the mechanism of SMC-kleisin ring opening. 著者: Kyle W Muir / Yan Li / Felix Weis / Daniel Panne /    要旨: Genome regulation requires control of chromosome organization by SMC-kleisin complexes. The cohesin complex contains the Smc1 and Smc3 subunits that associate with the kleisin Scc1 to form a ring- ...Genome regulation requires control of chromosome organization by SMC-kleisin complexes. The cohesin complex contains the Smc1 and Smc3 subunits that associate with the kleisin Scc1 to form a ring-shaped complex that can topologically engage chromatin to regulate chromatin structure. Release from chromatin involves opening of the ring at the Smc3-Scc1 interface in a reaction that is controlled by acetylation and engagement of the Smc ATPase head domains. To understand the underlying molecular mechanisms, we have determined the 3.2-Å resolution cryo-electron microscopy structure of the ATPγS-bound, heterotrimeric cohesin ATPase head module and the 2.1-Å resolution crystal structure of a nucleotide-free Smc1-Scc1 subcomplex from Saccharomyces cerevisiae and Chaetomium thermophilium. We found that ATP-binding and Smc1-Smc3 heterodimerization promote conformational changes within the ATPase that are transmitted to the Smc coiled-coil domains. Remodeling of the coiled-coil domain of Smc3 abrogates the binding surface for Scc1, thus leading to ring opening at the Smc3-Scc1 interface. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4616.map.gz emd_4616.map.gz | 9.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4616-v30.xml emd-4616-v30.xml emd-4616.xml emd-4616.xml | 20.2 KB 20.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_4616_fsc.xml emd_4616_fsc.xml | 12.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4616.png emd_4616.png | 53 KB | ||
| Filedesc metadata |  emd-4616.cif.gz emd-4616.cif.gz | 7.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4616 http://ftp.pdbj.org/pub/emdb/structures/EMD-4616 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4616 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4616 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_4616_validation.pdf.gz emd_4616_validation.pdf.gz | 189.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_4616_full_validation.pdf.gz emd_4616_full_validation.pdf.gz | 188.8 KB | 表示 | |
| XML形式データ |  emd_4616_validation.xml.gz emd_4616_validation.xml.gz | 501 B | 表示 | |
| CIF形式データ |  emd_4616_validation.cif.gz emd_4616_validation.cif.gz | 448 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4616 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4616 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4616 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4616 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4616.map.gz / 形式: CCP4 / 大きさ: 163.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4616.map.gz / 形式: CCP4 / 大きさ: 163.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.81 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Cohesin ATPase head module
| 全体 | 名称: Cohesin ATPase head module |
|---|---|
| 要素 |
|
-超分子 #1: Cohesin ATPase head module
| 超分子 | 名称: Cohesin ATPase head module / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|
-超分子 #2: Structural maintenance of chromosomes protein,Structural maintena...
| 超分子 | 名称: Structural maintenance of chromosomes protein,Structural maintenance of chromosomes protein タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Chaetomium thermophilum (strain DSM 1495 / CBS 144.50 / IMI 039719) (菌類) Chaetomium thermophilum (strain DSM 1495 / CBS 144.50 / IMI 039719) (菌類) |
-超分子 #3: Sister chromatid cohesion protein 1, Structural maintenance of ch...
| 超分子 | 名称: Sister chromatid cohesion protein 1, Structural maintenance of chromosomes protein 3,Structural maintenance of chromosomes protein 3 タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2-#4 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Structural maintenance of chromosomes protein,Structural maintena...
| 分子 | 名称: Structural maintenance of chromosomes protein,Structural maintenance of chromosomes protein タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Chaetomium thermophilum var. thermophilum DSM 1495 (菌類) Chaetomium thermophilum var. thermophilum DSM 1495 (菌類) |
| 分子量 | 理論値: 27.570475 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGKLIRLELF NFKSYKGHHT LLFGDSYFTS IIGPNGSGKS NSMDAISFVL GIKSSHLRSS NLRDLIYRGR VMKTSKIQDD GTTAPATNG DVNGYENGDA GDDEDTSQRT SRNDPKTAWV MAVYEDDAGE LHRWKRTITA NGTSEYRIND RVVNAQQYNE A LEKENILI ...文字列: MGKLIRLELF NFKSYKGHHT LLFGDSYFTS IIGPNGSGKS NSMDAISFVL GIKSSHLRSS NLRDLIYRGR VMKTSKIQDD GTTAPATNG DVNGYENGDA GDDEDTSQRT SRNDPKTAWV MAVYEDDAGE LHRWKRTITA NGTSEYRIND RVVNAQQYNE A LEKENILI KARNFLVFQG DVEAIASQSP QDLTRLIEQI SGSLEYKEEY ERLEEEVRQA TEEQAYKLQR RRAANSEIKQ YM EQ UniProtKB: Structural maintenance of chromosomes protein, Structural maintenance of chromosomes protein |
-分子 #2: Sister chromatid cohesion protein 1
| 分子 | 名称: Sister chromatid cohesion protein 1 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 9.48994 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MASKAIVQMA KILRKELSEE KEVIFTDVLK SQANTEPENI TKREASRGFF DILSLATEGC IGLSQTEAFG NIKIDAKPAL FERFI UniProtKB: Sister chromatid cohesion protein 1 |
-分子 #3: Structural maintenance of chromosomes protein 3,Structural mainte...
| 分子 | 名称: Structural maintenance of chromosomes protein 3,Structural maintenance of chromosomes protein 3 タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 61.051789 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAYIKRVIIK GFKTYRNETI IDNFSPHQNV IIGSNGSGKS NFFAAIRFVL SDDYSNLKRE ERQGLIHQGS GGSVMSASVE IVFHDPDHS MILPSGVLSR GDDEVTIRRT VGLKKDDYQL NDRNVTKGDI VRMLETAGFS MNNPYNIVPQ GKIVALTNAK D KERLQLLE ...文字列: MAYIKRVIIK GFKTYRNETI IDNFSPHQNV IIGSNGSGKS NFFAAIRFVL SDDYSNLKRE ERQGLIHQGS GGSVMSASVE IVFHDPDHS MILPSGVLSR GDDEVTIRRT VGLKKDDYQL NDRNVTKGDI VRMLETAGFS MNNPYNIVPQ GKIVALTNAK D KERLQLLE DVVGAKSFEV KLKASLKKME ETEQKKIQIN KEMGELNSKL SEMEQERKEL EKYNELERNR KIYQFTLYDR EL NEVINQM ERLDGDYNNT VYSSESSKHP TSLVPRGSDI TSDQLLQRLN DMNTEISGLK NVNKRAFENF KKFNERRKDL AER ASELDE SKDSIQDLIV KLKQQKVNAV DSTFQKVSEN FEAVFERLVP RGTAKLIIHR KNDNANDHDE SIDVDMDAES NESQ NGKDS EIMYTGVSIS VSFNSKQNEQ LHVEQLSGGQ KTVCAIALIL AIQMVDPASF YLFDEIDACL DKQYRTAVAT LLKEL SKNA QFICTTFRTD MLQVADKFFR VKYECKISTV IEVNREEAIG FIRGSNKFAE V UniProtKB: Structural maintenance of chromosomes protein 3, Structural maintenance of chromosomes protein 3 |
-分子 #4: Sister chromatid cohesion protein 1,Structural maintenance of chr...
| 分子 | 名称: Sister chromatid cohesion protein 1,Structural maintenance of chromosomes protein タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Chaetomium thermophilum var. thermophilum DSM 1495 (菌類) Chaetomium thermophilum var. thermophilum DSM 1495 (菌類) |
| 分子量 | 理論値: 42.292125 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MVTENPQRLT VLRLATNKGP LAQIWLASNM SNIPRGSVIQ THIAESAKEI AKASGSDDES GDNEYITLRT SGELLQGIVR VYSKQATFL LTDIKDTLTK ISMLFKTSQK MTSTVNRLNT VTRVHQLMLE DAVTEREVLV TPGLEFLDDT TIPVGLMAQE N PNLRAMDR ...文字列: MVTENPQRLT VLRLATNKGP LAQIWLASNM SNIPRGSVIQ THIAESAKEI AKASGSDDES GDNEYITLRT SGELLQGIVR VYSKQATFL LTDIKDTLTK ISMLFKTSQK MTSTVNRLNT VTRVHQLMLE DAVTEREVLV TPGLEFLDDT TIPVGLMAQE N PNLRAMDR LDHVRKQLEQ TEQEFEASKA KLRQARESFQ AVKQKRLELF NKAFTHIQEQ ITHVYKELTR SEAYPLGGQA YL DIEEDTD TPFLSGVKYH AMPPCKRFRD MEHLSGGEKT MAALALLFAI HSYQPSPFFV LDEVDCALDN ANVEKIKKYI REH AGPGMQ FIVISLKPAL FQASESLIGV YRDQEANTSR TLTLDLRKYR HHHHHH UniProtKB: Sister chromatid cohesion protein 1, Structural maintenance of chromosomes protein |
-分子 #5: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 5 / コピー数: 2 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #6: PHOSPHOTHIOPHOSPHORIC ACID-ADENYLATE ESTER
| 分子 | 名称: PHOSPHOTHIOPHOSPHORIC ACID-ADENYLATE ESTER / タイプ: ligand / ID: 6 / コピー数: 2 / 式: AGS |
|---|---|
| 分子量 | 理論値: 523.247 Da |
| Chemical component information |  ChemComp-AGS: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 42.08 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)