登録情報 データベース : EMDB / ID : EMD-45805タイトル Infectious bronchitis virus core polymerase complex 複合体 : Core polymerase complex of the infectious bronchitis virus composed of nsp12, nsp8, nsp7, and an dsRNA substrateタンパク質・ペプチド : RNA-directed RNA polymerase nsp12タンパク質・ペプチド : Non-structural protein 8タンパク質・ペプチド : Non-structural protein 7RNA : RNA PrimerRNA : RNA templateリガンド : ZINC ION / / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
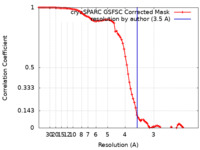
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 / 手法 / / 解像度 : 3.5 Å Hoferle PJ / Anderson TK / Kirchdoerfer RN 資金援助 Organization Grant number 国 National Institutes of Health/National Institute Of Allergy and Infectious Diseases (NIH/NIAID) AI158463
ジャーナル : bioRxiv / 年 : 2024タイトル : A genus-specific nsp12 region impacts polymerase assembly in Alpha- and Gammacoronaviruses.著者 : Peter J Hoferle / Thomas K Anderson / Robert N Kirchdoerfer 要旨 : Coronavirus relevancy for human health has surged over the past 20 years as they have a propensity for spillover into humans from animal reservoirs resulting in pandemics such as COVID-19. The ... Coronavirus relevancy for human health has surged over the past 20 years as they have a propensity for spillover into humans from animal reservoirs resulting in pandemics such as COVID-19. The diversity within the subfamily and high infection frequency in animal species worldwide creates a looming threat that calls for research across all genera within the subfamily. We sought to contribute to the limited structural knowledge within the genera and determined the structure of the viral core replication-transcription complex (RTC) from Infectious Bronchitis Virus (IBV) using single-particle cryo-EM. Comparison between our IBV structure with published RTC structures from other genera reveals structural differences across genera. Using biochemical assays, we characterized these differences and revealed their differing involvement in core RTC formation across different genera. Our findings highlight the value of cross-genera studies, as they show genera specific features in coronavirus genome replication. A broader knowledge of coronavirus replication will better prepare us for future coronavirus spillovers. 履歴 登録 2024年7月18日 - ヘッダ(付随情報) 公開 2024年7月31日 - マップ公開 2024年7月31日 - 更新 2025年6月4日 - 現状 2025年6月4日 処理サイト : RCSB / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報
 マップデータ
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Infectious bronchitis virus (伝染性気管支炎ウイルス) /
Infectious bronchitis virus (伝染性気管支炎ウイルス) / 
 データ登録者
データ登録者 米国, 1件
米国, 1件  引用
引用 ジャーナル: bioRxiv / 年: 2024
ジャーナル: bioRxiv / 年: 2024 構造の表示
構造の表示 ダウンロードとリンク
ダウンロードとリンク emd_45805.map.gz
emd_45805.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-45805-v30.xml
emd-45805-v30.xml emd-45805.xml
emd-45805.xml EMDBヘッダ
EMDBヘッダ emd_45805_fsc.xml
emd_45805_fsc.xml FSCデータファイル
FSCデータファイル emd_45805.png
emd_45805.png emd-45805.cif.gz
emd-45805.cif.gz emd_45805_half_map_1.map.gz
emd_45805_half_map_1.map.gz emd_45805_half_map_2.map.gz
emd_45805_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-45805
http://ftp.pdbj.org/pub/emdb/structures/EMD-45805 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-45805
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-45805 emd_45805_validation.pdf.gz
emd_45805_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_45805_full_validation.pdf.gz
emd_45805_full_validation.pdf.gz emd_45805_validation.xml.gz
emd_45805_validation.xml.gz emd_45805_validation.cif.gz
emd_45805_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-45805
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-45805 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-45805
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-45805
 F&H 検索
F&H 検索 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_45805.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_45805.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素 Infectious bronchitis virus (伝染性気管支炎ウイルス)
Infectious bronchitis virus (伝染性気管支炎ウイルス) Infectious bronchitis virus (伝染性気管支炎ウイルス)
Infectious bronchitis virus (伝染性気管支炎ウイルス)
 Infectious bronchitis virus (伝染性気管支炎ウイルス)
Infectious bronchitis virus (伝染性気管支炎ウイルス)
 Infectious bronchitis virus (伝染性気管支炎ウイルス)
Infectious bronchitis virus (伝染性気管支炎ウイルス)


 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)