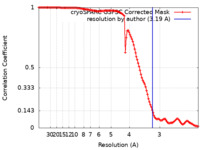
登録情報 データベース : EMDB / ID : EMD-45655タイトル Cryo-EM structure of alpha5beta1 integrin in complex with NeoNectin Sharpened map 3.19Angstrom 複合体 : Protein complex of wild type integrin alpha-5 beta-1 with computationally designed protein NeoNectinタンパク質・ペプチド : Integrin alpha-5タンパク質・ペプチド : Integrin beta-1タンパク質・ペプチド : NeoNectinリガンド : CALCIUM IONリガンド : 2-acetamido-2-deoxy-beta-D-glucopyranoseリガンド : MANGANESE (II) ION / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Homo sapiens (ヒト) / synthetic construct (人工物) 手法 / / 解像度 : 3.19 Å Werther R / Nguyen A / Estrada Alamo KA / Wang X / Campbell MG 資金援助 Organization Grant number 国 The Pew Charitable Trusts National Institutes of Health/National Institute of General Medical Sciences (NIH/NIGMS) R35 GM147414-01 National Institutes of Health/National Cancer Institute (NIH/NCI) P30 CA015704-40; RRID:SCR_022611 Other private Seattle Cancer Consortium Safeway Inc Pilot Grant
ジャーナル : bioRxiv / 年 : 2024タイトル : De Novo Design of Integrin α5β1 Modulating Proteins for Regenerative Medicine.著者: Xinru Wang / Jordi Guillem-Marti / Saurav Kumar / David S Lee / Daniel Cabrerizo-Aguado / Rachel Werther / Kevin Alexander Estrada Alamo / Yan Ting Zhao / Adam Nguyen / Irina Kopyeva / Buwei ... 著者 : Xinru Wang / Jordi Guillem-Marti / Saurav Kumar / David S Lee / Daniel Cabrerizo-Aguado / Rachel Werther / Kevin Alexander Estrada Alamo / Yan Ting Zhao / Adam Nguyen / Irina Kopyeva / Buwei Huang / Jing Li / Yuxin Hao / Xinting Li / Aritza Brizuela-Velasco / Analisa Murray / Stacey Gerben / Anindya Roy / Cole A DeForest / Timothy Springer / Hannele Ruohola-Baker / Jonathan A Cooper / Melody G Campbell / Jose Maria Manero / Maria-Pau Ginebra / David Baker / 要旨 : Integrin α5β1 is crucial for cell attachment and migration in development and tissue regeneration, and α5β1 binding proteins could have considerable utility in regenerative medicine and next- ... Integrin α5β1 is crucial for cell attachment and migration in development and tissue regeneration, and α5β1 binding proteins could have considerable utility in regenerative medicine and next-generation therapeutics. We use computational protein design to create de novo α5β1-specific modulating miniprotein binders, called NeoNectins, that bind to and stabilize the open state of α5β1. When immobilized onto titanium surfaces and throughout 3D hydrogels, the NeoNectins outperform native fibronectin and RGD peptide in enhancing cell attachment and spreading, and NeoNectin-grafted titanium implants outperformed fibronectin and RGD-grafted implants in animal models in promoting tissue integration and bone growth. NeoNectins should be broadly applicable for tissue engineering and biomedicine. 履歴 登録 2024年7月9日 - ヘッダ(付随情報) 公開 2024年7月17日 - マップ公開 2024年7月17日 - 更新 2025年9月10日 - 現状 2025年9月10日 処理サイト : RCSB / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報
 マップデータ
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト) / synthetic construct (人工物)
Homo sapiens (ヒト) / synthetic construct (人工物) データ登録者
データ登録者 米国, 4件
米国, 4件  引用
引用

 構造の表示
構造の表示 ダウンロードとリンク
ダウンロードとリンク emd_45655.map.gz
emd_45655.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-45655-v30.xml
emd-45655-v30.xml emd-45655.xml
emd-45655.xml EMDBヘッダ
EMDBヘッダ emd_45655_fsc.xml
emd_45655_fsc.xml FSCデータファイル
FSCデータファイル emd_45655.png
emd_45655.png emd_45655_msk_1.map
emd_45655_msk_1.map マスクマップ
マスクマップ emd-45655.cif.gz
emd-45655.cif.gz emd_45655_additional_1.map.gz
emd_45655_additional_1.map.gz emd_45655_additional_2.map.gz
emd_45655_additional_2.map.gz emd_45655_additional_3.map.gz
emd_45655_additional_3.map.gz emd_45655_half_map_1.map.gz
emd_45655_half_map_1.map.gz emd_45655_half_map_2.map.gz
emd_45655_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-45655
http://ftp.pdbj.org/pub/emdb/structures/EMD-45655 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-45655
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-45655 emd_45655_validation.pdf.gz
emd_45655_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_45655_full_validation.pdf.gz
emd_45655_full_validation.pdf.gz emd_45655_validation.xml.gz
emd_45655_validation.xml.gz emd_45655_validation.cif.gz
emd_45655_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-45655
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-45655 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-45655
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-45655 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_45655.map.gz / 形式: CCP4 / 大きさ: 166.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_45655.map.gz / 形式: CCP4 / 大きさ: 166.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) emd_45655_msk_1.map
emd_45655_msk_1.map 試料の構成要素
試料の構成要素 Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト)
 Homo sapiens (ヒト)
Homo sapiens (ヒト)


 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN ムービー
ムービー コントローラー
コントローラー
























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)