+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | LONP1 stall state bound to substrate and 4 ADPs | |||||||||
 マップデータ マップデータ | Sharpened map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | ATPase / protease / HYDROLASE | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
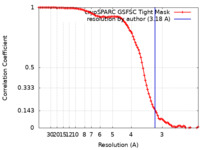
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.18 Å | |||||||||
 データ登録者 データ登録者 | Mindrebo JT / Lander GC | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: bioRxiv / 年: 2024 ジャーナル: bioRxiv / 年: 2024タイトル: Structural and mechanistic studies on human LONP1 redefine the hand-over-hand translocation mechanism. 著者: Jeffrey T Mindrebo / Gabriel C Lander /  要旨: AAA+ enzymes use energy from ATP hydrolysis to remodel diverse cellular targets. Structures of substrate-bound AAA+ complexes suggest that these enzymes employ a conserved hand-over-hand mechanism to ...AAA+ enzymes use energy from ATP hydrolysis to remodel diverse cellular targets. Structures of substrate-bound AAA+ complexes suggest that these enzymes employ a conserved hand-over-hand mechanism to thread substrates through their central pore. However, the fundamental aspects of the mechanisms governing motor function and substrate processing within specific AAA+ families remain unresolved. We used cryo-electron microscopy to structurally interrogate reaction intermediates from in vitro biochemical assays to inform the underlying regulatory mechanisms of the human mitochondrial AAA+ protease, LONP1. Our results demonstrate that substrate binding allosterically regulates proteolytic activity, and that LONP1 can adopt a configuration conducive to substrate translocation even when the ATPases are bound to ADP. These results challenge the conventional understanding of the hand-over-hand translocation mechanism, giving rise to an alternative model that aligns more closely with biochemical and biophysical data on related enzymes like ClpX, ClpA, the 26S proteasome, and Lon protease. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_45434.map.gz emd_45434.map.gz | 47.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-45434-v30.xml emd-45434-v30.xml emd-45434.xml emd-45434.xml | 19.9 KB 19.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_45434_fsc.xml emd_45434_fsc.xml | 10.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_45434.png emd_45434.png | 108.4 KB | ||
| Filedesc metadata |  emd-45434.cif.gz emd-45434.cif.gz | 5.8 KB | ||
| その他 |  emd_45434_additional_1.map.gz emd_45434_additional_1.map.gz emd_45434_half_map_1.map.gz emd_45434_half_map_1.map.gz emd_45434_half_map_2.map.gz emd_45434_half_map_2.map.gz | 46 MB 84.6 MB 84.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-45434 http://ftp.pdbj.org/pub/emdb/structures/EMD-45434 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-45434 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-45434 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_45434_validation.pdf.gz emd_45434_validation.pdf.gz | 1003.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_45434_full_validation.pdf.gz emd_45434_full_validation.pdf.gz | 1003.4 KB | 表示 | |
| XML形式データ |  emd_45434_validation.xml.gz emd_45434_validation.xml.gz | 17.7 KB | 表示 | |
| CIF形式データ |  emd_45434_validation.cif.gz emd_45434_validation.cif.gz | 22.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-45434 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-45434 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-45434 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-45434 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_45434.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_45434.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.15 Å | ||||||||||||||||||||
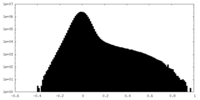
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Unsharpened map
| ファイル | emd_45434_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
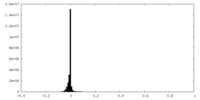
| 密度ヒストグラム |
-ハーフマップ: Half map B
| ファイル | emd_45434_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
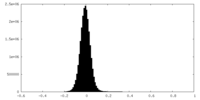
| 密度ヒストグラム |
-ハーフマップ: Half map A
| ファイル | emd_45434_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
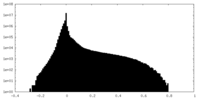
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : LONP1 bound to substrate and ADP
| 全体 | 名称: LONP1 bound to substrate and ADP |
|---|---|
| 要素 |
|
-超分子 #1: LONP1 bound to substrate and ADP
| 超分子 | 名称: LONP1 bound to substrate and ADP / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 583.95 KDa |
-分子 #1: Human mitochondrial Lon Protease homolog
| 分子 | 名称: Human mitochondrial Lon Protease homolog / タイプ: protein_or_peptide / ID: 1 / 詳細: homohexameric assembly / 光学異性体: LEVO / EC番号: endopeptidase La |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MHHHHHHENL YFQGAHMMTI PDVFPHLPLI AITRNPVFPR FIKIIEVKNK KLVELLRRKV RLAQPYVGVF LKRDDSNESD VVESLDEIYH TGTFAQIHEM QDLGDKLRMI VMGHRRVHIS RQLEVEPEEP EAENKHKPRR KSKRGKKEAE DELSARHPAE LAMEPTPELP ...文字列: MHHHHHHENL YFQGAHMMTI PDVFPHLPLI AITRNPVFPR FIKIIEVKNK KLVELLRRKV RLAQPYVGVF LKRDDSNESD VVESLDEIYH TGTFAQIHEM QDLGDKLRMI VMGHRRVHIS RQLEVEPEEP EAENKHKPRR KSKRGKKEAE DELSARHPAE LAMEPTPELP AEVLMVEVEN VVHEDFQVTE EVKALTAEIV KTIRDIIALN PLYRESVLQM MQAGQRVVDN PIYLSDMGAA LTGAESHELQ DVLEETNIPK RLYKALSLLK KEFELSKLQQ RLGREVEEKI KQTHRKYLLQ EQLKIIKKEL GLEKDDKDAI EEKFRERLKE LVVPKHVMDV VDEELSKLGL LDNHSSEFNV TRNYLDWLTS IPWGKYSNEN LDLARAQAVL EEDHYGMEDV KKRILEFIAV SQLRGSTQGK ILCFYGPPGV GKTSIARSIA RALNREYFRF SVGGMTDVAE IKGHRRTYVG AMPGKIIQCL KKTKTENPLI LIDEVDKIGR GYQGDPSSAL LELLDPEQNA NFLDHYLDVP VDLSKVLFIC TANVTDTIPE PLRDRMEMIN VSGYVAQEKL AIAERYLVPQ ARALCGLDES KAKLSSDVLT LLIKQYCRES GVRNLQKQVE KVLRKSAYKI VSGEAESVEV TPENLQDFVG KPVFTVERMY DVTPPGVVMG LAWTAMGGST LFVETSLRRP QDKDAKGDKD GSLEVTGQLG EVMKESARIA YTFARAFLMQ HAPANDYLVT SHIHLHVPEG ATPKDGPSAG CTIVTALLSL AMGRPVRQNL AMTGEVSLTG KILPVGGIKE KTIAAKRAGV TCIVLPAENK KDFYDLAAFI TEGLEVHFVE HYREIFDIAF PDEQAEALAV ER |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| |||||||||
| グリッド | モデル: Quantifoil / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 25 sec. / 前処理 - 雰囲気: OTHER | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK IV | |||||||||
| 詳細 | Sample was monodisperse with ~300 particles per image. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3840 pixel / デジタル化 - サイズ - 縦: 3712 pixel / 撮影したグリッド数: 1 / 実像数: 1944 / 平均露光時間: 9.8 sec. / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 倍率(補正後): 43478 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.6 µm / 最小 デフォーカス(公称値): 0.9 µm / 倍率(公称値): 36000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー








 Z
Z Y
Y X
X