+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 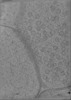 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-Electron Tomography of Cultured Neuronal Synapses | |||||||||
 マップデータ マップデータ | CryoCARE denoised tomogram of a cultured neuronal synapse. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Synapse / Neuron / Vesicles / EXOCYTOSIS | |||||||||
| 生物種 |  | |||||||||
| 手法 | 電子線トモグラフィー法 / クライオ電子顕微鏡法 | |||||||||
 データ登録者 データ登録者 | Held RG / Brunger AT / Liang J | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2024 ジャーナル: Proc Natl Acad Sci U S A / 年: 2024タイトル: Nanoscale architecture of synaptic vesicles and scaffolding complexes revealed by cryo-electron tomography. 著者: Richard G Held / Jiahao Liang / Axel T Brunger /  要旨: The spatial distribution of proteins and their arrangement within the cellular ultrastructure regulates the opening of α-amino-3-hydroxy-5-methyl-4-isoxazolepropionic acid (AMPA) receptors in ...The spatial distribution of proteins and their arrangement within the cellular ultrastructure regulates the opening of α-amino-3-hydroxy-5-methyl-4-isoxazolepropionic acid (AMPA) receptors in response to glutamate release at the synapse. Fluorescence microscopy imaging revealed that the postsynaptic density (PSD) and scaffolding proteins in the presynaptic active zone (AZ) align across the synapse to form a trans-synaptic "nanocolumn," but the relation to synaptic vesicle release sites is uncertain. Here, we employ focused-ion beam (FIB) milling and cryoelectron tomography to image synapses under near-native conditions. Improved image contrast, enabled by FIB milling, allows simultaneous visualization of supramolecular nanoclusters within the AZ and PSD and synaptic vesicles. Surprisingly, membrane-proximal synaptic vesicles, which fuse to release glutamate, are not preferentially aligned with AZ or PSD nanoclusters. These synaptic vesicles are linked to the membrane by peripheral protein densities, often consistent in size and shape with Munc13, as well as globular densities bridging the synaptic vesicle and plasma membrane, consistent with prefusion complexes of SNAREs, synaptotagmins, and complexin. Monte Carlo simulations of synaptic transmission events using biorealistic models guided by our tomograms predict that clustering AMPARs within PSD nanoclusters increases the variability of the postsynaptic response but not its average amplitude. Together, our data support a model in which synaptic strength is tuned at the level of single vesicles by the spatial relationship between scaffolding nanoclusters and single synaptic vesicle fusion sites. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_45161.map.gz emd_45161.map.gz | 103.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-45161-v30.xml emd-45161-v30.xml emd-45161.xml emd-45161.xml | 40.3 KB 40.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_45161.png emd_45161.png | 219.7 KB | ||
| Filedesc metadata |  emd-45161.cif.gz emd-45161.cif.gz | 5.4 KB | ||
| その他 |  emd_45161_additional_1.map.gz emd_45161_additional_1.map.gz emd_45161_additional_10.map.gz emd_45161_additional_10.map.gz emd_45161_additional_11.map.gz emd_45161_additional_11.map.gz emd_45161_additional_12.map.gz emd_45161_additional_12.map.gz emd_45161_additional_2.map.gz emd_45161_additional_2.map.gz emd_45161_additional_3.map.gz emd_45161_additional_3.map.gz emd_45161_additional_4.map.gz emd_45161_additional_4.map.gz emd_45161_additional_5.map.gz emd_45161_additional_5.map.gz emd_45161_additional_6.map.gz emd_45161_additional_6.map.gz emd_45161_additional_7.map.gz emd_45161_additional_7.map.gz emd_45161_additional_8.map.gz emd_45161_additional_8.map.gz emd_45161_additional_9.map.gz emd_45161_additional_9.map.gz | 318.8 MB 320.7 MB 289.7 MB 287.1 MB 291.3 MB 294.6 MB 300.6 MB 298.6 MB 297.4 MB 303.7 MB 311.8 MB 323.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-45161 http://ftp.pdbj.org/pub/emdb/structures/EMD-45161 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-45161 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-45161 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_45161_validation.pdf.gz emd_45161_validation.pdf.gz | 374.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_45161_full_validation.pdf.gz emd_45161_full_validation.pdf.gz | 373.9 KB | 表示 | |
| XML形式データ |  emd_45161_validation.xml.gz emd_45161_validation.xml.gz | 4.8 KB | 表示 | |
| CIF形式データ |  emd_45161_validation.cif.gz emd_45161_validation.cif.gz | 5.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-45161 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-45161 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-45161 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-45161 | HTTPS FTP |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_45161.map.gz / 形式: CCP4 / 大きさ: 120.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_45161.map.gz / 形式: CCP4 / 大きさ: 120.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CryoCARE denoised tomogram of a cultured neuronal synapse. | ||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 13.6 Å | ||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
+追加マップ: CryoCARE denoised tomogram of a cultured neuronal synapse.
+追加マップ: CryoCARE denoised tomogram of a cultured neuronal synapse.
+追加マップ: CryoCARE denoised tomogram of a cultured neuronal synapse.
+追加マップ: CryoCARE denoised tomogram of a cultured neuronal synapse.
+追加マップ: CryoCARE denoised tomogram of a cultured neuronal synapse.
+追加マップ: CryoCARE denoised tomogram of a cultured neuronal synapse.
+追加マップ: CryoCARE denoised tomogram of a cultured neuronal synapse.
+追加マップ: CryoCARE denoised tomogram of a cultured neuronal synapse.
+追加マップ: CryoCARE denoised tomogram of a cultured neuronal synapse.
+追加マップ: CryoCARE denoised tomogram of a cultured neuronal synapse.
+追加マップ: CryoCARE denoised tomogram of a cultured neuronal synapse.
+追加マップ: CryoCARE denoised tomogram of a cultured neuronal synapse.
- 試料の構成要素
試料の構成要素
-全体 : Tomograms of Cultured Neuronal Synapses
| 全体 | 名称: Tomograms of Cultured Neuronal Synapses |
|---|---|
| 要素 |
|
-超分子 #1: Tomograms of Cultured Neuronal Synapses
| 超分子 | 名称: Tomograms of Cultured Neuronal Synapses / タイプ: cell / ID: 1 / 親要素: 0 詳細: Tomograms of cryo-FIB lamellae of from cultured neurons. |
|---|---|
| 由来(天然) | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 電子線トモグラフィー法 |
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| グリッド | モデル: Quantifoil R2/2 / 材質: GOLD / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 45 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 298 K / 装置: LEICA EM GP |
| 詳細 | tomograms from FIB-milled lamellae of cultured mouse hippocampal neurons, targeting neuronal synapses. |
| 切片作成 | 集束イオンビーム - 装置: OTHER / 集束イオンビーム - イオン: OTHER / 集束イオンビーム - 電圧: 30 / 集束イオンビーム - 電流: 0.03 / 集束イオンビーム - 時間: 600 / 集束イオンビーム - 温度: 83 K / 集束イオンビーム - Initial thickness: 1000 / 集束イオンビーム - 最終 厚さ: 175 集束イオンビーム - 詳細: The milling sequence was the following (all at 30 keV): rough milling: 0.3 nA, 3 um pattern separation in Y; medium milling: 0.1 nA, 1 um pattern separation; fine ...集束イオンビーム - 詳細: The milling sequence was the following (all at 30 keV): rough milling: 0.3 nA, 3 um pattern separation in Y; medium milling: 0.1 nA, 1 um pattern separation; fine milling: 50 pA, 500 nm pattern separation; polishing: 30 pA, 175 nm pattern separation.. The value given for _em_focused_ion_beam.instrument is TFS Aquilos 2. This is not in a list of allowed values {'OTHER', 'DB235'} so OTHER is written into the XML file. |
| 位置合わせマーカー | 直径: 2.5 nm |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 3.2 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 9.0 µm / 最小 デフォーカス(公称値): 3.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | アルゴリズム: BACK PROJECTION / ソフトウェア - 名称:  IMOD IMOD詳細: IMOD was used for tilt-series alignment and weighted back-projection, followed by denoising using CryoCARE. 使用した粒子像数: 41 |
|---|
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)