+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of Mnx H340A complex from Bacillus sp. PL-12 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Complex / multicopper oxidase / manganese biomineralization / METAL TRANSPORT | |||||||||
| 機能・相同性 | Multicopper oxidase, copper-binding site / Multicopper oxidases signature 2. / Multicopper oxidase, N-terminal / Multicopper oxidase / Cupredoxin / copper ion binding / MnxE / MnxF / MnxG 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  | |||||||||
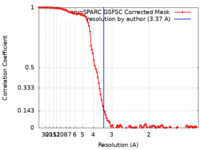
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.37 Å | |||||||||
 データ登録者 データ登録者 | Novikova IV / Evans JE | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: J Am Chem Soc / 年: 2024 ジャーナル: J Am Chem Soc / 年: 2024タイトル: Cryo-EM Structure of the Mnx Protein Complex Reveals a Tunnel Framework for the Mechanism of Manganese Biomineralization. 著者: Irina V Novikova / Alexandra V Soldatova / Trevor H Moser / Stephanie M Thibert / Christine A Romano / Mowei Zhou / Bradley M Tebo / James E Evans / Thomas G Spiro /  要旨: The global manganese cycle relies on microbes to oxidize soluble Mn(II) to insoluble Mn(IV) oxides. Some microbes require peroxide or superoxide as oxidants, but others can use O directly, via ...The global manganese cycle relies on microbes to oxidize soluble Mn(II) to insoluble Mn(IV) oxides. Some microbes require peroxide or superoxide as oxidants, but others can use O directly, via multicopper oxidase (MCO) enzymes. One of these, MnxG from strain PL-12, was isolated in tight association with small accessory proteins, MnxE and MnxF. The protein complex, called Mnx, has eluded crystallization efforts, but we now report the 3D structure of a point mutant using cryo-EM single particle analysis, cross-linking mass spectrometry, and AlphaFold Multimer prediction. The β-sheet-rich complex features MnxG enzyme, capped by a heterohexameric ring of alternating MnxE and MnxF subunits, and a tunnel that runs through MnxG and its MnxEF cap. The tunnel dimensions and charges can accommodate the mechanistically inferred binuclear manganese intermediates. Comparison with the Fe(II)-oxidizing MCO, ceruloplasmin, identifies likely coordinating groups for the Mn(II) substrate, at the entrance to the tunnel. Thus, the 3D structure provides a rationale for the established manganese oxidase mechanism, and a platform for further experiments to elucidate mechanistic details of manganese biomineralization. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_45001.map.gz emd_45001.map.gz | 68.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-45001-v30.xml emd-45001-v30.xml emd-45001.xml emd-45001.xml | 17.1 KB 17.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_45001_fsc.xml emd_45001_fsc.xml | 10.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_45001.png emd_45001.png | 113.4 KB | ||
| マスクデータ |  emd_45001_msk_1.map emd_45001_msk_1.map | 137.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-45001.cif.gz emd-45001.cif.gz | 6.4 KB | ||
| その他 |  emd_45001_half_map_1.map.gz emd_45001_half_map_1.map.gz emd_45001_half_map_2.map.gz emd_45001_half_map_2.map.gz | 127.4 MB 127.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-45001 http://ftp.pdbj.org/pub/emdb/structures/EMD-45001 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-45001 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-45001 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_45001_validation.pdf.gz emd_45001_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_45001_full_validation.pdf.gz emd_45001_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_45001_validation.xml.gz emd_45001_validation.xml.gz | 19.7 KB | 表示 | |
| CIF形式データ |  emd_45001_validation.cif.gz emd_45001_validation.cif.gz | 25.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-45001 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-45001 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-45001 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-45001 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  9bxaMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_45001.map.gz / 形式: CCP4 / 大きさ: 137.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_45001.map.gz / 形式: CCP4 / 大きさ: 137.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.68 Å | ||||||||||||||||||||||||||||||||||||
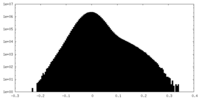
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_45001_msk_1.map emd_45001_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
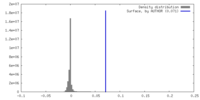
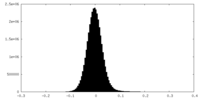
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_45001_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
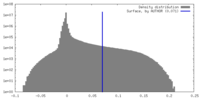
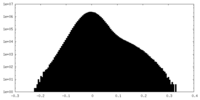
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_45001_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
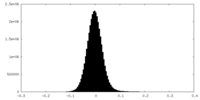
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Mnx H340A complex
| 全体 | 名称: Mnx H340A complex |
|---|---|
| 要素 |
|
-超分子 #1: Mnx H340A complex
| 超分子 | 名称: Mnx H340A complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 210 KDa |
-分子 #1: MnxG
| 分子 | 名称: MnxG / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 138.340672 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MLRKFHVVGI STRIVVNTFG DHNPNGRIYV LKENESKLKD LVRKNPYKPI DLVQPLAIRA NEGDIVEILF ENQLSFSAGM HFQEADYSV LSSDGADAGY NPDTTVEPGG EILYRLNVNQ EGICFFTDLG NVSSTEQGSS VQGLFGALLV QKRGSSWTDP V TGGPINSG ...文字列: MLRKFHVVGI STRIVVNTFG DHNPNGRIYV LKENESKLKD LVRKNPYKPI DLVQPLAIRA NEGDIVEILF ENQLSFSAGM HFQEADYSV LSSDGADAGY NPDTTVEPGG EILYRLNVNQ EGICFFTDLG NVSSTEQGSS VQGLFGALLV QKRGSSWTDP V TGGPINSG VYADIHHPFL PSFREYAWFF NDEMEIRDLT GERPLNPMTN QEAESFHGVN LRYEPMTNRK RLMEAGVVCP DC DSEEVHH DSWVFGDPAT PILRGYVGDP AVIRLIHGGV KETHVFHYHV HQWLGDSSNI NAEILDAQSI SPQTHYSIQP LYG LGSLHG AIGDSIIHCH LYPAFGIGMW GMNRVFDTLQ DGSQCYPNGV RIKALMPLPD RPEPPKPTPE KPGFPNFIPG KVGY KAPRP PLGIVGGREM TELERNAAIE NPRPGAVFVD PCLDQDPVVV EFNVSAIEMP VVYNKQGWHD PKARFYVMDE DLDDI LSGK KEPEPLVFHV PAGTCIRMNY TNRMPHILDG DAFQLVTRTY ENGFHIHFVK FDVLACDGGN VGWNYDSAVL PGQTIR YEW YAETELKAFF FHDHLFANSH QQHGVFGAGV IQPRFSKFLD SRTGDEVDHG TQISVEHPLI PDYRDQTLFV HDFALLF DK NGRPIQPPEY PGSEDDPGVF GVNFKCEPLK FRLGEDCDPA YSFSSYVHGD PVTPILRAYE GDPIRIRLLQ GAHEESHS F NIHGLRWKEE RPDLGSSMKA QQHIGISESF TFETEIPASG DYLWAFEDEE DVWLGTWGLI RAYKGRMEDL IVLTDREAL PEGSAETPKP TGKPPEKANP LASLPPGAYQ GSPVKKFEVV AFQTPIQYNS YGDHDPYGII FALKEDVEDI LTGKKNPVPL ILRANVGDL VEVTLTSELK KELFPFQDGI HPYPPVKEQS FYPPSLRISL HTSLLNYDVK TSSGDTVGYN PDQTVGPGET I TYRWFVDG QFGMCSMWDM ADLRNHRSFG TFGAFVAESR FTTYLDPYSL EKAITGENVI LRHPLLPATR EFVLILHDGV RL EDKDGKV IIDPMDGVVP DTEELEEVDT YDYGSRGFNY RSERLINRYK EHPVMHELFS SEVFGDPATP LFEAYPGEPV VMR ITTPAE RRRAHTFHLH GHYWKFDSKD LDSRIQSFLG HMVTGHTDDL RLIGGAGGVF NFPGDYLYRS GNIRWDIELG MWGI FRVHK DSKENLPRLE EVEGGWDNEE KA UniProtKB: MnxG |
-分子 #2: MnxE
| 分子 | 名称: MnxE / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 12.185235 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MHDSPLKSLS AASNVASVND PLFDFFNKHM GKQILIITES SQLNILGQTF RPIFCGKVAE VEPGHLTLSP VTIKILNAPF HKFPIPLSI PFEKIAHFTT DVDCSMRIPL V UniProtKB: MnxE |
-分子 #3: MnxF
| 分子 | 名称: MnxF / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 12.026734 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MEALFPMSTD YSKMTDVNEI HDSAILEHFR NGIGHKTLVI SPSYPYMFVG IIKELIGDTV MIDVETTHFA QLENREWYIH IHNIEVFYI ERPGAPKIPK LEDY UniProtKB: MnxF |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 4.5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.8 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % |
| 詳細 | contains 80mM CHAPSO |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 130000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー



 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)