+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | SARS CoV2 spike in complex with NTD-directed antibody Fab DH1052 | |||||||||
 マップデータ マップデータ | CoV2 spike in complex with antibody Fab DH1052 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | SARS coronavirus spike antibody /  VIRAL PROTEIN (ウイルスタンパク質) VIRAL PROTEIN (ウイルスタンパク質) | |||||||||
| 生物種 |   Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) / Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) /   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
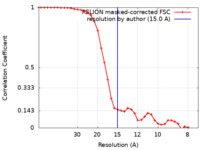
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  ネガティブ染色法 / 解像度: 15.0 Å ネガティブ染色法 / 解像度: 15.0 Å | |||||||||
 データ登録者 データ登録者 | Edwards RJ / Mansouri K | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: PLoS Pathog. ジャーナル: PLoS Pathog.タイトル: Non-neutralizing SARS-CoV-2 N-terminal domain antibodies protects mice against severe disease using Fc effector functions 著者: Pierre CN / Adams LE / Anasti K / Goodman D / Stanfield-Oakley S / Li D / Rountree W / Wang Y / Edwards RJ / Alam SM / Ferrari G / Tomaras GD / Haynes BF / Baric RS / Saunders KO | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_44644.map.gz emd_44644.map.gz | 3.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-44644-v30.xml emd-44644-v30.xml emd-44644.xml emd-44644.xml | 18.2 KB 18.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_44644_fsc.xml emd_44644_fsc.xml | 3.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_44644.png emd_44644.png | 51.6 KB | ||
| Filedesc metadata |  emd-44644.cif.gz emd-44644.cif.gz | 5.1 KB | ||
| その他 |  emd_44644_half_map_1.map.gz emd_44644_half_map_1.map.gz emd_44644_half_map_2.map.gz emd_44644_half_map_2.map.gz | 2.4 MB 2.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-44644 http://ftp.pdbj.org/pub/emdb/structures/EMD-44644 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-44644 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-44644 | HTTPS FTP |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_44644.map.gz / 形式: CCP4 / 大きさ: 3.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_44644.map.gz / 形式: CCP4 / 大きさ: 3.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CoV2 spike in complex with antibody Fab DH1052 | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3.74 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half map 1
| ファイル | emd_44644_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 | ||||||||||||
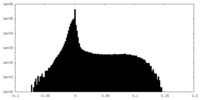
| 投影像・断面図 |
| ||||||||||||
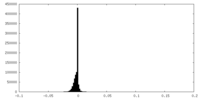
| 密度ヒストグラム |
-ハーフマップ: Half map 2
| ファイル | emd_44644_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 | ||||||||||||
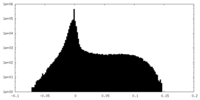
| 投影像・断面図 |
| ||||||||||||
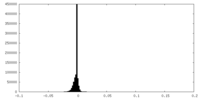
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex of SARS CoV2 spike with NTD-directed antibody Fab DH1052
| 全体 | 名称: Complex of SARS CoV2 spike with NTD-directed antibody Fab DH1052 |
|---|---|
| 要素 |
|
-超分子 #1: Complex of SARS CoV2 spike with NTD-directed antibody Fab DH1052
| 超分子 | 名称: Complex of SARS CoV2 spike with NTD-directed antibody Fab DH1052 タイプ: complex / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:   Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) |
| 分子量 | 理論値: 372 KDa |
-超分子 #2: DH1052 antibody Fab
| 超分子 | 名称: DH1052 antibody Fab / タイプ: complex / ID: 2 / 親要素: 1 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #3: SARS CoV2 spike
| 超分子 | 名称: SARS CoV2 spike / タイプ: complex / ID: 3 / 親要素: 1 |
|---|---|
| 由来(天然) | 生物種:   Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) |
-実験情報
-構造解析
| 手法 |  ネガティブ染色法 ネガティブ染色法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.1 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
| |||||||||
| 染色 | タイプ: NEGATIVE / 材質: Uranyl formate 詳細: A 5 microliter drop of complex was incubated on the grid for 10 to 15 s, blotted and then stained with 2 percent uranyl formate for 1 minute, then blotted and air dried. | |||||||||
| グリッド | モデル: Homemade / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 3 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 20 sec. | |||||||||
| 詳細 | Spike protein and Fab protein at 1 mg per ml were stored at minus 80 degrees C until use. Spike was thawed in an aluminum block at 37 degrees C for 5 minutes, Fab was thawed in an aluminum block at room temperature for 5 minutes. To for the complex, 2 microliters of spike was mixed with Fab at a 1 to 9 ratio spike to Fab, and incubated at 37 degrees C for 1 hour. The complex was then crosslinked by diluting to a final spike concentration of 0.1 mg per ml into room temperature buffer containing 150 mM NaCl, 20 mM HEPES pH 7.4, 5 percent glycerol, and 7.5 mM glutaraldehyde. After 5 minutes cross-linking, excess glutaraldehyde was quenched by adding sufficient 1 M Tris pH 7.4 stock to give a final concentration of 75 mM Tris and incubated for 5 minutes. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI/PHILIPS EM420 |
|---|---|
| 電子線 | 加速電圧: 120 kV / 電子線源: LAB6 |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.0 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 82000 Bright-field microscopy / Cs: 2.0 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 82000 |
| 試料ステージ | 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC |
| 撮影 | フィルム・検出器のモデル: OTHER / デジタル化 - サイズ - 横: 2048 pixel / デジタル化 - サイズ - 縦: 2048 pixel / 撮影したグリッド数: 1 / 実像数: 200 / 平均露光時間: 0.5 sec. / 平均電子線量: 32.0 e/Å2 |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: cross correlation |
 ムービー
ムービー コントローラー
コントローラー




 Z
Z Y
Y X
X