+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM Structure of E.coli produced recombinant N-acetyltransferase 10 (NAT10) in complex with cytidine-amide-CoA bisubstrate probe and ADP. | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | RNA binding protein / ac4C modification / RNA acetyltransferase | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報tRNA cytidine N4-acetyltransferase activity / rRNA acetylation involved in maturation of SSU-rRNA / 18S rRNA cytidine N-acetyltransferase activity / tRNA acetylation / 90S preribosome / 転移酵素; アシル基を移すもの; アミノアシル基以外のアシル基を移すもの / tRNA binding / nucleolus / ATP binding 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Thermochaetoides thermophila DSM 1495 (菌類) Thermochaetoides thermophila DSM 1495 (菌類) | ||||||||||||
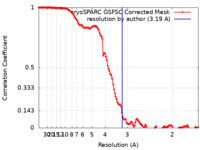
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.19 Å | ||||||||||||
 データ登録者 データ登録者 | Zhou M / Marmorstein R | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: bioRxiv / 年: 2024 ジャーナル: bioRxiv / 年: 2024タイトル: Molecular Basis for RNA Cytidine Acetylation by NAT10. 著者: Mingyang Zhou / Supuni Thalalla Gamage / Khoa A Tran / David Bartee / Xuepeng Wei / Boyu Yin / Shelley Berger / Jordan L Meier / Ronen Marmorstein 要旨: Human NAT10 acetylates the N4 position of cytidine in RNA, predominantly on rRNA and tRNA, to facilitate ribosome biogenesis and protein translation. NAT10 has been proposed as a therapeutic target ...Human NAT10 acetylates the N4 position of cytidine in RNA, predominantly on rRNA and tRNA, to facilitate ribosome biogenesis and protein translation. NAT10 has been proposed as a therapeutic target in cancers as well as aging-associated pathologies such as Hutchinson-Gilford Progeria Syndrome (HGPS). The ∼120 kDa NAT10 protein uses its acetyl-CoA-dependent acetyltransferase, ATP-dependent helicase, and RNA binding domains in concert to mediate RNA-specific N4-cytidine acetylation. While the biochemical activity of NAT10 is well known, the molecular basis for catalysis of eukaryotic RNA acetylation remains relatively undefined. To provide molecular insights into the RNA-specific acetylation by NAT10, we determined the single particle cryo-EM structures of NAT10 ( NAT10) bound to a bisubstrate cytidine-CoA probe with and without ADP. The structures reveal that NAT10 forms a symmetrical heart-shaped dimer with conserved functional domains surrounding the acetyltransferase active sites harboring the cytidine-CoA probe. Structure-based mutagenesis with analysis of mutants supports the catalytic role of two conserved active site residues (His548 and Tyr549 in NAT10), and two basic patches, both proximal and distal to the active site for RNA-specific acetylation. Yeast complementation analyses and senescence assays in human cells also implicates NAT10 catalytic activity in yeast thermoadaptation and cellular senescence. Comparison of the NAT10 structure to protein lysine and N-terminal acetyltransferase enzymes reveals an unusually open active site suggesting that these enzymes have been evolutionarily tailored for RNA recognition and cytidine-specific acetylation. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_44038.map.gz emd_44038.map.gz | 167.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-44038-v30.xml emd-44038-v30.xml emd-44038.xml emd-44038.xml | 17.5 KB 17.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_44038_fsc.xml emd_44038_fsc.xml | 12 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_44038.png emd_44038.png | 99.7 KB | ||
| Filedesc metadata |  emd-44038.cif.gz emd-44038.cif.gz | 6.6 KB | ||
| その他 |  emd_44038_half_map_1.map.gz emd_44038_half_map_1.map.gz emd_44038_half_map_2.map.gz emd_44038_half_map_2.map.gz | 165.3 MB 165.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-44038 http://ftp.pdbj.org/pub/emdb/structures/EMD-44038 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-44038 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-44038 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_44038_validation.pdf.gz emd_44038_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_44038_full_validation.pdf.gz emd_44038_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_44038_validation.xml.gz emd_44038_validation.xml.gz | 20.3 KB | 表示 | |
| CIF形式データ |  emd_44038_validation.cif.gz emd_44038_validation.cif.gz | 26.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44038 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44038 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44038 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44038 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_44038.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_44038.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.83 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : E.coli produced recombinant NAT10 in complex with ADP and chemica...
| 全体 | 名称: E.coli produced recombinant NAT10 in complex with ADP and chemically synthetic cytidine-amide-CoA bisubstrate probe |
|---|---|
| 要素 |
|
-超分子 #1: E.coli produced recombinant NAT10 in complex with ADP and chemica...
| 超分子 | 名称: E.coli produced recombinant NAT10 in complex with ADP and chemically synthetic cytidine-amide-CoA bisubstrate probe タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Thermochaetoides thermophila DSM 1495 (菌類) Thermochaetoides thermophila DSM 1495 (菌類) |
| 分子量 | 理論値: 247 kDa/nm |
-分子 #1: RNA cytidine acetyltransferase
| 分子 | 名称: RNA cytidine acetyltransferase / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO EC番号: 転移酵素; アシル基を移すもの; アミノアシル基以外のアシル基を移すもの |
|---|---|
| 由来(天然) | 生物種:  Thermochaetoides thermophila DSM 1495 (菌類) Thermochaetoides thermophila DSM 1495 (菌類) |
| 分子量 | 理論値: 121.475914 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: HHHHHHENLY FQGMTVQKTV DSRIPTLIRN GLQTKKRSFF VVVGDHAKEA IVHLYYIMSS MDVRQNKSVL WAYKKELLGF TSHRKKREA KIKKEIKRGI REPNQADPFE LFISLNDIRY CYYKETDKIL GNTYGMCILQ DFEAITPNIL ARTIETVEGG G LVVLLLKG ...文字列: HHHHHHENLY FQGMTVQKTV DSRIPTLIRN GLQTKKRSFF VVVGDHAKEA IVHLYYIMSS MDVRQNKSVL WAYKKELLGF TSHRKKREA KIKKEIKRGI REPNQADPFE LFISLNDIRY CYYKETDKIL GNTYGMCILQ DFEAITPNIL ARTIETVEGG G LVVLLLKG MTSLKQLYTM TMDVHARYRT EAHDDVIARF NERFLLSLGS CESCLVIDDE LNVLPISGGK GVKPLPPPDE DE ELSPAAK ELKKIKDELE DTQPIGSLIK LARTVDQAKA LLTFVDAIAE KTLRNTVTLT AARGRGKSAA MGVAIAAAVA YGY SNIFIT SPSPENLKTL FEFVFKGFDA LDYKDHADYT IIQSTNPEFN KAIVRVNIHR NHRQTIQYIR PQDAHVLGQA ELVV IDEAA AIPLPLVKKL MGPYLVFMAS TISGYEGTGR SLSLKLIKQL REQSRAGANP NGGNAVEVDR STLKATKETT SVGGR SLKE ITLSEPIRYA QGDNVEKWLN TLLCLDATLP RSKISTTGCP DPSQCELLHV NRDTLFSFHP VSEKFLQQMV ALYVAS HYK NSPNDLQLMS DAPAHELFVL TGPIQEGRLP EPLCVIQVSL EGKISKQSIL KSLSRGQQPA GDLIPWLVSQ QFQDDEF AS LSGARIVRIA TNPDYMSMGY GSKALQLLVD YYEGKFADLS EDAAAEVPRS IPRVTDAELS KGSLFDDIKV RDMHELPP L FSKLSERRPE KLDYVGVSYG LTQQLHKFWK RAQFVPVYLR QTANDLTGEH TCVMIRPLQD GNDPSWLGAF AADFHKRFL SLLSYKFREF PSILALTIEE SANAGAMLDP SNAPTELTKA ELDQLFTPFD HKRLESYANG LLDYHVVLDL MPTIAQLYFT GRLREAVKL SGLQQAILLA LGLQRKDIDT LATELNLPGS QVLAIFMKIM RKVTQHFGAL VSGAIAAELP DPNKTVGVSK E NAMGIHDD EVVGLKFEAL EQRLEDELDE GGDEALRELR KKQRELIDSL PLDQYEIDGD DDAWKEAEKR VASAAKSGKK VD GTLVSVP SAKAAKRKAE EMAALRDELE KMEKGKERGS KKAKKEKRR UniProtKB: RNA cytidine acetyltransferase |
-分子 #2: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 2 / コピー数: 2 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-分子 #3: [[(2~{R},3~{S},4~{S},5~{R})-5-(6-aminopurin-9-yl)-4-oxidanyl-3-ph...
| 分子 | 名称: [[(2~{R},3~{S},4~{S},5~{R})-5-(6-aminopurin-9-yl)-4-oxidanyl-3-phosphonooxy-oxolan-2-yl]methoxy-oxidanyl-phosphoryl] [(3~{R})-4-[[3-[2-[2-[[1-[(2~{R},3~{S},4~{R},5~{R})-5-(hydroxymethyl)-3,4- ...名称: [[(2~{R},3~{S},4~{S},5~{R})-5-(6-aminopurin-9-yl)-4-oxidanyl-3-phosphonooxy-oxolan-2-yl]methoxy-oxidanyl-phosphoryl] [(3~{R})-4-[[3-[2-[2-[[1-[(2~{R},3~{S},4~{R},5~{R})-5-(hydroxymethyl)-3,4-bis(oxidanyl)oxolan-2-yl]-2-oxidanylidene-pyrimidin-4-yl]amino]-2-oxidanylidene-ethyl]sulfanylethylamino]-3-oxidanylidene-propyl]amino]-2,2-dimethyl-3-oxidanyl-4-oxidanylidene-butyl] hydrogen phosphate タイプ: ligand / ID: 3 / コピー数: 2 / 式: A1AH4 |
|---|---|
| 分子量 | 理論値: 1.050772 KDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.8 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 280.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 45.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: Other / Chain - Initial model type: experimental model 詳細: The unpublished cryo-EM Structure of E.coli produced recombinant N-acetyltransferase 10 (NAT10) in complex with cytidine-acetate-CoA bisubstrate probe |
|---|---|
| ソフトウェア | 名称:  Coot (ver. 0.9.8.7) Coot (ver. 0.9.8.7) |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
| 得られたモデル |  PDB-9b0e: |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)