+ Open data
Open data
- Basic information
Basic information
| Entry |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | SthK R120A R124A in the presence of PIP2 and cAMP | |||||||||
 Map data Map data | unsharpened map after 3D-refinement | |||||||||
 Sample Sample |
| |||||||||
 Keywords Keywords | cyclic-nucleotide binding / potassium channel / lipid modulation / TRANSPORT PROTEIN | |||||||||
| Function / homology |  Function and homology information Function and homology informationintracellularly cyclic nucleotide-activated monoatomic cation channel activity / protein-containing complex binding / membrane Similarity search - Function | |||||||||
| Biological species |  Spirochaeta thermophila (bacteria) Spirochaeta thermophila (bacteria) | |||||||||
| Method | single particle reconstruction / cryo EM / Resolution: 2.5 Å | |||||||||
 Authors Authors | Schmidpeter PAM / Thon O / Nimigean CM | |||||||||
| Funding support |  United States, 1 items United States, 1 items
| |||||||||
 Citation Citation |  Journal: Nat Commun / Year: 2024 Journal: Nat Commun / Year: 2024Title: PIP2 inhibits pore opening of the cyclic nucleotide-gated channel SthK. Authors: Oliver Thon / Zhihan Wang / Philipp A M Schmidpeter / Crina M Nimigean /   Abstract: The signaling lipid phosphatidylinositol-4,5-bisphosphate (PIP2) regulates many ion channels. It inhibits eukaryotic cyclic nucleotide-gated (CNG) channels while activating their relatives, the ...The signaling lipid phosphatidylinositol-4,5-bisphosphate (PIP2) regulates many ion channels. It inhibits eukaryotic cyclic nucleotide-gated (CNG) channels while activating their relatives, the hyperpolarization-activated and cyclic nucleotide-modulated (HCN) channels. The prokaryotic SthK channel from Spirochaeta thermophila shares features with CNG and HCN channels and is an established model for this channel family. Here, we show SthK activity is inhibited by PIP2. A cryo-EM structure of SthK in nanodiscs reveals a PIP2-fitting density coordinated by arginine and lysine residues from the S4 helix and the C-linker, located between voltage-sensing and pore domains of adjacent subunits. Mutation of two arginine residues weakens PIP2 inhibition with the double mutant displaying insensitivity to PIP2. We propose that PIP2 inhibits SthK by gluing S4 and S6 together, stabilizing a resting channel conformation. The PIP2 binding site is partially conserved in CNG channels suggesting the possibility of a similar inhibition mechanism in the eukaryotic homologs. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Supplemental images |
|---|
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_43522.map.gz emd_43522.map.gz | 101.6 MB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-43522-v30.xml emd-43522-v30.xml emd-43522.xml emd-43522.xml | 18 KB 18 KB | Display Display |  EMDB header EMDB header |
| FSC (resolution estimation) |  emd_43522_fsc.xml emd_43522_fsc.xml | 11.6 KB | Display |  FSC data file FSC data file |
| Images |  emd_43522.png emd_43522.png | 62.9 KB | ||
| Filedesc metadata |  emd-43522.cif.gz emd-43522.cif.gz | 6.5 KB | ||
| Others |  emd_43522_half_map_1.map.gz emd_43522_half_map_1.map.gz emd_43522_half_map_2.map.gz emd_43522_half_map_2.map.gz | 103.4 MB 103.4 MB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-43522 http://ftp.pdbj.org/pub/emdb/structures/EMD-43522 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-43522 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-43522 | HTTPS FTP |
-Related structure data
| Related structure data |  8vtbM M: atomic model generated by this map C: citing same article ( |
|---|---|
| Similar structure data | Similarity search - Function & homology  F&H Search F&H Search |
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| Related items in Molecule of the Month |
- Map
Map
| File |  Download / File: emd_43522.map.gz / Format: CCP4 / Size: 137.1 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_43522.map.gz / Format: CCP4 / Size: 137.1 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | unsharpened map after 3D-refinement | ||||||||||||||||||||||||||||||||||||
| Projections & slices | Image control
Images are generated by Spider. | ||||||||||||||||||||||||||||||||||||
| Voxel size | X=Y=Z: 0.9 Å | ||||||||||||||||||||||||||||||||||||
| Density |
| ||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
|
-Supplemental data
-Half map: half-map 1
| File | emd_43522_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | half-map 1 | ||||||||||||
| Projections & Slices |
| ||||||||||||
| Density Histograms |
-Half map: half-map 2
| File | emd_43522_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | half-map 2 | ||||||||||||
| Projections & Slices |
| ||||||||||||
| Density Histograms |
- Sample components
Sample components
-Entire : tetrameric SthK R120A R124A protein
| Entire | Name: tetrameric SthK R120A R124A protein |
|---|---|
| Components |
|
-Supramolecule #1: tetrameric SthK R120A R124A protein
| Supramolecule | Name: tetrameric SthK R120A R124A protein / type: complex / ID: 1 / Parent: 0 / Macromolecule list: #1 Details: SthK R120A R124A in complex with cAMP reconstituted into MSP1E3 nanodiscs containing PIP2 |
|---|---|
| Source (natural) | Organism:  Spirochaeta thermophila (bacteria) Spirochaeta thermophila (bacteria) |
| Molecular weight | Theoretical: 200 KDa |
-Macromolecule #1: Transcriptional regulator, Crp/Fnr family
| Macromolecule | Name: Transcriptional regulator, Crp/Fnr family / type: protein_or_peptide / ID: 1 / Number of copies: 4 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  Spirochaeta thermophila (bacteria) Spirochaeta thermophila (bacteria) |
| Molecular weight | Theoretical: 50.588918 KDa |
| Recombinant expression | Organism:  |
| Sequence | String: MAKDIGINSD PNSSSVMKSS GVSNPTYTLV WKVWILAVTL YYAIRIPLTL VFPSLFSPLL PLDILASLAL IADIPLDLAF ESRRTSGRK PTLLAPSRLP DLLAALPLDL LVFALHLPSP LSLLSLVRLL KLISVQASAT AILSYRINPA LLRLLSLVGF I LLAAHGIA ...String: MAKDIGINSD PNSSSVMKSS GVSNPTYTLV WKVWILAVTL YYAIRIPLTL VFPSLFSPLL PLDILASLAL IADIPLDLAF ESRRTSGRK PTLLAPSRLP DLLAALPLDL LVFALHLPSP LSLLSLVRLL KLISVQASAT AILSYRINPA LLRLLSLVGF I LLAAHGIA CGWMSLQPPS ENPAGTRYLS AFYWTITTLT TIGYGDITPS TPTQTVYTIV IELLGAAMYG LVIGNIASLV SK LDAAKLL HRERVERVTA FLSYKRISPE LQRRIIEYFD YLWETRRGYE EREVLKELPH PLRLAVAMEI HGDVIEKVPL FKG AGEEFI RDIILHLEPV IYGPGEYIIR AGEMGSDVYF INRGSVEVLS ADEKTRYAIL SEGQFFGEMA LILRAPRTAT VRAR AFCDL YRLDKETFDR ILSRYPEIAA QIQELAVRRK ELESSGLVPR GSVKHHHH UniProtKB: Transcriptional regulator, Crp/Fnr family |
-Macromolecule #2: ADENOSINE-3',5'-CYCLIC-MONOPHOSPHATE
| Macromolecule | Name: ADENOSINE-3',5'-CYCLIC-MONOPHOSPHATE / type: ligand / ID: 2 / Number of copies: 4 / Formula: CMP |
|---|---|
| Molecular weight | Theoretical: 329.206 Da |
| Chemical component information | 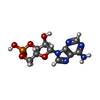 ChemComp-CMP: |
-Macromolecule #3: 1,2-DIOLEOYL-SN-GLYCERO-3-PHOSPHOCHOLINE
| Macromolecule | Name: 1,2-DIOLEOYL-SN-GLYCERO-3-PHOSPHOCHOLINE / type: ligand / ID: 3 / Number of copies: 24 / Formula: PCW |
|---|---|
| Molecular weight | Theoretical: 787.121 Da |
| Chemical component information |  ChemComp-PCW: |
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | single particle reconstruction |
| Aggregation state | particle |
- Sample preparation
Sample preparation
| Concentration | 7.5 mg/mL | ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| Buffer | pH: 7.4 Component:
| ||||||||||
| Grid | Model: UltrAuFoil R1.2/1.3 / Material: GOLD / Mesh: 300 / Support film - Material: GOLD / Support film - topology: HOLEY / Pretreatment - Type: GLOW DISCHARGE / Pretreatment - Time: 80 sec. | ||||||||||
| Vitrification | Cryogen name: ETHANE / Chamber humidity: 100 % / Chamber temperature: 298 K / Instrument: FEI VITROBOT MARK IV / Details: 30 s incubation 2 s blotting blot force -4. | ||||||||||
| Details | nanodisc reconstitution was carried out in the presence of DOPC, POPG, and PIP2 |
- Electron microscopy
Electron microscopy
| Microscope | TFS KRIOS |
|---|---|
| Image recording | Film or detector model: GATAN K3 BIOQUANTUM (6k x 4k) / Average electron dose: 56.64 e/Å2 |
| Electron beam | Acceleration voltage: 300 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | Illumination mode: FLOOD BEAM / Imaging mode: BRIGHT FIELD / Nominal defocus max: 2.5 µm / Nominal defocus min: 0.5 µm / Nominal magnification: 105000 |
| Sample stage | Cooling holder cryogen: NITROGEN |
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
 Movie
Movie Controller
Controller











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)






































