+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | DNA origami colloid for self-assembly of tubules: (10,0) monomer | |||||||||
 マップデータ マップデータ | Primary map of the (10,0) monomer. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | DNA origami / synthetic construct / self-assembly / DNA | |||||||||
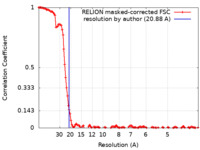
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 20.88 Å | |||||||||
 データ登録者 データ登録者 | Videbaek TE / Rogers WB | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2024 ジャーナル: Sci Adv / 年: 2024タイトル: Economical routes to size-specific assembly of self-closing structures. 著者: Thomas E Videbæk / Daichi Hayakawa / Gregory M Grason / Michael F Hagan / Seth Fraden / W Benjamin Rogers /  要旨: Programmable self-assembly has seen an explosion in the diversity of synthetic crystalline materials, but developing strategies that target "self-limiting" assemblies has remained a challenge. Among ...Programmable self-assembly has seen an explosion in the diversity of synthetic crystalline materials, but developing strategies that target "self-limiting" assemblies has remained a challenge. Among these, self-closing structures, in which the local curvature defines the finite global size, are prone to polymorphism due to thermal bending fluctuations, a problem that worsens with increasing target size. Here, we show that assembly complexity can be used to eliminate this source of polymorphism in the assembly of tubules. Using many distinct components, we prune the local density of off-target geometries, increasing the selectivity of the tubule width and helicity to nearly 100%. We further show that by reducing the design constraints to target either the pitch or the width alone, fewer components are needed to reach complete selectivity. Combining experiments with theory, we reveal an economical limit, which determines the minimum number of components required to create arbitrary assembly sizes with full selectivity. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_43227.map.gz emd_43227.map.gz | 162.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-43227-v30.xml emd-43227-v30.xml emd-43227.xml emd-43227.xml | 14.2 KB 14.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_43227_fsc.xml emd_43227_fsc.xml | 13 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_43227.png emd_43227.png | 62.7 KB | ||
| マスクデータ |  emd_43227_msk_1.map emd_43227_msk_1.map | 178 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-43227.cif.gz emd-43227.cif.gz | 4 KB | ||
| その他 |  emd_43227_half_map_1.map.gz emd_43227_half_map_1.map.gz emd_43227_half_map_2.map.gz emd_43227_half_map_2.map.gz | 140.7 MB 140.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-43227 http://ftp.pdbj.org/pub/emdb/structures/EMD-43227 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-43227 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-43227 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_43227_validation.pdf.gz emd_43227_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_43227_full_validation.pdf.gz emd_43227_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_43227_validation.xml.gz emd_43227_validation.xml.gz | 20.1 KB | 表示 | |
| CIF形式データ |  emd_43227_validation.cif.gz emd_43227_validation.cif.gz | 26.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43227 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43227 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43227 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43227 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_43227.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_43227.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Primary map of the (10,0) monomer. | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.023 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
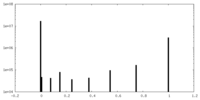
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_43227_msk_1.map emd_43227_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
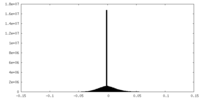
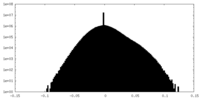
| 密度ヒストグラム |
-ハーフマップ: Half-map of the structure.
| ファイル | emd_43227_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map of the structure. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
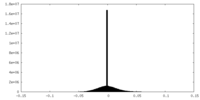
| 密度ヒストグラム |
-ハーフマップ: Half-map of the structure.
| ファイル | emd_43227_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map of the structure. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : DNA origami colloid for self-assembly of tubules: (10,0) monomer
| 全体 | 名称: DNA origami colloid for self-assembly of tubules: (10,0) monomer |
|---|---|
| 要素 |
|
-超分子 #1: DNA origami colloid for self-assembly of tubules: (10,0) monomer
| 超分子 | 名称: DNA origami colloid for self-assembly of tubules: (10,0) monomer タイプ: complex / ID: 1 / 親要素: 0 |
|---|---|
| 分子量 | 理論値: 5.2 MDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| グリッド | モデル: C-flat-1.2/1.3 / 材質: COPPER / メッシュ: 400 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 293 K / 装置: FEI VITROBOT MARK I |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS TUNDRA |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 検出モード: INTEGRATING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 実像数: 775 / 平均露光時間: 1.4 sec. / 平均電子線量: 69.8 e/Å2 |
| 電子線 | 加速電圧: 100 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD / Cs: 1.6 mm / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
 ムービー
ムービー コントローラー
コントローラー





 Z
Z Y
Y X
X