+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the Human Respirovirus 3 Fusion Protein Bound to Camelid Nanobodies 4C03 and 4C06 | |||||||||
 マップデータ マップデータ | Final refinement volume. DeepEMhancer sharpened. Used for building coordinates. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | viral fusion protein / camelid nanobodies / viral glycoprotein / membrane fusion / VIRAL PROTEIN / VIRAL PROTEIN-IMMUNE SYSTEM complex | |||||||||
| 機能・相同性 | Precursor fusion glycoprotein F0, Paramyxoviridae / Fusion glycoprotein F0 / fusion of virus membrane with host plasma membrane / viral envelope / symbiont entry into host cell / host cell plasma membrane / virion membrane / membrane / Fusion glycoprotein F0 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Human respirovirus 3 (ウイルス) / Human respirovirus 3 (ウイルス) /  | |||||||||
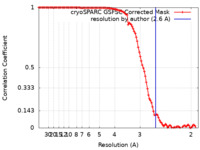
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.6 Å | |||||||||
 データ登録者 データ登録者 | Johnson NV / Ramamohan AR / McLellan JS | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Structural basis for potent neutralization of human respirovirus type 3 by protective single-domain camelid antibodies. 著者: Nicole V Johnson / Revina C van Scherpenzeel / Mark J G Bakkers / Ajit R Ramamohan / Daan van Overveld / Lam Le / Johannes P M Langedijk / Joost A Kolkman / Jason S McLellan /    要旨: Respirovirus 3 is a leading cause of severe acute respiratory infections in vulnerable human populations. Entry into host cells is facilitated by the attachment glycoprotein and the fusion ...Respirovirus 3 is a leading cause of severe acute respiratory infections in vulnerable human populations. Entry into host cells is facilitated by the attachment glycoprotein and the fusion glycoprotein (F). Because of its crucial role, F represents an attractive therapeutic target. Here, we identify 13 F-directed heavy-chain-only antibody fragments that neutralize recombinant respirovirus 3. High-resolution cryo-EM structures of antibody fragments bound to the prefusion conformation of F reveal three distinct, previously uncharacterized epitopes. All three antibody fragments bind quaternary epitopes on F, suggesting mechanisms for neutralization that may include stabilization of the prefusion conformation. Studies in cotton rats demonstrate the prophylactic efficacy of these antibody fragments in reducing viral load in the lungs and nasal passages. These data highlight the potential of heavy-chain-only antibody fragments as effective interventions against respirovirus 3 infection and identify neutralizing epitopes that can be targeted for therapeutic development. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_42983.map.gz emd_42983.map.gz | 105.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-42983-v30.xml emd-42983-v30.xml emd-42983.xml emd-42983.xml | 18.7 KB 18.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_42983_fsc.xml emd_42983_fsc.xml | 10.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_42983.png emd_42983.png | 101.1 KB | ||
| Filedesc metadata |  emd-42983.cif.gz emd-42983.cif.gz | 6.1 KB | ||
| その他 |  emd_42983_additional_1.map.gz emd_42983_additional_1.map.gz emd_42983_half_map_1.map.gz emd_42983_half_map_1.map.gz emd_42983_half_map_2.map.gz emd_42983_half_map_2.map.gz | 117.9 MB 115.8 MB 115.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-42983 http://ftp.pdbj.org/pub/emdb/structures/EMD-42983 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42983 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42983 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8v5kMC  8v62C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_42983.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_42983.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Final refinement volume. DeepEMhancer sharpened. Used for building coordinates. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.94 Å | ||||||||||||||||||||||||||||||||||||
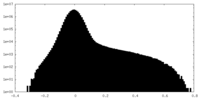
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Final refined volume. Sharpened map. Not DeepEMhancer sharpened.
| ファイル | emd_42983_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Final refined volume. Sharpened map. Not DeepEMhancer sharpened. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
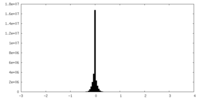
| 密度ヒストグラム |
-ハーフマップ: half map B
| ファイル | emd_42983_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
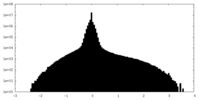
| 密度ヒストグラム |
-ハーフマップ: half map A
| ファイル | emd_42983_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
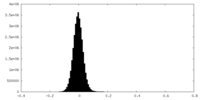
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Prefusion Human Respirovirus 3 Fusion Protein bound to Camelid na...
| 全体 | 名称: Prefusion Human Respirovirus 3 Fusion Protein bound to Camelid nanobodies 4C03 and 4C06 |
|---|---|
| 要素 |
|
-超分子 #1: Prefusion Human Respirovirus 3 Fusion Protein bound to Camelid na...
| 超分子 | 名称: Prefusion Human Respirovirus 3 Fusion Protein bound to Camelid nanobodies 4C03 and 4C06 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:  Human respirovirus 3 (ウイルス) Human respirovirus 3 (ウイルス) |
| 分子量 | 理論値: 277 KDa |
-分子 #1: Fusion glycoprotein F0
| 分子 | 名称: Fusion glycoprotein F0 / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Human respirovirus 3 (ウイルス) Human respirovirus 3 (ウイルス) |
| 分子量 | 理論値: 55.364965 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QIDITKLQHV GVLVNSPKGM KISQNFETRY LILSLIPKIE DSNSCGDQQI KQYKRLLDRL IIPLYDGLRL QKDVIVTNQE SNENTDPRT ERFFGGVIGT IALGVATSAQ ITAAVALVEA KQARSDIEKL KEAIRDTNKA VQSVQSSPGN LIVAIKSVQD Y VNKEIVPC ...文字列: QIDITKLQHV GVLVNSPKGM KISQNFETRY LILSLIPKIE DSNSCGDQQI KQYKRLLDRL IIPLYDGLRL QKDVIVTNQE SNENTDPRT ERFFGGVIGT IALGVATSAQ ITAAVALVEA KQARSDIEKL KEAIRDTNKA VQSVQSSPGN LIVAIKSVQD Y VNKEIVPC IARLGCEACG LLLGLALDQH YSELTNIFGD NIGSLQEKGI KLQGIASLYR TNITEIFTTS TVDKYDIYDL LF TESIKVR VIDVDLNDYS ITLQVRLPLL TRLLNTQIYK VDSISYNIQN REWYIPLPSH IMTKGAFLGG ADVKECIEAF SSY ICPSDP GFVLNHEMES CLSGNISQCP RTTVTSDIVP RYAFVNGGVV ANCITTTCTC NGIGNRINQP PDQGVKIITH KECN TIGIN GMLFNTNKEG TLAFYTPDDI TLNNSVALNP IDISIELNKA KSDLEESKEW IRRSNQKLDS IEDKIEEILS KIYHI ENEI ARIKKLIGEA EPEA UniProtKB: Fusion glycoprotein F0 |
-分子 #2: Camelid Nanobody 4C03
| 分子 | 名称: Camelid Nanobody 4C03 / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 13.726373 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: EVQLVESGGG LVRAGGSLRL SCAASLRDLH TRTFYMGWFR QDPGKEREFV AAIDWNTGAA SYPDSVKGRF TISKDNARNA VYLQMNNLK PEDTAVYYCA VGRPPLNRPT LAYYWGQGTQ VTVSS |
-分子 #3: Camelid Nanobody 4C06
| 分子 | 名称: Camelid Nanobody 4C06 / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 12.487816 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: EVQLVESGGG LVQPGGSLRL SCSASGSLST IKALGWYRRA PGRERELVAS ITSAGETNYA DSAKGRFTVS TDNAKNTVDL RMNSLKPED TAVYYCYAES FVLNIYWGQG TQVTVSSG |
-分子 #4: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 4 / コピー数: 3 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)