+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the unliganded hexameric prenyltransferase in bifunctional copalyl diphosphate synthase from Penicillium fellutanum with an open conformation | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | terpene / biosynthesis / enzyme / TRANSFERASE | |||||||||
| 生物種 |  Penicillium fellutanum ATCC 48694 (菌類) Penicillium fellutanum ATCC 48694 (菌類) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.81 Å | |||||||||
 データ登録者 データ登録者 | Gaynes MN / Christianson DW | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: J Struct Biol / 年: 2024 ジャーナル: J Struct Biol / 年: 2024タイトル: Structure of the prenyltransferase in bifunctional copalyl diphosphate synthase from Penicillium fellutanum reveals an open hexamer conformation. 著者: Matthew N Gaynes / Trey A Ronnebaum / Kollin Schultz / Jacque L Faylo / Ronen Marmorstein / David W Christianson /  要旨: Copalyl diphosphate synthase from Penicillium fellutanum (PfCPS) is an assembly-line terpene synthase that contains both prenyltransferase and class II cyclase activities. The prenyltransferase ...Copalyl diphosphate synthase from Penicillium fellutanum (PfCPS) is an assembly-line terpene synthase that contains both prenyltransferase and class II cyclase activities. The prenyltransferase catalyzes processive chain elongation reactions using dimethylallyl diphosphate and three equivalents of isopentenyl diphosphate to yield geranylgeranyl diphosphate, which is then utilized as a substrate by the class II cyclase domain to generate copalyl diphosphate. Here, we report the 2.81 Å-resolution cryo-EM structure of the hexameric prenyltransferase of full-length PfCPS, which is surrounded by randomly splayed-out class II cyclase domains connected by disordered polypeptide linkers. The hexamer can be described as a trimer of dimers; surprisingly, one of the three dimer-dimer interfaces is separated to yield an open hexamer conformation, thus breaking the D3 symmetry typically observed in crystal structures of other prenyltransferase hexamers such as wild-type human GGPP synthase (hGGPPS). Interestingly, however, an open hexamer conformation was previously observed in the crystal structure of D188Y hGGPPS, apparently facilitated by hexamer-hexamer packing in the crystal lattice. The cryo-EM structure of the PfCPS prenyltransferase hexamer is the first to reveal that an open conformation can be achieved even in the absence of a point mutation or interaction with another hexamer. Even though PfCPS octamers are not detected, we suggest that the open hexamer conformation represents an intermediate in the hexamer-octamer equilibrium for those prenyltransferases that do exhibit oligomeric heterogeneity. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_42853.map.gz emd_42853.map.gz | 643.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-42853-v30.xml emd-42853-v30.xml emd-42853.xml emd-42853.xml | 19 KB 19 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_42853_fsc.xml emd_42853_fsc.xml | 19.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_42853.png emd_42853.png | 53.1 KB | ||
| Filedesc metadata |  emd-42853.cif.gz emd-42853.cif.gz | 6.4 KB | ||
| その他 |  emd_42853_additional_1.map.gz emd_42853_additional_1.map.gz emd_42853_half_map_1.map.gz emd_42853_half_map_1.map.gz emd_42853_half_map_2.map.gz emd_42853_half_map_2.map.gz | 361.6 MB 675.3 MB 675.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-42853 http://ftp.pdbj.org/pub/emdb/structures/EMD-42853 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42853 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42853 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_42853_validation.pdf.gz emd_42853_validation.pdf.gz | 863.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_42853_full_validation.pdf.gz emd_42853_full_validation.pdf.gz | 863.2 KB | 表示 | |
| XML形式データ |  emd_42853_validation.xml.gz emd_42853_validation.xml.gz | 28.7 KB | 表示 | |
| CIF形式データ |  emd_42853_validation.cif.gz emd_42853_validation.cif.gz | 37.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42853 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42853 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42853 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42853 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_42853.map.gz / 形式: CCP4 / 大きさ: 729 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_42853.map.gz / 形式: CCP4 / 大きさ: 729 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.55 Å | ||||||||||||||||||||||||||||||||||||
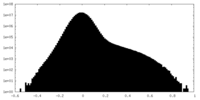
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Unsharpened C2 map of the PfCPS prenyltransferase generated...
| ファイル | emd_42853_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened C2 map of the PfCPS prenyltransferase generated in non-uniform refinement | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
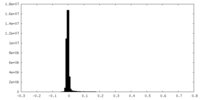
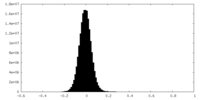
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_42853_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
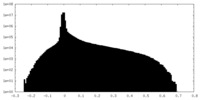
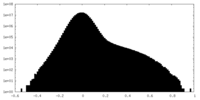
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_42853_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
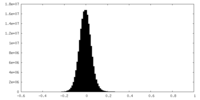
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Hexameric prenyltransferase from the bifunctional copalyl diphosp...
| 全体 | 名称: Hexameric prenyltransferase from the bifunctional copalyl diphosphate synthase of Penicillium fellutanum |
|---|---|
| 要素 |
|
-超分子 #1: Hexameric prenyltransferase from the bifunctional copalyl diphosp...
| 超分子 | 名称: Hexameric prenyltransferase from the bifunctional copalyl diphosphate synthase of Penicillium fellutanum タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Penicillium fellutanum ATCC 48694 (菌類) Penicillium fellutanum ATCC 48694 (菌類) |
| 分子量 | 理論値: 634.002 KDa |
-分子 #1: Copalyl diphosphate synthase
| 分子 | 名称: Copalyl diphosphate synthase / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Penicillium fellutanum ATCC 48694 (菌類) Penicillium fellutanum ATCC 48694 (菌類) |
| 分子量 | 理論値: 108.48407 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSSHHHHHH SSGENLYFQG HMASMESASL DQSAALLVKE LTEHIDDSNG LGFMSPAIYD TAWVSMIKKT DNDQTFWLFP KSFHYILEN QLENGGWVTY ASEIDGILNT SASLLSLKRH FDLPLQISTE SQTSMENRIR KATDALRVLL RTWDVDATLH V GFEILVPA ...文字列: MGSSHHHHHH SSGENLYFQG HMASMESASL DQSAALLVKE LTEHIDDSNG LGFMSPAIYD TAWVSMIKKT DNDQTFWLFP KSFHYILEN QLENGGWVTY ASEIDGILNT SASLLSLKRH FDLPLQISTE SQTSMENRIR KATDALRVLL RTWDVDATLH V GFEILVPA LLDYLQVEGL TFDFPGRDKL FQIRDQKLSR FKPEFIYAPF QTTALHSLEA FIGLIDFDRV QHHKVRGSFM AS PSSTAAV LMNATEWDID CEEYIRHVIE HASGKASGGV PSAFPSTIFE ITWTLSTLLK AGFNLSSNDS SNVQKACSYL LGV LTAEKG AIGFVPSVCA DADDTAKTIL VLSLLRENVL PDGMLKAFEV ENHFKTYPLE RDPSFSANCN VLLALLHLEN PSQY TLQIE KATRFLYTHF RESNLNVRDK WNLSPFYSWM LMAQAIARLD ELCKGSQLKN LHDHVESDLI PLLQEMTVSV MHQQN KDGS WGTKLSKEET AYAVLMLTYA VSFEALVGPR RQIRNAIEEG CLFLRSGKDA TSERLWVEKV TYESQMLSRA YTLAAL KNA LDVLKKDDMK IAFIGNSVNE SFTEIEVVNG KNVTEPSSNT YDLKEVKMDK AEFTPPDTPP RSKSTSVDPI EEAICAH EN RGAISDHTSR AIDLCRNPPL WGTDQEQTLL GPFEYLESIP GKNIRSQFIE AFNTWLQIPQ DHLQIVGKVI SMLHTASL L VDDIEDNSLL RRGQPVAHSI FGTAQTFNSG NYVYFLALQE VQKLNSPRAI SIFVDALTQL HRGQGMDVFW RDSLICPTE EEYLDMVANK TGALFCLAIE LLQIKSTVQL DFLPLVRLLG IIFQICDDYL NLKSTNYTQK KGLCEDITEG KFSFPIIHSI RTKPGNRQL INVLRQKSKE DDVKRFALAY MESTQSFDYT RDFVKILNGE ALRMIEDLEQ QGLHRNIEIR NILARMSLEQ |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK I |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 実像数: 12467 / 平均露光時間: 2.15 sec. / 平均電子線量: 43.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: AlphaFold / Chain - Initial model type: in silico model |
|---|---|
| 精密化 | プロトコル: AB INITIO MODEL |
| 得られたモデル |  PDB-8v0f: |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)