+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of GeoCas9 in complex with sgRNA and target DNA | |||||||||
 マップデータ マップデータ | Sharp map of deactivated WT-GeoCas9 with sgRNA and target DNA. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | RNA-guided DNA endonuclease / ternary complex / CRISPR / hydrolase / HYDROLASE-RNA-DNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報maintenance of CRISPR repeat elements / endonuclease activity / defense response to virus / 加水分解酵素; エステル加水分解酵素 / DNA binding / RNA binding / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |   Geobacillus stearothermophilus (バクテリア) / Geobacillus stearothermophilus (バクテリア) /  | |||||||||
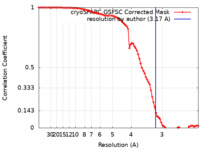
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.17 Å | |||||||||
 データ登録者 データ登録者 | Eggers AR / Soczek KM / Tuck OT / Doudna JA | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2024 ジャーナル: Cell / 年: 2024タイトル: Rapid DNA unwinding accelerates genome editing by engineered CRISPR-Cas9. 著者: Amy R Eggers / Kai Chen / Katarzyna M Soczek / Owen T Tuck / Erin E Doherty / Bryant Xu / Marena I Trinidad / Brittney W Thornton / Peter H Yoon / Jennifer A Doudna /  要旨: Thermostable clustered regularly interspaced short palindromic repeats (CRISPR) and CRISPR-associated (Cas9) enzymes could improve genome-editing efficiency and delivery due to extended protein ...Thermostable clustered regularly interspaced short palindromic repeats (CRISPR) and CRISPR-associated (Cas9) enzymes could improve genome-editing efficiency and delivery due to extended protein lifetimes. However, initial experimentation demonstrated Geobacillus stearothermophilus Cas9 (GeoCas9) to be virtually inactive when used in cultured human cells. Laboratory-evolved variants of GeoCas9 overcome this natural limitation by acquiring mutations in the wedge (WED) domain that produce >100-fold-higher genome-editing levels. Cryoelectron microscopy (cryo-EM) structures of the wild-type and improved GeoCas9 (iGeoCas9) enzymes reveal extended contacts between the WED domain of iGeoCas9 and DNA substrates. Biochemical analysis shows that iGeoCas9 accelerates DNA unwinding to capture substrates under the magnesium-restricted conditions typical of mammalian but not bacterial cells. These findings enabled rational engineering of other Cas9 orthologs to enhance genome-editing levels, pointing to a general strategy for editing enzyme improvement. Together, these results uncover a new role for the Cas9 WED domain in DNA unwinding and demonstrate how accelerated target unwinding dramatically improves Cas9-induced genome-editing activity. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_42837.map.gz emd_42837.map.gz | 60.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-42837-v30.xml emd-42837-v30.xml emd-42837.xml emd-42837.xml | 23.4 KB 23.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_42837_fsc.xml emd_42837_fsc.xml | 8.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_42837.png emd_42837.png | 98.5 KB | ||
| マスクデータ |  emd_42837_msk_1.map emd_42837_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-42837.cif.gz emd-42837.cif.gz | 7.4 KB | ||
| その他 |  emd_42837_additional_1.map.gz emd_42837_additional_1.map.gz emd_42837_half_map_1.map.gz emd_42837_half_map_1.map.gz emd_42837_half_map_2.map.gz emd_42837_half_map_2.map.gz | 49 MB 59.4 MB 59.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-42837 http://ftp.pdbj.org/pub/emdb/structures/EMD-42837 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42837 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42837 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_42837_validation.pdf.gz emd_42837_validation.pdf.gz | 883.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_42837_full_validation.pdf.gz emd_42837_full_validation.pdf.gz | 882.8 KB | 表示 | |
| XML形式データ |  emd_42837_validation.xml.gz emd_42837_validation.xml.gz | 16.4 KB | 表示 | |
| CIF形式データ |  emd_42837_validation.cif.gz emd_42837_validation.cif.gz | 21.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42837 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42837 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42837 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42837 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8uzaMC  8uzbC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_42837.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_42837.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharp map of deactivated WT-GeoCas9 with sgRNA and target DNA. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.93 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_42837_msk_1.map emd_42837_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Map of deactivated WT-GeoCas9 with sgRNA and target DNA.
| ファイル | emd_42837_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map of deactivated WT-GeoCas9 with sgRNA and target DNA. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map of deactivated WT-GeoCas9 with sgRNA and target DNA.
| ファイル | emd_42837_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map of deactivated WT-GeoCas9 with sgRNA and target DNA. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map of deactivated WT-GeoCas9 with sgRNA and target DNA.
| ファイル | emd_42837_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map of deactivated WT-GeoCas9 with sgRNA and target DNA. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Ternary complex of deactivated wild type GeoCas9 with sgRNA and t...
| 全体 | 名称: Ternary complex of deactivated wild type GeoCas9 with sgRNA and target DNA |
|---|---|
| 要素 |
|
-超分子 #1: Ternary complex of deactivated wild type GeoCas9 with sgRNA and t...
| 超分子 | 名称: Ternary complex of deactivated wild type GeoCas9 with sgRNA and target DNA タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Geobacillus stearothermophilus (バクテリア) Geobacillus stearothermophilus (バクテリア) |
| 分子量 | 理論値: 203 KDa |
-分子 #1: CRISPR-associated endonuclease Cas9
| 分子 | 名称: CRISPR-associated endonuclease Cas9 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Geobacillus stearothermophilus (バクテリア) Geobacillus stearothermophilus (バクテリア) |
| 分子量 | 理論値: 127.043883 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MRYKIGLAIG ITSVGWAVMN LDIPRIEDLG VRIFDRAENP QTGESLALPR RLARSARRRL RRRKHRLERI RRLVIREGIL TKEELDKLF EEKHEIDVWQ LRVEALDRKL NNDELARVLL HLAKRRGFKS NRKSERSNKE NSTMLKHIEE NRAILSSYRT V GEMIVKDP ...文字列: MRYKIGLAIG ITSVGWAVMN LDIPRIEDLG VRIFDRAENP QTGESLALPR RLARSARRRL RRRKHRLERI RRLVIREGIL TKEELDKLF EEKHEIDVWQ LRVEALDRKL NNDELARVLL HLAKRRGFKS NRKSERSNKE NSTMLKHIEE NRAILSSYRT V GEMIVKDP KFALHKRNKG ENYTNTIARD DLEREIRLIF SKQREFGNMS CTEEFENEYI TIWASQRPVA SKDDIEKKVG FC TFEPKEK RAPKATYTFQ SFIAWEHINK LRLISPSGAR GLTDEERRLL YEQAFQKNKI TYHDIRTLLH LPDDTYFKGI VYD RGESRK QNENIRFLEL DAYHQIRKAV DKVYGKGKSS SFLPIDFDTF GYALTLFKDD ADIHSYLRNE YEQNGKRMPN LANK VYDNE LIEELLNLSF TKFGHLSLKA LRSILPYMEQ GEVYSSACER AGYTFTGPKK KQKTMLLPNI PPIANPVVMR ALTQA RKVV NAIIKKYGSP VSIHIELARD LSQTFDERRK TKKEQDENRK KNETAIRQLM EYGLTLNPTG HDIVKFKLWS EQNGRC AYS LQPIEIERLL EPGYVEVDAV IPYSRSLDDS YTNKVLVLTR ENREKGNRIP AEYLGVGTER WQQFETFVLT NKQFSKK KR DRLLRLHYDE NEETEFKNRN LNDTRYISRF FANFIREHLK FAESDDKQKV YTVNGRVTAH LRSRWEFNKN REESDLHH A VDAVIVACTT PSDIAKVTAF YQRREQNKEL AKKTEPHFPQ PWPHFADELR ARLSKHPKES IKALNLGNYD DQKLESLQP VFVSRMPKRS VTGAAHQETL RRYVGIDERS GKIQTVVKTK LSEIKLDASG HFPMYGKESD PRTYEAIRQR LLEHNNDPKK AFQEPLYKP KKNGEPGPVI RTVKIIDTKN QVIPLNDGKT VAYNSNIVRV DVFEKDGKYY CVPVYTMDIM KGILPNKAIE P NKPYSEWK EMTEDYTFRF SLYPNDLIRI ELPREKTVKT AAGEEINVKD VFVYYKTIDS ANGGLELISH DHRFSLRGVG SR TLKRFEK YQVDVLGNIY KVRGEKRVGL ASSAHSKPGK TIRPLQSTRD UniProtKB: CRISPR-associated endonuclease Cas9 |
-分子 #2: sgRNA (107-MER)
| 分子 | 名称: sgRNA (107-MER) / タイプ: rna / ID: 2 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:   Geobacillus stearothermophilus (バクテリア) Geobacillus stearothermophilus (バクテリア) |
| 分子量 | 理論値: 44.488117 KDa |
| 配列 | 文字列: CACUGCAUUC UAGUUGUGGU UGUCAUAGUU CCCCUGAGAA AUCAGGGUUA CUAUGAUAAG GGCUUUCUGC CUAAGGCAGA CUGACCCGC GGCGUUGGGG AUCGCCUGUC GCCCGCUUUU GGCGGGCAUU CCCCAUCCUU |
-分子 #3: Target strand DNA
| 分子 | 名称: Target strand DNA / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 15.797221 KDa |
| 配列 | 文字列: (DT)(DA)(DC)(DA)(DT)(DT)(DG)(DA)(DT)(DG) (DA)(DG)(DT)(DT)(DT)(DG)(DG)(DA)(DC)(DA) (DA)(DA)(DC)(DC)(DA)(DC)(DA)(DA)(DC) (DT)(DA)(DG)(DA)(DA)(DT)(DG)(DC)(DA)(DG) (DT) (DG)(DA)(DA)(DA)(DA) ...文字列: (DT)(DA)(DC)(DA)(DT)(DT)(DG)(DA)(DT)(DG) (DA)(DG)(DT)(DT)(DT)(DG)(DG)(DA)(DC)(DA) (DA)(DA)(DC)(DC)(DA)(DC)(DA)(DA)(DC) (DT)(DA)(DG)(DA)(DA)(DT)(DG)(DC)(DA)(DG) (DT) (DG)(DA)(DA)(DA)(DA)(DA)(DA)(DA) (DT)(DG)(DC) |
-分子 #4: Non-target strand DNA
| 分子 | 名称: Non-target strand DNA / タイプ: dna / ID: 4 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 15.618021 KDa |
| 配列 | 文字列: (DG)(DC)(DA)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DC)(DA)(DC)(DT)(DG)(DC)(DA)(DT)(DT)(DC) (DT)(DA)(DG)(DT)(DT)(DG)(DT)(DG)(DG) (DT)(DT)(DT)(DG)(DT)(DC)(DC)(DA)(DA)(DA) (DC) (DT)(DC)(DA)(DT)(DC) ...文字列: (DG)(DC)(DA)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DC)(DA)(DC)(DT)(DG)(DC)(DA)(DT)(DT)(DC) (DT)(DA)(DG)(DT)(DT)(DG)(DT)(DG)(DG) (DT)(DT)(DT)(DG)(DT)(DC)(DC)(DA)(DA)(DA) (DC) (DT)(DC)(DA)(DT)(DC)(DA)(DA)(DT) (DG)(DT)(DA) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 281 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||
|---|---|---|---|---|---|---|---|
| 精密化 | 空間: REAL / プロトコル: OTHER | ||||||
| 得られたモデル |  PDB-8uza: |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)