+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4266 | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | ACP2 crosslinked to the KS of the loading/condensing region of the CTB1 PKS | ||||||||||||||||||
 マップデータ マップデータ | |||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
 キーワード キーワード | NR-PKS / PKS / iPKS / iterative PKS / non-reducing / SAT / starter acyl / condensing / loading / polyketide / fungal / crosslink / ACP / acyl carrier / transferase / biosynthetic protein | ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報secondary metabolite biosynthetic process / fatty acid synthase activity / phosphopantetheine binding / 3-oxoacyl-[acyl-carrier-protein] synthase activity / 転移酵素; アシル基を移すもの; アミノアシル基以外のアシル基を移すもの / fatty acid biosynthetic process 類似検索 - 分子機能 | ||||||||||||||||||
| 生物種 |  Cercospora nicotianae (菌類) Cercospora nicotianae (菌類) | ||||||||||||||||||
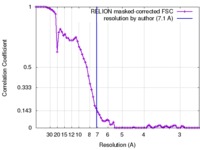
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 7.1 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Herbst DA / Huitt-Roehl CR / Jakob RP / Townsend CA / Maier T | ||||||||||||||||||
| 資金援助 |  スイス, スイス,  米国, 5件 米国, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Chem Biol / 年: 2018 ジャーナル: Nat Chem Biol / 年: 2018タイトル: The structural organization of substrate loading in iterative polyketide synthases. 著者: Dominik A Herbst / Callie R Huitt-Roehl / Roman P Jakob / Jacob M Kravetz / Philip A Storm / Jamie R Alley / Craig A Townsend / Timm Maier /   要旨: Polyketide synthases (PKSs) are microbial multienzymes for the biosynthesis of biologically potent secondary metabolites. Polyketide production is initiated by the loading of a starter unit onto an ...Polyketide synthases (PKSs) are microbial multienzymes for the biosynthesis of biologically potent secondary metabolites. Polyketide production is initiated by the loading of a starter unit onto an integral acyl carrier protein (ACP) and its subsequent transfer to the ketosynthase (KS). Initial substrate loading is achieved either by multidomain loading modules or by the integration of designated loading domains, such as starter unit acyltransferases (SAT), whose structural integration into PKS remains unresolved. A crystal structure of the loading/condensing region of the nonreducing PKS CTB1 demonstrates the ordered insertion of a pseudodimeric SAT into the condensing region, which is aided by the SAT-KS linker. Cryo-electron microscopy of the post-loading state trapped by mechanism-based crosslinking of ACP to KS reveals asymmetry across the CTB1 loading/-condensing region, in accord with preferential 1:2 binding stoichiometry. These results are critical for re-engineering the loading step in polyketide biosynthesis and support functional relevance of asymmetric conformations of PKSs. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4266.map.gz emd_4266.map.gz | 32.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4266-v30.xml emd-4266-v30.xml emd-4266.xml emd-4266.xml | 22.1 KB 22.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_4266_fsc.xml emd_4266_fsc.xml | 7.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4266.png emd_4266.png | 46 KB | ||
| Filedesc metadata |  emd-4266.cif.gz emd-4266.cif.gz | 7.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4266 http://ftp.pdbj.org/pub/emdb/structures/EMD-4266 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4266 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4266 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_4266_validation.pdf.gz emd_4266_validation.pdf.gz | 532.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_4266_full_validation.pdf.gz emd_4266_full_validation.pdf.gz | 531.6 KB | 表示 | |
| XML形式データ |  emd_4266_validation.xml.gz emd_4266_validation.xml.gz | 9.6 KB | 表示 | |
| CIF形式データ |  emd_4266_validation.cif.gz emd_4266_validation.cif.gz | 12.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4266 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4266 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4266 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4266 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4266.map.gz / 形式: CCP4 / 大きさ: 35.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4266.map.gz / 形式: CCP4 / 大きさ: 35.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.326 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : CTB1-SAT0-KS-MAT0=ACP2
| 全体 | 名称: CTB1-SAT0-KS-MAT0=ACP2 |
|---|---|
| 要素 |
|
-超分子 #1: CTB1-SAT0-KS-MAT0=ACP2
| 超分子 | 名称: CTB1-SAT0-KS-MAT0=ACP2 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Cercospora nicotianae (菌類) Cercospora nicotianae (菌類) |
-分子 #1: Polyketide synthase
| 分子 | 名称: Polyketide synthase / タイプ: protein_or_peptide / ID: 1 / 詳細: C553 in chain A is crosslinked to S1816 in chain C / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Cercospora nicotianae (菌類) Cercospora nicotianae (菌類) |
| 分子量 | 理論値: 140.255 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MEDGAQMRVV AFGDQTYDCS EAVSQLLRVR DDAIVVDFLE RAPAVLKAEL ARLSSEQQEE TPRFATLAEL VPRYRAGTLN PAVSQALTC IAQLGLFIRQ HSSGQEAYPT AHDSCITGVA TGALTAVAVG SASSVTALVP LALHTVAVAV RLGARAWEIG S CLADARRG ...文字列: MEDGAQMRVV AFGDQTYDCS EAVSQLLRVR DDAIVVDFLE RAPAVLKAEL ARLSSEQQEE TPRFATLAEL VPRYRAGTLN PAVSQALTC IAQLGLFIRQ HSSGQEAYPT AHDSCITGVA TGALTAVAVG SASSVTALVP LALHTVAVAV RLGARAWEIG S CLADARRG ANGRYASWTS AVGGISPQDL QDRISAYTAE QALASVSVPY LSAAVGPGQS SVSAAPVILD AFLSTLLRPL TT TRLPITA PYHAPHLFTA KDVQHVTDCL PPSEAWPTVR IPIISFSRDE AVSRGASFPA AMSEAVRDCL IRPIALDRMA VSI ANHARD LGKDSVLPSP IALSFSDKLG PQVNSHLPGA KAPTPELTSK SIPSAIGAEQ QPMAKSPIAI LAASGRFPQS SSMD QFWDV LINGVDTHEL VPPTRWNAAT HVSEDPKAKN VSGTGFGCWL HEAGEFDAAY FNMSPREAPQ VDPAQRLALL TATEA LEQA GVVPNRTSST QKNRVGVWYG ATSNDWMETN SAQNVDTYFI PGGNRAFIPG RVNYFHKFSG PSYTIDTACS SSLAAL HMA CNALWRGEVD TAIVGGTNVL TNPDMTAGLD AGHFLSRSGN CKTFDDEADG YCRGEAVVTL ILKRLPDAQA DKDPIQA SI LGIATNHSAE AASITRPHAG AQQDLFQQVL TETGLTANDI SVCEMHGTGT QAGDSGETTS VVETLAPLNR SGSAVRTT P LYIGAVKSNV GHAESAAGVS SLAKILLMLK HSKIPPHVGI KTKLNHRLPD LAARNTHIAR SEVPWPRPKN GKRRVLLNN FSAAGGNTCL VLEDAPEPED SQEVDPREHH IVALSAKTPD SMVNNLTNMI TWIDKHSGDS LATLPQLSYT TTARRVHHRH RAVATGTDL LQIRSSLQEQ LDRRVSGERS IPHPPNGPSF VLAFTGQGSA FAGMGVDLYK RFASFRSDIA RYDQICEGMS L PSIKAMFE DEKVFSTASP TLQQLTHVCF QMALYRLWKS LGVQAKAVVG HALGEYAALY AAGVLSQSDT LYLVGRRAQL ME KHLSQGT HAMLAVRAKE EAIVAAIDGP PGEAYEFSCR NGEQRNVLGG TVAQIQAAKA ALEAKKIRCQ YLDTPMAFHT GQV DPILPE LLQVAAACSI QDPQIPVISP AYGKVIRSAK DFQPEYFTHH CRSSVNMVDA LQSAVEEGLL DKNVIGLEIG PGPV VTQFV KEAVGTTMQT FASINKDKDT WQLMTQALAK FYLAGASVEW SRYHEDFPGA QKVLELPAYG WALKNYWLQY VNDWS LRKG DPAVVVAASA AALEHHHHHH UniProtKB: Non-reducing polyketide synthase CTB1 |
-分子 #2: Polyketide synthase
| 分子 | 名称: Polyketide synthase / タイプ: protein_or_peptide / ID: 2 / 詳細: S1816 is crosslinked to C553 in chain A / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Cercospora nicotianae (菌類) Cercospora nicotianae (菌類) |
| 分子量 | 理論値: 9.892837 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GSHMDPSPNE IGTVWRDALK ILSEESGLTD EELTDDTSFA DVGVDSLMSL VITSRLRDEL DIDFPDRALF EECQTIFDLR KRFSGSTE UniProtKB: Non-reducing polyketide synthase CTB1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.27 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil Lacey carbon grid / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: LACEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK III | ||||||||||||
| 詳細 | The proteins have been crosslinked via site specific crosslinking. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - エネルギー下限: 0 eV エネルギーフィルター - エネルギー上限: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 41.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 最大 デフォーカス(補正後): 4.5 µm / 最小 デフォーカス(補正後): 0.8 µm / 倍率(補正後): 27707 / 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)