+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
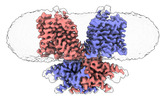
| タイトル | Cryo-EM structure of dolphin Prestin in low Cl buffer | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Solute Carrier / Electromotility / Voltage Sensitive / Mechanosensitive / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cochlear outer hair cell electromotile response / secondary active sulfate transmembrane transporter activity / sensory perception of sound / regulation of cell shape / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
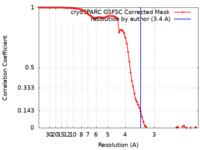
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.4 Å | |||||||||
 データ登録者 データ登録者 | Haller P / Bavi N / Perozo E | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2023 ジャーナル: Elife / 年: 2023タイトル: Folding of prestin's anion-binding site and the mechanism of outer hair cell electromotility. 著者: Xiaoxuan Lin / Patrick R Haller / Navid Bavi / Nabil Faruk / Eduardo Perozo / Tobin R Sosnick /  要旨: Prestin responds to transmembrane voltage fluctuations by changing its cross-sectional area, a process underlying the electromotility of outer hair cells and cochlear amplification. Prestin belongs ...Prestin responds to transmembrane voltage fluctuations by changing its cross-sectional area, a process underlying the electromotility of outer hair cells and cochlear amplification. Prestin belongs to the SLC26 family of anion transporters yet is the only member capable of displaying electromotility. Prestin's voltage-dependent conformational changes are driven by the putative displacement of residue R399 and a set of sparse charged residues within the transmembrane domain, following the binding of a Cl anion at a conserved binding site formed by the amino termini of the TM3 and TM10 helices. However, a major conundrum arises as to how an anion that binds in proximity to a positive charge (R399), can promote the voltage sensitivity of prestin. Using hydrogen-deuterium exchange mass spectrometry, we find that prestin displays an unstable anion-binding site, where folding of the amino termini of TM3 and TM10 is coupled to Cl binding. This event shortens the TM3-TM10 electrostatic gap, thereby connecting the two helices, resulting in reduced cross-sectional area. These folding events upon anion binding are absent in SLC26A9, a non-electromotile transporter closely related to prestin. Dynamics of prestin embedded in a lipid bilayer closely match that in detergent micelle, except for a destabilized lipid-facing helix TM6 that is critical to prestin's mechanical expansion. We observe helix fraying at prestin's anion-binding site but cooperative unfolding of multiple lipid-facing helices, features that may promote prestin's fast electromechanical rearrangements. These results highlight a novel role of the folding equilibrium of the anion-binding site, and help define prestin's unique voltage-sensing mechanism and electromotility. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_42112.map.gz emd_42112.map.gz | 59.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-42112-v30.xml emd-42112-v30.xml emd-42112.xml emd-42112.xml | 18.7 KB 18.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_42112_fsc.xml emd_42112_fsc.xml | 8.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_42112.png emd_42112.png | 102 KB | ||
| Filedesc metadata |  emd-42112.cif.gz emd-42112.cif.gz | 6.4 KB | ||
| その他 |  emd_42112_additional_1.map.gz emd_42112_additional_1.map.gz emd_42112_half_map_1.map.gz emd_42112_half_map_1.map.gz emd_42112_half_map_2.map.gz emd_42112_half_map_2.map.gz | 30.7 MB 59.1 MB 59.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-42112 http://ftp.pdbj.org/pub/emdb/structures/EMD-42112 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42112 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42112 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_42112_validation.pdf.gz emd_42112_validation.pdf.gz | 806.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_42112_full_validation.pdf.gz emd_42112_full_validation.pdf.gz | 806.1 KB | 表示 | |
| XML形式データ |  emd_42112_validation.xml.gz emd_42112_validation.xml.gz | 16.3 KB | 表示 | |
| CIF形式データ |  emd_42112_validation.cif.gz emd_42112_validation.cif.gz | 21 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42112 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42112 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42112 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42112 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8uc1MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_42112.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_42112.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.068 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: #1
| ファイル | emd_42112_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_42112_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_42112_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Dolphin Prestin solubilized in GDN in low Cl buffer
| 全体 | 名称: Dolphin Prestin solubilized in GDN in low Cl buffer |
|---|---|
| 要素 |
|
-超分子 #1: Dolphin Prestin solubilized in GDN in low Cl buffer
| 超分子 | 名称: Dolphin Prestin solubilized in GDN in low Cl buffer / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Prestin
| 分子 | 名称: Prestin / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 80.97375 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MDHVEETEIL AATQRYYVER PIFSHPVLQE RLHKKDKISE SIGDKLKQAF TCTPKKIRNI IYMFLPITKW LPAYRFKEYV LGDIVSGIS TGVLQLPQGL AFAMLAAVPP VFGLYSSFYP VIMYCFFGTS RHISIGPFAV ISLMIGGVAV RLVPDDIVIP G GVNATNST ...文字列: MDHVEETEIL AATQRYYVER PIFSHPVLQE RLHKKDKISE SIGDKLKQAF TCTPKKIRNI IYMFLPITKW LPAYRFKEYV LGDIVSGIS TGVLQLPQGL AFAMLAAVPP VFGLYSSFYP VIMYCFFGTS RHISIGPFAV ISLMIGGVAV RLVPDDIVIP G GVNATNST EARDALRVKV AMSVTLLTGI IQFCLGVCRF GFVAIYLTEP LVRGFTTAAA VHVFTSMLKY LFGVKTKRYS GI FSVVYST VAVLQNVKNL NVCSLGVGLM VFGLLLGGKE FNERFKEKLP APIPLEFFAV VMGTGISAGF SLHESYNVDV VGT LPLGLL PPANPDTSLF HLVYVDAIAI AIVGFSVTIS MAKTLANKHG YQVDGNQELI ALGLCNSTGS LFQTFAISCS LSRS LVQEG TGGKTQLAGC LASLMILLVI LATGFLFESL PQAVLSAIVI VNLKGMFMQF SDLPFFWRTS KIELTIWLTT FVSSL FLGL DYGLITAVII ALMTVIYRTQ SPSYIVLGQL PDTDVYIDID AYEEVKEVPG IKIFQINAPI YYANSDLYSS ALKRKT GVN PAFILGARRK AMKKYAKEVG NANMANATVV KVDAEVDAED GTKPEEEEDE IKYPPIVTKS TLPEELQRFM PPGDNVH TI ILDFTQVNFM DSVGVKTLAG IVKEYGDVGI YVYLAGCSAQ VVSDLTQNQF FENPALLDLL FHSIHDAVLG SQVREALA E QEATAAPPQE DSEPNATPEA UniProtKB: Prestin |
-分子 #2: CHLORIDE ION
| 分子 | 名称: CHLORIDE ION / タイプ: ligand / ID: 2 / コピー数: 2 / 式: CL |
|---|---|
| 分子量 | 理論値: 35.453 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
詳細: 190 mM HEPES, 95 mM Tris-base, 1mM NaCl, 3mM DTT, 1mM EDTA, 0.02 % GDN | |||||||||
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 雰囲気: OTHER | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK IV / 詳細: 3.5s Blot time, Blot force 1. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 平均電子線量: 1.2 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.1 µm 最小 デフォーカス(公称値): 0.7000000000000001 µm 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)