+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Human RADX tetramer bound to ssDNA | |||||||||
 マップデータ マップデータ | RADX tetramer bound to ssDNA dT25 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | oligomer OB-fold / RAD51 regulator / DNA BINDING PROTEIN / DNA BINDING PROTEIN-DNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of double-strand break repair via homologous recombination / regulation of DNA repair / replication fork / single-stranded DNA binding / nuclear speck / RNA binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.0 Å | |||||||||
 データ登録者 データ登録者 | Balakrishnan S / Chazin WJ | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2024 ジャーナル: Proc Natl Acad Sci U S A / 年: 2024タイトル: Structure of RADX and mechanism for regulation of RAD51 nucleofilaments. 著者: Swati Balakrishnan / Madison Adolph / Miaw-Sheue Tsai / Tae Akizuki / Kaitlyn Gallagher / David Cortez / Walter J Chazin /  要旨: Replication fork reversal is a fundamental process required for resolution of encounters with DNA damage. A key step in the stabilization and eventual resolution of reversed forks is formation of ...Replication fork reversal is a fundamental process required for resolution of encounters with DNA damage. A key step in the stabilization and eventual resolution of reversed forks is formation of RAD51 nucleoprotein filaments on exposed single strand DNA (ssDNA). To avoid genome instability, RAD51 filaments are tightly controlled by a variety of positive and negative regulators. RADX (RPA-related RAD51-antagonist on the X chromosome) is a recently discovered negative regulator that binds tightly to ssDNA, directly interacts with RAD51, and regulates replication fork reversal and stabilization in a context-dependent manner. Here, we present a structure-based investigation of RADX's mechanism of action. Mass photometry experiments showed that RADX forms multiple oligomeric states in a concentration-dependent manner, with a predominance of trimers in the presence of ssDNA. The structure of RADX, which has no structurally characterized orthologs, was determined ab initio by cryo-electron microscopy (cryo-EM) from maps in the 2 to 4 Å range. The structure reveals the molecular basis for RADX oligomerization and the coupled multi-valent binding of ssDNA binding. The interaction of RADX with RAD51 filaments was imaged by negative stain EM, which showed a RADX oligomer at the end of filaments. Based on these results, we propose a model in which RADX functions by capping and restricting the end of RAD51 filaments. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_41940.map.gz emd_41940.map.gz | 447 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-41940-v30.xml emd-41940-v30.xml emd-41940.xml emd-41940.xml | 18.2 KB 18.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_41940_fsc.xml emd_41940_fsc.xml | 16.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_41940.png emd_41940.png | 74.8 KB | ||
| Filedesc metadata |  emd-41940.cif.gz emd-41940.cif.gz | 6.5 KB | ||
| その他 |  emd_41940_half_map_1.map.gz emd_41940_half_map_1.map.gz emd_41940_half_map_2.map.gz emd_41940_half_map_2.map.gz | 474.4 MB 474.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-41940 http://ftp.pdbj.org/pub/emdb/structures/EMD-41940 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41940 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41940 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_41940_validation.pdf.gz emd_41940_validation.pdf.gz | 1.2 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_41940_full_validation.pdf.gz emd_41940_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  emd_41940_validation.xml.gz emd_41940_validation.xml.gz | 26.5 KB | 表示 | |
| CIF形式データ |  emd_41940_validation.cif.gz emd_41940_validation.cif.gz | 34.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41940 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41940 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41940 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41940 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8u61MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_41940.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_41940.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | RADX tetramer bound to ssDNA dT25 | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.818 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: half map A
| ファイル | emd_41940_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
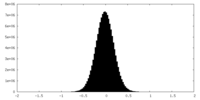
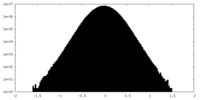
| 密度ヒストグラム |
-ハーフマップ: half map B
| ファイル | emd_41940_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
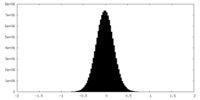
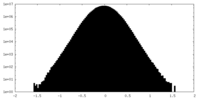
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : RADX tetramer bound to ssDNA dT25
| 全体 | 名称: RADX tetramer bound to ssDNA dT25 |
|---|---|
| 要素 |
|
-超分子 #1: RADX tetramer bound to ssDNA dT25
| 超分子 | 名称: RADX tetramer bound to ssDNA dT25 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 600 KDa |
-分子 #1: RPA-related protein RADX
| 分子 | 名称: RPA-related protein RADX / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 97.680094 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MSGESGQPEA GPSHAGLDWP NPERNRAGVP GGVIRRAGSQ GPRSWIQKVL EQIMDSPRQC VTPSEVVPVT VLAVQRYLLE DEPRDTVPK PPLYCYDVTI SDGVYQEKCY LDPSLNSLVY QNILKVGIQM RISRVSCLYN EKRIGQGILC IDNVHCGETS D SISLETPF ...文字列: MSGESGQPEA GPSHAGLDWP NPERNRAGVP GGVIRRAGSQ GPRSWIQKVL EQIMDSPRQC VTPSEVVPVT VLAVQRYLLE DEPRDTVPK PPLYCYDVTI SDGVYQEKCY LDPSLNSLVY QNILKVGIQM RISRVSCLYN EKRIGQGILC IDNVHCGETS D SISLETPF RNRAHQEKPE RPLRGGKSHY LALWNNEDPY GDIWLTDKQP EEHNFSDTKI ISLSHLEMTW TNRRNFPALL VR ILHKSKL RYYGKPDKKM IEPYQTFLEV ADSSGTVSVI MWNALCPEWY KSLRVGLVLL LQDYSVKKSY PFRIQPVPVD PQI KLISTM EICLNLRDPP TNIIIIPEKQ VKPEWRLPKL NHRFTTRSEL DDMPENCICD VIGLLVFVGR VQRSKKKENR EDFW SYRWI HIADGTSEQP FIVELFSTSQ PEIFENIYPM AYFVCTQLKV VRNDNQVPKL LYLTTTNESG VFITGHRGQP YTYDA KVKN FIQWIRTKSD SGEQKNMVIG GYYPYPPVPE TFSKYSSSIK VESLLTAISE VRKEIEDLQY REQKRIAIQG IITAIK YIP HSSATESASA SETLRNANRP STSQAARVEI QERNGKRHQD DEPVNSQYFQ TTSTNLSLSN KIRILQGPHA NPVAVPQ PG ASVQTKGIKP GMPSIFNRRA NINANLQGKA RKTISDRWES QLWREKKFGL IDHLHYSRVY PESIPRKFMF EHRKFLSD Q YNSQPAKYVP PEGRPPKLDD FKSARSLGHF EVTILGLNHE IAIDVAFLPM YCPEDIRTSQ IDTLLTSMNY SCAYPQDTT GNDRLPGPRA VAGDIIKAAT ELDRVHIVGI LDICNLGNNK VEVYLHKIYS PENTS UniProtKB: RPA-related protein RADX |
-分子 #2: dT25 DNA (25-MER)
| 分子 | 名称: dT25 DNA (25-MER) / タイプ: dna / ID: 2 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 7.559863 KDa |
| 配列 | 文字列: (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT)(DT)(DT)(DT)(DT) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.145 mg/mL | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 20 sec. | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV | ||||||||||||||||||
| 詳細 | Sample was cross-linked using BS3 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 平均電子線量: 48.38 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-8u61: |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)