+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of vaccine-elicited CD4 binding site antibody DH1285 bound to HIV-1 CH505TFchim.6R.SOSIP.664v4.1 Env Local Refinement | |||||||||
 マップデータ マップデータ | Locally refined map showing the vaccine elicited DH1285 antibody fab bound to one of pg120 protomer of CH505M5chimer.6R.SOSIP.664v4.1 Env | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | HIV-1 Vaccine induced neutralizing antibody / CD4 binding site antibody / gp120 / DH1285 / IgG / rhesus macaques / CH505M5chimer.6R.SOSIP.664v4.1 Env / VIRAL PROTEIN | |||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) / Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) /  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.25 Å | |||||||||
 データ登録者 データ登録者 | Thakur B / Stalls VD / Acharya P | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2024 ジャーナル: Cell / 年: 2024タイトル: Vaccine induction of CD4-mimicking HIV-1 broadly neutralizing antibody precursors in macaques. 著者: Kevin O Saunders / James Counts / Bhishem Thakur / Victoria Stalls / Robert Edwards / Kartik Manne / Xiaozhi Lu / Katayoun Mansouri / Yue Chen / Rob Parks / Maggie Barr / Laura Sutherland / ...著者: Kevin O Saunders / James Counts / Bhishem Thakur / Victoria Stalls / Robert Edwards / Kartik Manne / Xiaozhi Lu / Katayoun Mansouri / Yue Chen / Rob Parks / Maggie Barr / Laura Sutherland / Joena Bal / Nicholas Havill / Haiyan Chen / Emily Machiele / Nolan Jamieson / Bhavna Hora / Megan Kopp / Katarzyna Janowska / Kara Anasti / Chuancang Jiang / Elizabeth Van Itallie / Sravani Venkatayogi / Amanda Eaton / Rory Henderson / Christopher Barbosa / S Munir Alam / Sampa Santra / Drew Weissman / M Anthony Moody / Derek W Cain / Ying K Tam / Mark Lewis / Wilton B Williams / Kevin Wiehe / David C Montefiori / Priyamvada Acharya / Barton F Haynes /   要旨: The CD4-binding site (CD4bs) is a conserved epitope on HIV-1 envelope (Env) that can be targeted by protective broadly neutralizing antibodies (bnAbs). HIV-1 vaccines have not elicited CD4bs bnAbs ...The CD4-binding site (CD4bs) is a conserved epitope on HIV-1 envelope (Env) that can be targeted by protective broadly neutralizing antibodies (bnAbs). HIV-1 vaccines have not elicited CD4bs bnAbs for many reasons, including the occlusion of CD4bs by glycans, expansion of appropriate naive B cells with immunogens, and selection of functional antibody mutations. Here, we demonstrate that immunization of macaques with a CD4bs-targeting immunogen elicits neutralizing bnAb precursors with structural and genetic features of CD4-mimicking bnAbs. Structures of the CD4bs nAb bound to HIV-1 Env demonstrated binding angles and heavy-chain interactions characteristic of all known human CD4-mimicking bnAbs. Macaque nAb were derived from variable and joining gene segments orthologous to the genes of human VH1-46-class bnAb. This vaccine study initiated in primates the B cells from which CD4bs bnAbs can derive, accomplishing the key first step in the development of an effective HIV-1 vaccine. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_41810.map.gz emd_41810.map.gz | 117.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-41810-v30.xml emd-41810-v30.xml emd-41810.xml emd-41810.xml | 25 KB 25 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_41810.png emd_41810.png | 23.2 KB | ||
| Filedesc metadata |  emd-41810.cif.gz emd-41810.cif.gz | 7.8 KB | ||
| その他 |  emd_41810_half_map_1.map.gz emd_41810_half_map_1.map.gz emd_41810_half_map_2.map.gz emd_41810_half_map_2.map.gz | 115.5 MB 115.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-41810 http://ftp.pdbj.org/pub/emdb/structures/EMD-41810 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41810 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41810 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_41810_validation.pdf.gz emd_41810_validation.pdf.gz | 884.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_41810_full_validation.pdf.gz emd_41810_full_validation.pdf.gz | 883.8 KB | 表示 | |
| XML形式データ |  emd_41810_validation.xml.gz emd_41810_validation.xml.gz | 13.9 KB | 表示 | |
| CIF形式データ |  emd_41810_validation.cif.gz emd_41810_validation.cif.gz | 16.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41810 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41810 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41810 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41810 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_41810.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_41810.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Locally refined map showing the vaccine elicited DH1285 antibody fab bound to one of pg120 protomer of CH505M5chimer.6R.SOSIP.664v4.1 Env | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||||||||||||||||||
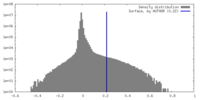
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_41810_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
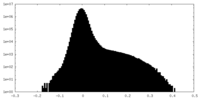
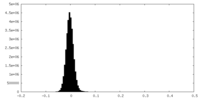
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_41810_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
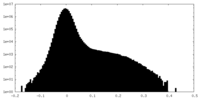
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : The complex of vaccine elicited CD4 binding site neutralizing ant...
| 全体 | 名称: The complex of vaccine elicited CD4 binding site neutralizing antibody DH1285 Fab with CH505M5chimer.6R.SOSIP.664v4.1 Env |
|---|---|
| 要素 |
|
-超分子 #1: The complex of vaccine elicited CD4 binding site neutralizing ant...
| 超分子 | 名称: The complex of vaccine elicited CD4 binding site neutralizing antibody DH1285 Fab with CH505M5chimer.6R.SOSIP.664v4.1 Env タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 詳細: One of the gp120 protomer bound to DH1285 locally refined to get precise information about the interacting residues. |
|---|---|
| 分子量 | 理論値: 308 KDa |
-超分子 #2: Envelope glycoprotein gp120
| 超分子 | 名称: Envelope glycoprotein gp120 / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
-超分子 #3: DH1285 Heavy Chain, DH1285 Light Chain
| 超分子 | 名称: DH1285 Heavy Chain, DH1285 Light Chain / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2-#3 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Envelope glycoprotein gp120
| 分子 | 名称: Envelope glycoprotein gp120 / タイプ: protein_or_peptide / ID: 1 / 詳細: CH505M5chimer.6R.SOSIP.664v4.1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 72.669078 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MPMGSLQPLA TLYLLGMLVA SVLAAENLWV TVYYGVPVWK EAKTTLFCAS DAKAYEKKVH NVWATHACVP TDPNPQEMVL KNVTENFNM WKNDMVDQMH EDVISLWDQS LKPCVKLTPL CVTLNCTNAT ASNSSIIEGM KNCSFNITTE LRDKREKKNA L FYKLDIVQ ...文字列: MPMGSLQPLA TLYLLGMLVA SVLAAENLWV TVYYGVPVWK EAKTTLFCAS DAKAYEKKVH NVWATHACVP TDPNPQEMVL KNVTENFNM WKNDMVDQMH EDVISLWDQS LKPCVKLTPL CVTLNCTNAT ASNSSIIEGM KNCSFNITTE LRDKREKKNA L FYKLDIVQ LDGNSSQYRL INCNTSVITQ ACPKVSFDPI PIHYCAPAGY AILKCNNKTF TGTGPCNNVS TVQCTHGIKP VV STQLLLN GSLAEGEIII RSENITNNVK TIIVHLNESV KIECTRPNNK TRTSIRIGPG QWFYATGQVI GDIREAYCNI NES KWNETL QRVSKKLKEY FPHKNITFQP SSGGDLEITT HSFNCGGEFF YCNTSSLFNR TYMANSTDMA NSTETNSTRT ITIH CRIKQ IINMWQEVGR AMYAPPIAGN ITCISNITGL LLTRDGGKNN TETFRPGGGN MKDNWRSELY KYKVVKIEPL GVAPT RCKR RVVGRRRRRR AVGIGAVFLG FLGAAGSTMG AASMTLTVQA RNLLSGIVQQ QSNLLRAPEA QQHLLKLTVW GIKQLQ ARV LAVERYLRDQ QLLGIWGCSG KLICCTNVPW NSSWSNRNLS EIWDNMTWLQ WDKEISNYTQ IIYGLLEESQ NQQEKNE QD LLALD |
-分子 #2: DH1285 Heavy Chain
| 分子 | 名称: DH1285 Heavy Chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 26.396316 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MGWSCIILFL VATATGVHAQ VHLEQSGAEV KEPGSSVRLS CEASGYTFTD YYIHWVRQSP RQGLEWMGWI NPYYGNTHYA EKFQGRVAM TRDRSTTTAY MDLSSLTSED TAVYYCARDE GGSGSYSYFD SWGQGVLVTV SSASTKGPSV FPLAPSSRST S ESTAALGC ...文字列: MGWSCIILFL VATATGVHAQ VHLEQSGAEV KEPGSSVRLS CEASGYTFTD YYIHWVRQSP RQGLEWMGWI NPYYGNTHYA EKFQGRVAM TRDRSTTTAY MDLSSLTSED TAVYYCARDE GGSGSYSYFD SWGQGVLVTV SSASTKGPSV FPLAPSSRST S ESTAALGC LVKDYFPEPV TVSWNSGSLT SGVHTFPAVL QSSGLYSLSS VVTVPSSSLG TQTYVCNVNH KPSNTKVDKR VE IKT |
-分子 #3: DH1285 Light Chain
| 分子 | 名称: DH1285 Light Chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 25.240955 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MGWSCIILFL VATATGVHAD IQMTQSPSSL SASVGDTVTI TCRASQDINN HLSWYQQKPG RAPKALIYSA SSLETGVPSR FSGSGSGTD YTLTISSLQP EDFATYYCQH YSTSPYTFGR GTKVDIKRAV AAPSVFIFPP SEDQVKSGTV SVVCLLNNFY P REASVKWK ...文字列: MGWSCIILFL VATATGVHAD IQMTQSPSSL SASVGDTVTI TCRASQDINN HLSWYQQKPG RAPKALIYSA SSLETGVPSR FSGSGSGTD YTLTISSLQP EDFATYYCQH YSTSPYTFGR GTKVDIKRAV AAPSVFIFPP SEDQVKSGTV SVVCLLNNFY P REASVKWK VDGVLKTGNS QESVTEQDSK DNTYSLSSTL TLSSTDYQSH NVYACEVTHQ GLSSPVTKSF NRGEC |
-分子 #4: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 4 / コピー数: 1 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 295 K / 装置: LEICA EM GP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 59.1 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL |
|---|---|
| 得られたモデル |  PDB-8u1d: |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)