[日本語] English
 万見
万見- EMDB-41761: Cryo-EM structure of Vibrio cholerae FtsE/FtsX/EnvC complex, shortened -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Vibrio cholerae FtsE/FtsX/EnvC complex, shortened | |||||||||
 マップデータ マップデータ | full map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | membrane protein / enzyme / TRANSPORT PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cell division site / transmembrane transporter activity / metalloendopeptidase activity / cell division / ATP hydrolysis activity / ATP binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.55 Å | |||||||||
 データ登録者 データ登録者 | Hao A / Lee S-Y | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2024 ジャーナル: Structure / 年: 2024タイトル: Structural insights into the FtsEX-EnvC complex regulation on septal peptidoglycan hydrolysis in Vibrio cholerae. 著者: Aili Hao / Yang Suo / Seok-Yong Lee /  要旨: During bacterial cell division, hydrolysis of septal peptidoglycan (sPG) is crucial for cell separation. This sPG hydrolysis is performed by the enzyme amidases whose activity is regulated by the ...During bacterial cell division, hydrolysis of septal peptidoglycan (sPG) is crucial for cell separation. This sPG hydrolysis is performed by the enzyme amidases whose activity is regulated by the integral membrane protein complex FtsEX-EnvC. FtsEX is an ATP-binding cassette transporter, and EnvC is a long coiled-coil protein that interacts with and activates the amidases. The molecular mechanism by which the FtsEX-EnvC complex activates amidases remains largely unclear. We present the cryo-electron microscopy structure of the FtsEX-EnvC complex from the pathogenic bacteria V. cholerae (FtsEX-EnvC). FtsEX-EnvC in the presence of ADP adopts a distinct conformation where EnvC is "horizontally extended" rather than "vertically extended". Subsequent structural studies suggest that EnvC can swing between these conformations in space in a nucleotide-dependent manner. Our structural analysis and functional studies suggest that FtsEX-EnvC employs spatial control of EnvC for amidase activation, providing mechanistic insights into the FtsEX-EnvC regulation on septal peptidoglycan hydrolysis. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_41761.map.gz emd_41761.map.gz | 179.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-41761-v30.xml emd-41761-v30.xml emd-41761.xml emd-41761.xml | 21 KB 21 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_41761.png emd_41761.png | 80.1 KB | ||
| マスクデータ |  emd_41761_msk_1.map emd_41761_msk_1.map | 347.6 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-41761.cif.gz emd-41761.cif.gz | 6.9 KB | ||
| その他 |  emd_41761_half_map_1.map.gz emd_41761_half_map_1.map.gz emd_41761_half_map_2.map.gz emd_41761_half_map_2.map.gz | 323 MB 323.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-41761 http://ftp.pdbj.org/pub/emdb/structures/EMD-41761 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41761 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41761 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_41761_validation.pdf.gz emd_41761_validation.pdf.gz | 854.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_41761_full_validation.pdf.gz emd_41761_full_validation.pdf.gz | 853.9 KB | 表示 | |
| XML形式データ |  emd_41761_validation.xml.gz emd_41761_validation.xml.gz | 17 KB | 表示 | |
| CIF形式データ |  emd_41761_validation.cif.gz emd_41761_validation.cif.gz | 20.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41761 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41761 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41761 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41761 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8tzkMC  8tzjC  8tzlC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_41761.map.gz / 形式: CCP4 / 大きさ: 347.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_41761.map.gz / 形式: CCP4 / 大きさ: 347.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | full map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_41761_msk_1.map emd_41761_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
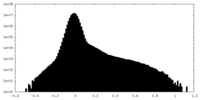
| 密度ヒストグラム |
-ハーフマップ: half map 2
| ファイル | emd_41761_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 1
| ファイル | emd_41761_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : FtsEX
| 全体 | 名称: FtsEX |
|---|---|
| 要素 |
|
-超分子 #1: FtsEX
| 超分子 | 名称: FtsEX / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 200 KDa |
-分子 #1: Cell division ATP-binding protein FtsE
| 分子 | 名称: Cell division ATP-binding protein FtsE / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 25.882668 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: PALSGGDGVI RFQQVSKAYR GGRQALQKVD FHLRRGEMAF LGGHSGAGKS TLLKLICAIE RPTDGKISFN GHDITRIPNK DIPFLRRNI GIVFQDHRLL MDRSIYDNVA LPMRIESISE NEIKRRVSAA LDKTGLLDKA RCLPSQLSGG EQQRVGIARA V VNRPTLLL ...文字列: PALSGGDGVI RFQQVSKAYR GGRQALQKVD FHLRRGEMAF LGGHSGAGKS TLLKLICAIE RPTDGKISFN GHDITRIPNK DIPFLRRNI GIVFQDHRLL MDRSIYDNVA LPMRIESISE NEIKRRVSAA LDKTGLLDKA RCLPSQLSGG EQQRVGIARA V VNRPTLLL ADEPTGNLDP ELSSRVLRLF EEFNRAGVTI LLATHDIHLV NSRPQYRHLE LNQGFLSEVA DYGR UniProtKB: Cell division ATP-binding protein FtsE |
-分子 #2: Cell division protein FtsX
| 分子 | 名称: Cell division protein FtsX / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 36.516773 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAVKPGNQKI SKTTKSTKSK PRDVKRAKTD SFLAIHFKQA KASFAALWRR PLGNILTLAV ISMALALPAS LYLLSKNIAS VAERVAEPS QLSVYLHIDT PEPRIIVLKD DLERRDEIAK VKYISPQQGL DDLSQYAGFE QAISLLDNAT LPAVLVVTPK V DSREQIQT ...文字列: MAVKPGNQKI SKTTKSTKSK PRDVKRAKTD SFLAIHFKQA KASFAALWRR PLGNILTLAV ISMALALPAS LYLLSKNIAS VAERVAEPS QLSVYLHIDT PEPRIIVLKD DLERRDEIAK VKYISPQQGL DDLSQYAGFE QAISLLDNAT LPAVLVVTPK V DSREQIQT LAKALQAEEG VTDVRMDEDW FARLDAIRHL ATIVVISLSS LMLMSVFLIV GNTLRFNVQA NKEEIQTMKL IG ATDAYIL RPYLYSGMWF GLLGAVAAWL LTALMTILLN GAVEALAQLY DSRFRLIGLG WDESLLLLML GVFLGCVAAK VSA KRHLKE IEPV UniProtKB: Cell division protein FtsX |
-分子 #3: Peptidase M23
| 分子 | 名称: Peptidase M23 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 43.410895 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTATDPHAIF SDFLGKTLTH RLLACLLFMV SPSLFAATQQ ELTGVKSEIS RQQQSLAEQQ KSLDQLQQAL KQQELGINSI ENQITKTKN DLENANRNIA QLNSNIQALE TQKQQQADKL ERLLQTYYLT KRSLTNGQFF HRSADEDRIS QYYQHLAKSR A QAIEALEK ...文字列: MTATDPHAIF SDFLGKTLTH RLLACLLFMV SPSLFAATQQ ELTGVKSEIS RQQQSLAEQQ KSLDQLQQAL KQQELGINSI ENQITKTKN DLENANRNIA QLNSNIQALE TQKQQQADKL ERLLQTYYLT KRSLTNGQFF HRSADEDRIS QYYQHLAKSR A QAIEALEK TQTELNSNQK QRQTEREQIE KLLAEQTQQR DKLAKTQSER KQTVKKIESS ISGNKTYLAE LQRNETRLKA EI AKAAKRN AVLMNGIASQ RGKLPWPLKG RVLHNFGERQ TGQIDWKGLV IDANYGQEVK AVYPGTIVFA EYLRGYGLVV LLD HGKGDM TLYGFNQTLL KKEGDKVTTG ETIALAGDTG GQSRPALYFE IRRNSRAENP SQWLQR UniProtKB: Peptidase M23 |
-分子 #4: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 4 / コピー数: 2 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #5: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 5 / コピー数: 2 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 8 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 277 K / 装置: LEICA EM GP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 1 / 平均露光時間: 2.4 sec. / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: AlphaFold / Chain - Initial model type: in silico model |
|---|---|
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
| 得られたモデル |  PDB-8tzk: |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)