+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4111 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Respiratory complex I of Yarrowia lipolytica with sulfur transferase subunit ST1 bound | |||||||||
 マップデータ マップデータ | Complex I from Yarrowia lipolyticsa with ST1 subunit bound | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Yarrowia lipolytica (酵母) Yarrowia lipolytica (酵母) | |||||||||
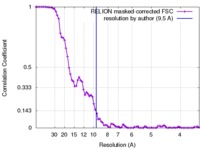
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 9.5 Å | |||||||||
 データ登録者 データ登録者 | D'Imprima E / Mills DJ / Parey K / Brandt U / Kuehlbrandt W / Zickermann V / Vonck J | |||||||||
 引用 引用 |  ジャーナル: Biochim Biophys Acta / 年: 2016 ジャーナル: Biochim Biophys Acta / 年: 2016タイトル: Cryo-EM structure of respiratory complex I reveals a link to mitochondrial sulfur metabolism. 著者: Edoardo D'Imprima / Deryck J Mills / Kristian Parey / Ulrich Brandt / Werner Kühlbrandt / Volker Zickermann / Janet Vonck /  要旨: Mitochondrial complex I is a 1MDa membrane protein complex with a central role in aerobic energy metabolism. The bioenergetic core functions are executed by 14 central subunits that are conserved ...Mitochondrial complex I is a 1MDa membrane protein complex with a central role in aerobic energy metabolism. The bioenergetic core functions are executed by 14 central subunits that are conserved from bacteria to man. Despite recent progress in structure determination, our understanding of the function of the ~30 accessory subunits associated with the mitochondrial complex is still limited. We have investigated the structure of complex I from the aerobic yeast Yarrowia lipolytica by cryo-electron microscopy. Our density map at 7.9Å resolution closely matches the 3.6-3.9Å X-ray structure of the Yarrowia lipolytica complex. However, the cryo-EM map indicated an additional subunit on the side of the matrix arm above the membrane surface, pointing away from the membrane arm. The density, which is not present in any previously described complex I structure and occurs in about 20 % of the particles, was identified as the accessory sulfur transferase subunit ST1. The Yarrowia lipolytica complex I preparation is active in generating HS from the cysteine derivative 3-mercaptopyruvate, catalyzed by ST1. We thus provide evidence for a link between respiratory complex I and mitochondrial sulfur metabolism. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4111.map.gz emd_4111.map.gz | 39.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4111-v30.xml emd-4111-v30.xml emd-4111.xml emd-4111.xml | 13.2 KB 13.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_4111_fsc.xml emd_4111_fsc.xml | 7.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4111.png emd_4111.png | 57.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4111 http://ftp.pdbj.org/pub/emdb/structures/EMD-4111 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4111 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4111 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_4111_validation.pdf.gz emd_4111_validation.pdf.gz | 266.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_4111_full_validation.pdf.gz emd_4111_full_validation.pdf.gz | 266.1 KB | 表示 | |
| XML形式データ |  emd_4111_validation.xml.gz emd_4111_validation.xml.gz | 10.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4111 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4111 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4111 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4111 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4111.map.gz / 形式: CCP4 / 大きさ: 42.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4111.map.gz / 形式: CCP4 / 大きさ: 42.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Complex I from Yarrowia lipolyticsa with ST1 subunit bound | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.77 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : NADH:ubiquinone oxidoreductase
| 全体 | 名称: NADH:ubiquinone oxidoreductase |
|---|---|
| 要素 |
|
-超分子 #1: NADH:ubiquinone oxidoreductase
| 超分子 | 名称: NADH:ubiquinone oxidoreductase / タイプ: complex / ID: 1 / 親要素: 0 詳細: complex I with ST1 subunit bound to the peripheral arm |
|---|---|
| 由来(天然) | 生物種:  Yarrowia lipolytica (酵母) Yarrowia lipolytica (酵母) |
| 分子量 | 理論値: 1 MDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR 詳細: Before glow discharge the grids were pretreated in chloroform for 1-2 hrs | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 70 % / チャンバー内温度: 283 K / 装置: FEI VITROBOT MARK IV / 詳細: The samples were blotted for 8-10 seconds. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 検出モード: INTEGRATING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / デジタル化 - サンプリング間隔: 14.0 µm / デジタル化 - 画像ごとのフレーム数: 2-24 / 実像数: 2250 / 平均露光時間: 1.5 sec. / 平均電子線量: 84.0 e/Å2 詳細: Images were collected in movi-mode at 17 frames per second |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 最大 デフォーカス(補正後): 4.5 µm / 最小 デフォーカス(補正後): 1.3 µm / 倍率(補正後): 79096 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 4.5 µm / 最小 デフォーカス(公称値): 1.3 µm / 倍率(公称値): 59000 |
| 試料ステージ | 試料ホルダーモデル: OTHER / ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: RIGID BODY FIT |
|---|
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)