+ Open data
Open data
- Basic information
Basic information
| Entry |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | Cryo-EM structure of RNA device 43, holo state | |||||||||
 Map data Map data | unsharpened map | |||||||||
 Sample Sample |
| |||||||||
 Keywords Keywords | RNA device / RNA | |||||||||
| Biological species | synthetic construct (others) | |||||||||
| Method | single particle reconstruction / cryo EM / Resolution: 2.98 Å | |||||||||
 Authors Authors | Stagno JR / Deme JC / Lee Y-T / Wang Y-X / Lea SM | |||||||||
| Funding support |  United States, 1 items United States, 1 items
| |||||||||
 Citation Citation |  Journal: To Be Published Journal: To Be PublishedTitle: Cryo-EM structure of RNA device 43 full-length, holo state Authors: Stagno JR / Deme JC / Lee Y-T / Wang Y-X / Lea SM | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Supplemental images |
|---|
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_41059.map.gz emd_41059.map.gz | 284.2 MB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-41059-v30.xml emd-41059-v30.xml emd-41059.xml emd-41059.xml | 19.2 KB 19.2 KB | Display Display |  EMDB header EMDB header |
| FSC (resolution estimation) |  emd_41059_fsc.xml emd_41059_fsc.xml | 16.5 KB | Display |  FSC data file FSC data file |
| Images |  emd_41059.png emd_41059.png | 41.7 KB | ||
| Masks |  emd_41059_msk_1.map emd_41059_msk_1.map | 325 MB |  Mask map Mask map | |
| Filedesc metadata |  emd-41059.cif.gz emd-41059.cif.gz | 5.3 KB | ||
| Others |  emd_41059_additional_1.map.gz emd_41059_additional_1.map.gz emd_41059_additional_2.map.gz emd_41059_additional_2.map.gz emd_41059_half_map_1.map.gz emd_41059_half_map_1.map.gz emd_41059_half_map_2.map.gz emd_41059_half_map_2.map.gz | 307 MB 269.6 MB 302 MB 302 MB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-41059 http://ftp.pdbj.org/pub/emdb/structures/EMD-41059 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41059 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41059 | HTTPS FTP |
-Validation report
| Summary document |  emd_41059_validation.pdf.gz emd_41059_validation.pdf.gz | 726 KB | Display |  EMDB validaton report EMDB validaton report |
|---|---|---|---|---|
| Full document |  emd_41059_full_validation.pdf.gz emd_41059_full_validation.pdf.gz | 725.5 KB | Display | |
| Data in XML |  emd_41059_validation.xml.gz emd_41059_validation.xml.gz | 23.6 KB | Display | |
| Data in CIF |  emd_41059_validation.cif.gz emd_41059_validation.cif.gz | 30.9 KB | Display | |
| Arichive directory |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41059 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41059 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41059 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41059 | HTTPS FTP |
-Related structure data
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- Map
Map
| File |  Download / File: emd_41059.map.gz / Format: CCP4 / Size: 325 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_41059.map.gz / Format: CCP4 / Size: 325 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | unsharpened map | ||||||||||||||||||||||||||||||||||||
| Projections & slices | Image control
Images are generated by Spider. | ||||||||||||||||||||||||||||||||||||
| Voxel size | X=Y=Z: 0.693 Å | ||||||||||||||||||||||||||||||||||||
| Density |
| ||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
|
-Supplemental data
-Mask #1
| File |  emd_41059_msk_1.map emd_41059_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
| Density Histograms |
-Additional map: B-factor sharpened, global
| File | emd_41059_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | B-factor sharpened, global | ||||||||||||
| Projections & Slices |
| ||||||||||||
| Density Histograms |
-Additional map: deepEMhancer postprocessed
| File | emd_41059_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | deepEMhancer postprocessed | ||||||||||||
| Projections & Slices |
| ||||||||||||
| Density Histograms |
-Half map: half map 2
| File | emd_41059_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | half map 2 | ||||||||||||
| Projections & Slices |
| ||||||||||||
| Density Histograms |
-Half map: half map 1
| File | emd_41059_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | half map 1 | ||||||||||||
| Projections & Slices |
| ||||||||||||
| Density Histograms |
- Sample components
Sample components
-Entire : Synthetic RNA device containing hammerhead ribozyme and tetracycl...
| Entire | Name: Synthetic RNA device containing hammerhead ribozyme and tetracycline-binding aptamer |
|---|---|
| Components |
|
-Supramolecule #1: Synthetic RNA device containing hammerhead ribozyme and tetracycl...
| Supramolecule | Name: Synthetic RNA device containing hammerhead ribozyme and tetracycline-binding aptamer type: complex / ID: 1 / Parent: 0 / Macromolecule list: #1 |
|---|---|
| Source (natural) | Organism: synthetic construct (others) |
| Molecular weight | Theoretical: 40.229 KDa |
-Macromolecule #1: RNA Device 43
| Macromolecule | Name: RNA Device 43 / type: rna / ID: 1 / Number of copies: 1 |
|---|---|
| Source (natural) | Organism: synthetic construct (others) |
| Molecular weight | Theoretical: 40.005895 KDa |
| Sequence | String: GUGAGGUGCA GGUACAUCCA GCUGAUGAGU CCCAAAUAGG ACAAAAAGGG AGAGGUGAAG AAUACGACCA CCUAGGCUCG AAAGAGCCU AAAACAUACC UUUCCUGGAU UCCACUGCUA UCCAC |
-Macromolecule #2: TETRACYCLINE
| Macromolecule | Name: TETRACYCLINE / type: ligand / ID: 2 / Number of copies: 1 / Formula: TAC |
|---|---|
| Molecular weight | Theoretical: 444.435 Da |
| Chemical component information | 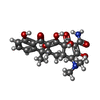 ChemComp-TAC: |
-Macromolecule #3: MAGNESIUM ION
| Macromolecule | Name: MAGNESIUM ION / type: ligand / ID: 3 / Number of copies: 15 / Formula: MG |
|---|---|
| Molecular weight | Theoretical: 24.305 Da |
-Macromolecule #4: water
| Macromolecule | Name: water / type: ligand / ID: 4 / Number of copies: 2 / Formula: HOH |
|---|---|
| Molecular weight | Theoretical: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | single particle reconstruction |
| Aggregation state | particle |
- Sample preparation
Sample preparation
| Concentration | 1.5 mg/mL | ||||||||
|---|---|---|---|---|---|---|---|---|---|
| Buffer | pH: 6.8 Component:
| ||||||||
| Grid | Model: Quantifoil R1.2/1.3 / Material: GOLD / Mesh: 300 / Pretreatment - Type: GLOW DISCHARGE / Pretreatment - Time: 60 sec. / Details: 30 s, 30 mA on both sides | ||||||||
| Vitrification | Cryogen name: ETHANE / Chamber humidity: 100 % / Chamber temperature: 284 K / Instrument: FEI VITROBOT MARK IV | ||||||||
| Details | in vitro transcribed RNA |
- Electron microscopy
Electron microscopy
| Microscope | TFS KRIOS |
|---|---|
| Image recording | Film or detector model: FEI FALCON IV (4k x 4k) / Average electron dose: 50.9 e/Å2 |
| Electron beam | Acceleration voltage: 300 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | Illumination mode: FLOOD BEAM / Imaging mode: BRIGHT FIELD / Nominal defocus max: 2.0 µm / Nominal defocus min: 0.5 µm |
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
 Movie
Movie Controller
Controller





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





























































