+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | 5TU-t1 - heterodimeric triplet polymerase ribozyme | |||||||||||||||||||||||||||
 マップデータ マップデータ | Map sharpened with deepEMhancer | |||||||||||||||||||||||||||
 試料 試料 |
| |||||||||||||||||||||||||||
 キーワード キーワード | Polymerase / Ribozyme / heterodimer / RNA | |||||||||||||||||||||||||||
| 生物種 | synthetic construct (人工物) | |||||||||||||||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 5.0 Å | |||||||||||||||||||||||||||
 データ登録者 データ登録者 | McRae EKS / Kristoffersen E / Gallego I / Hansen K / Holliger P / Andersen ES | |||||||||||||||||||||||||||
| 資金援助 |  デンマーク, デンマーク,  カナダ, カナダ,  英国, 英国,  ドイツ, 8件 ドイツ, 8件
| |||||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2024 ジャーナル: Proc Natl Acad Sci U S A / 年: 2024タイトル: Cryo-EM structure and functional landscape of an RNA polymerase ribozyme. 著者: Ewan K S McRae / Christopher J K Wan / Emil L Kristoffersen / Kalinka Hansen / Edoardo Gianni / Isaac Gallego / Joseph F Curran / James Attwater / Philipp Holliger / Ebbe S Andersen /   要旨: The emergence of an RNA replicase capable of self-replication is considered an important stage in the origin of life. RNA polymerase ribozymes (PR) - including a variant that uses trinucleotide ...The emergence of an RNA replicase capable of self-replication is considered an important stage in the origin of life. RNA polymerase ribozymes (PR) - including a variant that uses trinucleotide triphosphates (triplets) as substrates - have been created by in vitro evolution and are the closest functional analogues of the replicase, but the structural basis for their function is poorly understood. Here we use single-particle cryogenic electron microscopy (cryo-EM) and high-throughput mutation analysis to obtain the structure of a triplet polymerase ribozyme (TPR) apoenzyme and map its functional landscape. The cryo-EM structure at 5-Å resolution reveals the TPR as an RNA heterodimer comprising a catalytic subunit and a noncatalytic, auxiliary subunit, resembling the shape of a left hand with thumb and fingers at a 70° angle. The two subunits are connected by two distinct kissing-loop (KL) interactions that are essential for polymerase function. Our combined structural and functional data suggest a model for templated RNA synthesis by the TPR holoenzyme, whereby heterodimer formation and KL interactions preorganize the TPR for optimal primer-template duplex binding, triplet substrate discrimination, and templated RNA synthesis. These results provide a better understanding of TPR structure and function and should aid the engineering of more efficient PRs. | |||||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_40984.map.gz emd_40984.map.gz | 30.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-40984-v30.xml emd-40984-v30.xml emd-40984.xml emd-40984.xml | 20.5 KB 20.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_40984_fsc.xml emd_40984_fsc.xml | 6.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_40984.png emd_40984.png | 35.5 KB | ||
| マスクデータ |  emd_40984_msk_1.map emd_40984_msk_1.map | 34.3 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-40984.cif.gz emd-40984.cif.gz | 5.5 KB | ||
| その他 |  emd_40984_additional_1.map.gz emd_40984_additional_1.map.gz emd_40984_half_map_1.map.gz emd_40984_half_map_1.map.gz emd_40984_half_map_2.map.gz emd_40984_half_map_2.map.gz | 17.1 MB 31.9 MB 31.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-40984 http://ftp.pdbj.org/pub/emdb/structures/EMD-40984 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40984 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40984 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_40984_validation.pdf.gz emd_40984_validation.pdf.gz | 708.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_40984_full_validation.pdf.gz emd_40984_full_validation.pdf.gz | 708.1 KB | 表示 | |
| XML形式データ |  emd_40984_validation.xml.gz emd_40984_validation.xml.gz | 14 KB | 表示 | |
| CIF形式データ |  emd_40984_validation.cif.gz emd_40984_validation.cif.gz | 18.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40984 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40984 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40984 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40984 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_40984.map.gz / 形式: CCP4 / 大きさ: 34.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_40984.map.gz / 形式: CCP4 / 大きさ: 34.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map sharpened with deepEMhancer | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.29 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_40984_msk_1.map emd_40984_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
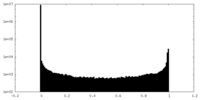
| 密度ヒストグラム |
-追加マップ: Unsharpened map
| ファイル | emd_40984_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
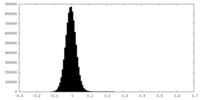
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_40984_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
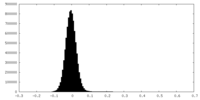
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_40984_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : 5TU-t1 - Triplet Polymerase Ribozyme
| 全体 | 名称: 5TU-t1 - Triplet Polymerase Ribozyme |
|---|---|
| 要素 |
|
-超分子 #1: 5TU-t1 - Triplet Polymerase Ribozyme
| 超分子 | 名称: 5TU-t1 - Triplet Polymerase Ribozyme / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Heterodimeric complex of two RNA strands comprising a functional triplet polymerase ribozyme. |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 92.7 KDa |
-分子 #1: RNA (135-MER)
| 分子 | 名称: RNA (135-MER) / タイプ: rna / ID: 1 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 43.328703 KDa |
| 配列 | 文字列: GACCAAUCUG CCCUCAGAGC UCGAGAACAU CUUCGGAUGC AGAGGAGGCA GGCUUCGGUG GCGCGAUAGC GCCAACGUCC UCAACCUCC AAUGCAUCCC ACCACAUGAU GAUGCCUGAA GAGCCUUGGU UUUUUG |
-分子 #2: RNA (152-MER)
| 分子 | 名称: RNA (152-MER) / タイプ: rna / ID: 2 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 48.929094 KDa |
| 配列 | 文字列: GGAUCUUCUC GAUCUAACAA AAAAGACAAA UCUGCCACAA AGCUUGAGAG CAUCUUCGGA UGCAGAGGCG GCAGCCUUCG GUGGCGCGA UAGCGCCAAC GUUCUCAACU AUGACACGCA AAACGCGUGC UCCGUUGAAU GGAGUUUAUC AUG |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 45 sec. / 前処理 - 雰囲気: AIR / 詳細: 15mA on a GloQube Plus |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 288 K / 装置: LEICA EM GP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 詳細 | Model was initially built using DRRAFTER and then improved using ISOLDE in ChimeraX, Coot, Phenix RSR and QRNAS. |
|---|---|
| 精密化 | プロトコル: AB INITIO MODEL |
| 得られたモデル |  PDB-8t2p: |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)