+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of NLRP3 open octamer | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Activated NLRP3 / Cryo-EM / IMMUNE SYSTEM | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報detection of biotic stimulus / molecular sensor activity / phosphatidylinositol phosphate binding / positive regulation of T-helper 2 cell differentiation / NLRP3 inflammasome complex assembly / positive regulation of T-helper 2 cell cytokine production / interphase microtubule organizing center / positive regulation of type 2 immune response / NLRP3 inflammasome complex / cysteine-type endopeptidase activator activity ...detection of biotic stimulus / molecular sensor activity / phosphatidylinositol phosphate binding / positive regulation of T-helper 2 cell differentiation / NLRP3 inflammasome complex assembly / positive regulation of T-helper 2 cell cytokine production / interphase microtubule organizing center / positive regulation of type 2 immune response / NLRP3 inflammasome complex / cysteine-type endopeptidase activator activity / peptidoglycan binding / osmosensory signaling pathway / phosphatidylinositol-4-phosphate binding / negative regulation of non-canonical NF-kappaB signal transduction / pattern recognition receptor signaling pathway / negative regulation of interleukin-1 beta production / pyroptotic inflammatory response / positive regulation of interleukin-4 production / microtubule organizing center / negative regulation of acute inflammatory response / The NLRP3 inflammasome / Purinergic signaling in leishmaniasis infection / signaling adaptor activity / protein maturation / positive regulation of interleukin-1 beta production / molecular condensate scaffold activity / defense response / positive regulation of non-canonical NF-kappaB signal transduction / Cytoprotection by HMOX1 / ADP binding / protein homooligomerization / cellular response to virus / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / negative regulation of inflammatory response / Metalloprotease DUBs / positive regulation of inflammatory response / positive regulation of NF-kappaB transcription factor activity / SARS-CoV-1 activates/modulates innate immune responses / cellular response to lipopolysaccharide / regulation of inflammatory response / protein-macromolecule adaptor activity / DNA-binding transcription factor binding / molecular adaptor activity / sequence-specific DNA binding / inflammatory response / Golgi membrane / innate immune response / apoptotic process / SARS-CoV-2 activates/modulates innate and adaptive immune responses / endoplasmic reticulum / signal transduction / positive regulation of transcription by RNA polymerase II / ATP hydrolysis activity / mitochondrion / extracellular region / ATP binding / identical protein binding / nucleus / membrane / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.39 Å | |||||||||
 データ登録者 データ登録者 | Yu X / Matico RE / Miller R / Schoubroeck BV / Grauwen K / Suarez J / Pietrak B / Haloi N / Yin Y / Tresadern GJ ...Yu X / Matico RE / Miller R / Schoubroeck BV / Grauwen K / Suarez J / Pietrak B / Haloi N / Yin Y / Tresadern GJ / Perez-Benito L / Lindahl E / Bottelbergs A / Oehlrich D / Opdenbosch NV / Sharma S | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Cryo-EM structures of NLRP3 reveal its self-activation mechanism 著者: Yu X / Matico RE / Miller R / Schoubroeck BV / Grauwen K / Suarez J / Pietrak B / Haloi N / Yin Y / Tresadern GJ / Perez-Benito L / Lindahl E / Bottelbergs A / Oehlrich D / Opdenbosch NV / Sharma S | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_40811.map.gz emd_40811.map.gz | 117.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-40811-v30.xml emd-40811-v30.xml emd-40811.xml emd-40811.xml | 16.2 KB 16.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_40811.png emd_40811.png | 106.1 KB | ||
| Filedesc metadata |  emd-40811.cif.gz emd-40811.cif.gz | 5.7 KB | ||
| その他 |  emd_40811_additional_1.map.gz emd_40811_additional_1.map.gz emd_40811_half_map_1.map.gz emd_40811_half_map_1.map.gz emd_40811_half_map_2.map.gz emd_40811_half_map_2.map.gz | 61.7 MB 116.1 MB 116.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-40811 http://ftp.pdbj.org/pub/emdb/structures/EMD-40811 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40811 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40811 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_40811.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_40811.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.82 Å | ||||||||||||||||||||||||||||||||||||
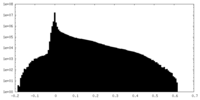
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: #1
| ファイル | emd_40811_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
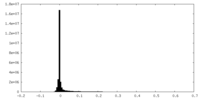
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_40811_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
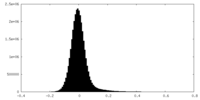
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_40811_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
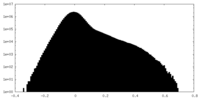
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : NLRP3 open octamer complex
| 全体 | 名称: NLRP3 open octamer complex |
|---|---|
| 要素 |
|
-超分子 #1: NLRP3 open octamer complex
| 超分子 | 名称: NLRP3 open octamer complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: NACHT, LRR and PYD domains-containing protein 3
| 分子 | 名称: NACHT, LRR and PYD domains-containing protein 3 / タイプ: protein_or_peptide / ID: 1 / コピー数: 8 / 光学異性体: LEVO EC番号: 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 103.675742 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: AEYCKMKKDY RKKYRKYVRS RFQCIEDRNA RLGESVSLNK RYTRLRLIKE HRSQQEREQE LLAIGKTKTC ESPVSPIKME LLFDPDDEH SEPVHTVVFQ GAAGIGKTIL ARKMMLDWAS GTLYQDRFDY LFYIHCREVS LVTQRSLGDL IMSCCPDPNP P IHKIVRKP ...文字列: AEYCKMKKDY RKKYRKYVRS RFQCIEDRNA RLGESVSLNK RYTRLRLIKE HRSQQEREQE LLAIGKTKTC ESPVSPIKME LLFDPDDEH SEPVHTVVFQ GAAGIGKTIL ARKMMLDWAS GTLYQDRFDY LFYIHCREVS LVTQRSLGDL IMSCCPDPNP P IHKIVRKP SRILFLMDGF DELQGAFDEH IGPLCTDWQK AERGDILLSS LIRKKLLPEA SLLITTRPVA LEKLQHLLDH PR HVEILGF SEAKRKEYFF KYFSDEAQAR AAFSLIQENE VLFTMCFIPL VCWIVCTGLK QQMESGKSLA QTSKTTTAVY VFF LSSLLQ PRGGSQEHGL CAHLWGLCSL AADGIWNQKI LFEESDLRNH GLQKADVSAF LRMNLFQKEV DCEKFYSFIH MTFQ EFFAA MYYLLEEEKE GRTNVPGSRL KLPSRDVTVL LENYGKFEKG YLIFVVRFLF GLVNQERTSY LEKKLSCKIS QQIRL ELLK WIEVKAKAKK LQIQPSQLEL FYCLYEMQEE DFVQRAMDYF PKIEINLSTR MDHMVSSFCI ENCHRVESLS LGFLHN MPK EEEEEEKEGR HLDMVQCVLP SSSHAACSHG LVNSHLTSSF CRGLFSVLST SQSLTELDLS DNSLGDPGMR VLCETLQ HP GCNIRRLWLG RCGLSHECCF DISLVLSSNQ KLVELDLSDN ALGDFGIRLL CVGLKHLLCN LKKLWLVSCC LTSACCQD L ASVLSTSHSL TRLYVGENAL GDSGVAILCE KAKNPQCNLQ KLGLVNSGLT SVCCSALSSV LSTNQNLTHL YLRGNTLGD KGIKLLCEGL LHPDCKLQVL ELDNCNLTSH CCWDLSTLLT SSQSLRKLSL GNNDLGDLGV MMFCEVLKQQ SCLLQNLGLS EMYFNYETK SALETLQEEK PELTVVFEPS W UniProtKB: NACHT, LRR and PYD domains-containing protein 3 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 52.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 1.2 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: INSILICO MODEL |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.39 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 466664 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)