+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structural basis and functional roles for Toll-like receptor binding to Latrophilin adhesion-GPCR in embryo development | |||||||||
 マップデータ マップデータ | Postprocessed map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | synapse / C. elegans / ADGR / GPCR / CELL ADHESION | |||||||||
| 生物種 |  | |||||||||
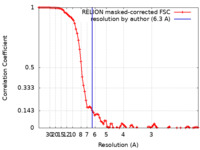
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 6.3 Å | |||||||||
 データ登録者 データ登録者 | Li J / Rosas GC / Arac D / Ozkan E | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Structural basis and functional roles for Toll-like receptor binding to Latrophilin adhesion-GPCR in embryo development 著者: Rosas GC / Li J / Smith J / Cheng S / Baltrusaitis E / Nawrocka W / Zhan M / Kratsios P / Arac D / Ozkan E | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_40786.map.gz emd_40786.map.gz | 59.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-40786-v30.xml emd-40786-v30.xml emd-40786.xml emd-40786.xml | 17.3 KB 17.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_40786_fsc.xml emd_40786_fsc.xml | 9.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_40786.png emd_40786.png | 50.9 KB | ||
| Filedesc metadata |  emd-40786.cif.gz emd-40786.cif.gz | 6.1 KB | ||
| その他 |  emd_40786_half_map_1.map.gz emd_40786_half_map_1.map.gz emd_40786_half_map_2.map.gz emd_40786_half_map_2.map.gz | 49.7 MB 49.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-40786 http://ftp.pdbj.org/pub/emdb/structures/EMD-40786 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40786 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40786 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_40786_validation.pdf.gz emd_40786_validation.pdf.gz | 823.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_40786_full_validation.pdf.gz emd_40786_full_validation.pdf.gz | 823.2 KB | 表示 | |
| XML形式データ |  emd_40786_validation.xml.gz emd_40786_validation.xml.gz | 15.9 KB | 表示 | |
| CIF形式データ |  emd_40786_validation.cif.gz emd_40786_validation.cif.gz | 21 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40786 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40786 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40786 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40786 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_40786.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_40786.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Postprocessed map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.063 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Halp map 1
| ファイル | emd_40786_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Halp map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 2
| ファイル | emd_40786_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : The binary complex of C. elegans TOL-1 with C. elegans LAT-1 extr...
| 全体 | 名称: The binary complex of C. elegans TOL-1 with C. elegans LAT-1 extracellular domains |
|---|---|
| 要素 |
|
-超分子 #1: The binary complex of C. elegans TOL-1 with C. elegans LAT-1 extr...
| 超分子 | 名称: The binary complex of C. elegans TOL-1 with C. elegans LAT-1 extracellular domains タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: The Binary complex includes C.elegans Toll-like receptor 1 (Tol-1) and C. elegans Latrophilin 1 (LAT-1). |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 172 KDa |
-分子 #1: Toll-like receptor 1/TOL-1
| 分子 | 名称: Toll-like receptor 1/TOL-1 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: ADPHHHHHHG SGLNDIFEAQ KIEWHEADPG YTDECPKFCK CAPDPVQPTS KLLLCDYSSK NTTITPIASS NYDQVANIRS LFISCDNNNF QFPDAYFKSL TALHHLRIVG CETTHFSVKL FEDLAALRRL ELDQISTAST SFEMTEDVLM PLARLEKFSL TRSRNIELPQ ...文字列: ADPHHHHHHG SGLNDIFEAQ KIEWHEADPG YTDECPKFCK CAPDPVQPTS KLLLCDYSSK NTTITPIASS NYDQVANIRS LFISCDNNNF QFPDAYFKSL TALHHLRIVG CETTHFSVKL FEDLAALRRL ELDQISTAST SFEMTEDVLM PLARLEKFSL TRSRNIELPQ RLLCSLPHLQ VLNISSNELP SLRREESCVA QQLLIVDLSR NRLTNIEQFL RGIPAIRQIS VAYNSIAELD LSLATPFLQQ LDAEANRIVD LTSLPGTVVH VNLAGNALKR VPDAVAELAS LVALNVSRNE IEAGNSSVFS SPELEMLDAS YNKLDSLPVE WLQKCEKRIA HLHLEHNSIE QLTGGVLANA TNLQTLDLSS NQLRVFRDEV LPENSKIGNL RLSNNSLELL EPSSLSGLKL ESLDLSHNKL TEVPAAIGKV EQLKKVDLSH NRIAKVYQYV LNKIKQLHTV DLSNNQLQSI GPYIFSDSSE LHSLDVSNNE ISLLFKDAFA RCPKLRKISM KMNKIKSLDE GLTEASGLRR LDVSHNEILV LKWSALPENL EILNADNNDI NLLTAASMSP STANLKSVSL SNNGITIMNA DQIPNSLESL DVSNNRLAKL GKTALAAKSQ LRRLNLKGNL LTVVATESMK VVEAVHPLKV EISENPLICD CQMGWMIGGA KPKVLIQDSE TASCSHAVDG HQIQIQSLSK KDLLCPYKSV CEPECICCQY GNCDCKSVCP ANCRCFRDDQ FNINIVRCHG NSSMVPKREF VVSELPVSAT EIILSGVTLP QLRTHSFIGR LRLQRLHING TGLRSIQPKA FHTLPALKTL DLSDNSLISL SGEEFLKCGE VSQLFLNGNR FSTLSRGIFE KLPNLKYLTL HNNSLEDIPQ VLHSTALSKI SLSSNPLRCD CSGGSQQHLH HRRDPKAHPF WEHNAAEWFS LHRHLVVDFP KVECWENVTK AFLTNDTTVL SAYPPNMGND VFVMPIEEFL RDYNSTICVP FSSGFFGQDP QNSDIQH |
-分子 #2: Latrophilin 1/LAT-1
| 分子 | 名称: Latrophilin 1/LAT-1 / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: AVPSNKPTTD ESGTISHTIC DGEAAELSCP AGKVISIVLG NYGRFSVAVC LPDNDIVPSN INCQNHKTKS ILEKKCNGDS MCYFTVDKKT FTEDPCPNTP KYLEVKYNCV VPATTTTTTT TTSTTTTDSS LIVDEEEEAQ KDALNSDVIK PVKKKEDVFC SATNRRGVNW ...文字列: AVPSNKPTTD ESGTISHTIC DGEAAELSCP AGKVISIVLG NYGRFSVAVC LPDNDIVPSN INCQNHKTKS ILEKKCNGDS MCYFTVDKKT FTEDPCPNTP KYLEVKYNCV VPATTTTTTT TTSTTTTDSS LIVDEEEEAQ KDALNSDVIK PVKKKEDVFC SATNRRGVNW QNTKSGTTSS APCPEGSSGK QLWACTEEGQ WLTEFPNSAG CESNWISSRN SVLSGVISSE DVSGLPEFLR NLGSETRRPM VGGDLPKVLH LLEKTVNVIA EESWAYQHLP LSNKGAVEVM NYMLRNQEIW GSWDVTKRKE FASRFILAAE KAMVASAKGM MTSAESNVIV QPAITVEISH KIKMSSQPTD YILFPSAALW NGQNVDNVNI PRDAILKINK DETQVFFSSF DNLGAQMTPS DVTVAIAGTD QTEVRKRRVV SRIVGASLIE NGKERRVENL TQPVRITFYH KESSVRHLSN PTCVWWNHHE LKWKPSGCKL SYHNKTMTSC DCTHLTHFAV LMDVRGHDLN EIDQTLLTHH HHHH |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.28 mg/mL |
|---|---|
| 緩衝液 | pH: 8.5 / 詳細: 10 mM Tris, pH 8.5, 150 mM NaCl |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 200 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 281.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 実像数: 3333 / 平均露光時間: 4.2 sec. / 平均電子線量: 60.1 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





 Z
Z Y
Y X
X