+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Isobutyryl-CoA mutase fused in the presence of GMPPCP | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | supramolecular complex / b12-binding / G-protein chaperone / mutase / ISOMERASE | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報isobutyryl-CoA mutase / pivalyl-CoA mutase activity / isobutyryl-CoA mutase activity / methylmalonyl-CoA mutase activity / acyl-CoA metabolic process / cobalamin binding / 加水分解酵素; 酸無水物に作用; GTPに作用・細胞または細胞小器官の運動に関与 / GTPase activity / GTP binding / magnesium ion binding 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Cupriavidus metallidurans CH34 (バクテリア) Cupriavidus metallidurans CH34 (バクテリア) | ||||||||||||
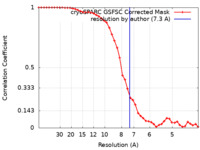
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 7.3 Å | ||||||||||||
 データ登録者 データ登録者 | Vaccaro FA / Drennan CL | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: J Biol Chem / 年: 2023 ジャーナル: J Biol Chem / 年: 2023タイトル: Structural insight into G-protein chaperone-mediated maturation of a bacterial adenosylcobalamin-dependent mutase. 著者: Francesca A Vaccaro / Daphne A Faber / Gisele A Andree / David A Born / Gyunghoon Kang / Dallas R Fonseca / Marco Jost / Catherine L Drennan /  要旨: G-protein metallochaperones are essential for the proper maturation of numerous metalloenzymes. The G-protein chaperone MMAA in humans (MeaB in bacteria) uses GTP hydrolysis to facilitate the ...G-protein metallochaperones are essential for the proper maturation of numerous metalloenzymes. The G-protein chaperone MMAA in humans (MeaB in bacteria) uses GTP hydrolysis to facilitate the delivery of adenosylcobalamin (AdoCbl) to AdoCbl-dependent methylmalonyl-CoA mutase, an essential metabolic enzyme. This G-protein chaperone also facilitates the removal of damaged cobalamin (Cbl) for repair. Although most chaperones are standalone proteins, isobutyryl-CoA mutase fused (IcmF) has a G-protein domain covalently attached to its target mutase. We previously showed that dimeric MeaB undergoes a 180° rotation to reach a state capable of GTP hydrolysis (an active G-protein state), in which so-called switch III residues of one protomer contact the G-nucleotide of the other protomer. However, it was unclear whether other G-protein chaperones also adopted this conformation. Here, we show that the G-protein domain in a fused system forms a similar active conformation, requiring IcmF oligomerization. IcmF oligomerizes both upon Cbl damage and in the presence of the nonhydrolyzable GTP analog, guanosine-5'-[(β,γ)-methyleno]triphosphate, forming supramolecular complexes observable by mass photometry and EM. Cryo-EM structural analysis reveals that the second protomer of the G-protein intermolecular dimer props open the mutase active site using residues of switch III as a wedge, allowing for AdoCbl insertion or damaged Cbl removal. With the series of structural snapshots now available, we now describe here the molecular basis of G-protein-assisted AdoCbl-dependent mutase maturation, explaining how GTP binding prepares a mutase for cofactor delivery and how GTP hydrolysis allows the mutase to capture the cofactor. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_40758.map.gz emd_40758.map.gz | 427.5 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-40758-v30.xml emd-40758-v30.xml emd-40758.xml emd-40758.xml | 17.2 KB 17.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_40758_fsc.xml emd_40758_fsc.xml | 4.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_40758.png emd_40758.png | 48.7 KB | ||
| その他 |  emd_40758_half_map_1.map.gz emd_40758_half_map_1.map.gz emd_40758_half_map_2.map.gz emd_40758_half_map_2.map.gz | 7.3 MB 7.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-40758 http://ftp.pdbj.org/pub/emdb/structures/EMD-40758 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40758 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40758 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_40758_validation.pdf.gz emd_40758_validation.pdf.gz | 651.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_40758_full_validation.pdf.gz emd_40758_full_validation.pdf.gz | 650.9 KB | 表示 | |
| XML形式データ |  emd_40758_validation.xml.gz emd_40758_validation.xml.gz | 11.2 KB | 表示 | |
| CIF形式データ |  emd_40758_validation.cif.gz emd_40758_validation.cif.gz | 14 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40758 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40758 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40758 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40758 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8staMC  8sslC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_40758.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_40758.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.0143 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_40758_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
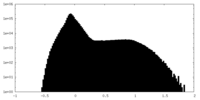
| 投影像・断面図 |
| ||||||||||||
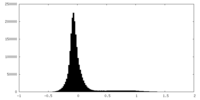
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_40758_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
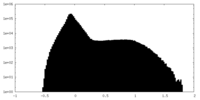
| 投影像・断面図 |
| ||||||||||||
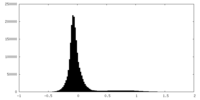
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Supramolecular complexes of isobutyryl-CoA mutase fused
| 全体 | 名称: Supramolecular complexes of isobutyryl-CoA mutase fused |
|---|---|
| 要素 |
|
-超分子 #1: Supramolecular complexes of isobutyryl-CoA mutase fused
| 超分子 | 名称: Supramolecular complexes of isobutyryl-CoA mutase fused タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Cupriavidus metallidurans CH34 (バクテリア) Cupriavidus metallidurans CH34 (バクテリア) |
-分子 #1: Isobutyryl-CoA mutase fused
| 分子 | 名称: Isobutyryl-CoA mutase fused / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO / EC番号: methylmalonyl-CoA mutase |
|---|---|
| 由来(天然) | 生物種:  Cupriavidus metallidurans CH34 (バクテリア) Cupriavidus metallidurans CH34 (バクテリア) |
| 分子量 | 理論値: 122.927586 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSSHHHHHH SSGLVPRGSH MTDLSDVSRT AAAKPPAVPG RGPANKVRFV TAASLFDGHD ASINIMRRIL QSQGCEVIHL GHNRSVQEV VTAALQEDVQ GIAISSYQGG HVEYFKYMID LLREHGGEHI QVFGGGGGVI VPDEIRELQA YGVARIYSPE D GQRMGLAG ...文字列: MGSSHHHHHH SSGLVPRGSH MTDLSDVSRT AAAKPPAVPG RGPANKVRFV TAASLFDGHD ASINIMRRIL QSQGCEVIHL GHNRSVQEV VTAALQEDVQ GIAISSYQGG HVEYFKYMID LLREHGGEHI QVFGGGGGVI VPDEIRELQA YGVARIYSPE D GQRMGLAG MITDMAQRCD IDLTRYAPTT LDTVVAGDRR ALAQLITALE NGKADPELVS ALHAQAKAAA VPVLGITGTG GA GKSSLTD ELIRRFRLDQ DDALSIAVIS IDPSRRKSGG ALLGDRIRMN AINHPNIFMR SLATREAGSE ISQALPDVIA ACK AARFDL VIVETSGIGQ GDAAIVPHVD LSLYVMTPEF GAASQLEKID MLDFADFVAI NKFDRKGAQD AWRDVAKQVQ RNRE QWHSR AEDMPVYGTQ ASRFNDDGVT MLYQGLVGAL GARGMSLKPG TLPNLEGRIS TGQNVIVPPA RSRYLAELAD TVRAY HRRV VAQSKLARER QQLRAAHDML QGAGHESAAL ETLASERDVS LGAVERKLLA MWPQMQQAYS GDEYVVKIRD KEIRTG LIS TTLSGTKIRK VVLPRFEDEG EILKWLMREN VPGSFPYTAG VFAFKREGED PTRMFAGEGD AFRTNRRFKL VSEGMEA KR LSTAFDSVTL YGEDPHERPD IYGKVGNSGV SIATLEDMKV LYDGFDLTNP STSVSMTING PAPTILAMFM NTAIDQQI D RFRADNGRDP TADEEAKIRA WVLQNVRGTV QADILKEDQG QNTCIFSTEF SLKVMGDIQE YFVHHQVRNF YSVSISGYH IAEAGANPIS QLAFTLANGF TYVEAYLARG MHIDDFAPNL SFFFSNGMDP EYSVLGRVAR RIWAVTMRDK YGANDRSQKL KYHIQTSGR SLHAQEIDFN DIRTTLQALI AIYDNCNSLH TNAYDEAITT PTAESVRRAL AIQLIINREW GVAKCENPNQ G SFLIEELT DLVEEAVLQE FERIAERGGV LGAMETGYQR GKIQEESLYY EQLKHDGTLP IIGVNTFRNP NGDPTPQTLE LA RSSEDEK QSQLHRLTEF HGAHQADAEA MLARLRQAVI DNRNVFAVLM DAVRVCSLGQ ITHALFEVGG QYRRNM UniProtKB: Fused isobutyryl-CoA mutase |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 濃度 | 0.25 mg/mL |
|---|---|
| 緩衝液 | pH: 8 詳細: 20 mM HEPES pH 8, 50 mM NaCl, 500 mM butyryl-CoA, 500 mM GMPPCP, 500 mM MgCl2 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 120 sec. / 前処理 - 気圧: 1.0 kPa 詳細: After glow discharge, the grid was coated with graphene oxide prior to use. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 281.15 K / 装置: FEI VITROBOT MARK IV |
| 詳細 | In the presence of the non-hydrolyzable GMPPCP, filamentous supramolecular complexes are formed |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 撮影したグリッド数: 1 / 実像数: 602 / 平均露光時間: 3.99 sec. / 平均電子線量: 42.81 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.1 µm / 最小 デフォーカス(公称値): 1.2 µm |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: B / Chain - Residue range: 22-1093 / Chain - Source name: PDB / Chain - Initial model type: experimental model 詳細: The initial model consisted of two different fragments: resi 22-442 and resi 443-1093 |
|---|---|
| 詳細 | Initial local fitting was done using ChimeraX and then Phenix real space refine was used for rigid body refinement of the defined fragments. |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
| 得られたモデル |  PDB-8sta: |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)