+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4061 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM map of E. coli BAM complex (BamABCDE) in DDM micelle | |||||||||
 マップデータ マップデータ | E. coli BamABCDE | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Bam protein complex / Gram-negative-bacterium-type cell outer membrane assembly / protein insertion into membrane / cell outer membrane / protein-macromolecule adaptor activity / cell adhesion / response to antibiotic / cell surface / identical protein binding / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
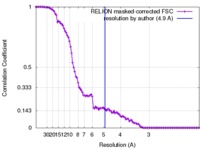
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.9 Å | |||||||||
 データ登録者 データ登録者 | Iadanza MG / Ranson NA / Radford SE / Higgins AJ / Schriffin B / Calabrase AN / Ashcroft AE / Brockwell DJ | |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2016 ジャーナル: Nat Commun / 年: 2016タイトル: Lateral opening in the intact β-barrel assembly machinery captured by cryo-EM. 著者: Matthew G Iadanza / Anna J Higgins / Bob Schiffrin / Antonio N Calabrese / David J Brockwell / Alison E Ashcroft / Sheena E Radford / Neil A Ranson /  要旨: The β-barrel assembly machinery (BAM) is a ∼203 kDa complex of five proteins (BamA-E), which is essential for viability in E. coli. BAM promotes the folding and insertion of β-barrel proteins ...The β-barrel assembly machinery (BAM) is a ∼203 kDa complex of five proteins (BamA-E), which is essential for viability in E. coli. BAM promotes the folding and insertion of β-barrel proteins into the outer membrane via a poorly understood mechanism. Several current models suggest that BAM functions through a 'lateral gating' motion of the β-barrel of BamA. Here we present a cryo-EM structure of the BamABCDE complex, at 4.9 Å resolution. The structure is in a laterally open conformation showing that gating is independent of BamB binding. We describe conformational changes throughout the complex and interactions between BamA, B, D and E, and the detergent micelle that suggest communication between BAM and the lipid bilayer. Finally, using an enhanced reconstitution protocol and functional assays, we show that for the outer membrane protein OmpT, efficient folding in vitro requires lateral gating in BAM. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4061.map.gz emd_4061.map.gz | 5.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4061-v30.xml emd-4061-v30.xml emd-4061.xml emd-4061.xml | 18.9 KB 18.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_4061_fsc.xml emd_4061_fsc.xml | 10.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4061.png emd_4061.png | 117.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4061 http://ftp.pdbj.org/pub/emdb/structures/EMD-4061 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4061 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4061 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_4061_validation.pdf.gz emd_4061_validation.pdf.gz | 232.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_4061_full_validation.pdf.gz emd_4061_full_validation.pdf.gz | 231.9 KB | 表示 | |
| XML形式データ |  emd_4061_validation.xml.gz emd_4061_validation.xml.gz | 11.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4061 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4061 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4061 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4061 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4061.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4061.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | E. coli BamABCDE | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.04 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : BAM complex
| 全体 | 名称: BAM complex |
|---|---|
| 要素 |
|
-超分子 #1: BAM complex
| 超分子 | 名称: BAM complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 分子量 | 実験値: 200 KDa |
-分子 #1: BamA
| 分子 | 名称: BamA / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAMKKLLIAS LLFSSATVYG AEGFVVKDIH FEGLQRVAVG AALLSMPVRT GDTVNDEDIS NTIRALFAT GNFEDVRVLR DGDTLLVQVK ERPTIASITF SGNKSVKDDM LKQNLEASGV R VGESLDRT TIADIEKGLE DFYYSVGKYS ASVKAVVTPL PRNRVDLKLV ...文字列: MAMKKLLIAS LLFSSATVYG AEGFVVKDIH FEGLQRVAVG AALLSMPVRT GDTVNDEDIS NTIRALFAT GNFEDVRVLR DGDTLLVQVK ERPTIASITF SGNKSVKDDM LKQNLEASGV R VGESLDRT TIADIEKGLE DFYYSVGKYS ASVKAVVTPL PRNRVDLKLV FQEGVSAEIQ QI NIVGNHA FTTDELISHF QLRDEVPWWN VVGDRKYQKQ KLAGDLETLR SYYLDRGYAR FNI DSTQVS LTPDKKGIYV TVNITEGDQY KLSGVEVSGN LAGHSAEIEQ LTKIEPGELY NGTK VTKME DDIKKLLGRY GYAYPRVQSM PEINDADKTV KLRVNVDAGN RFYVRKIRFE GNDTS KDAV LRREMRQMEG AWLGSDLVDQ GKERLNRLGF FETVDTDTQR VPGSPDQVDV VYKVKE RNT GSFNFGIGYG TESGVSFQAG VQQDNWLGTG YAVGINGTKN DYQTYAELSV TNPYFTV DG VSLGGRLFYN DFQADDADLS DYTNKSYGTD VTLGFPINEY NSLRAGLGYV HNSLSNMQ P QVAMWRYLYS MGEHPSTSDQ DNSFKTDDFT FNYGWTYNKL DRGYFPTDGS RVNLTGKVT IPGSDNEYYK VTLDTATYVP IDDDHKWVVL GRTRWGYGDG LGGKEMPFYE NFYAGGSSTV RGFQSNTIG PKAVYFPHQA SNYDPDYDYE CATQDGAKDL CKSDDAVGGN AMAVASLEFI T PTPFISDK YANSVRTSFF WDMGTVWDTN WDSSQYSGYP DYSDPSNIRM SAGIALQWMS PL GPLVFSY AQPFKKYDGD KAEQFQFNIG KTW |
-分子 #2: BamB
| 分子 | 名称: BamB / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MQLRKLLLPG LLSVTLLSGC SLFNSEEDVV KMSPLPTVEN QFTPTTAWST SVGSGIGNFY SNLHPALAD NVVYAADRAG LVKALNADDG KEIWSVSLAE KDGWFSKEPA LLSGGVTVSG G HVYIGSEK AQVYALNTSD GTVAWQTKVA GEALSRPVVS DGLVLIHTSN ...文字列: MQLRKLLLPG LLSVTLLSGC SLFNSEEDVV KMSPLPTVEN QFTPTTAWST SVGSGIGNFY SNLHPALAD NVVYAADRAG LVKALNADDG KEIWSVSLAE KDGWFSKEPA LLSGGVTVSG G HVYIGSEK AQVYALNTSD GTVAWQTKVA GEALSRPVVS DGLVLIHTSN GQLQALNEAD GA VKWTVNL DMPSLSLRGE SAPTTAFGAA VVGGDNGRVS AVLMEQGQMI WQQRISQATG STE IDRLSD VDTTPVVVNG VVFALAYNGN LTALDLRSGQ IMWKRELGSV NDFIVDGNRI YLVD QNDRV MALTIDGGVT LWTQSDLLHR LLTSPVLYNG NLVVGDSEGY LHWINVEDGR FVAQQ KVDS SGFQTEPVAA DGKLLIQAKD GTVYSITR |
-分子 #3: BamC
| 分子 | 名称: BamC / タイプ: protein_or_peptide / ID: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAYSVQKSRL AKVAGVSLVL LLAACSSDSR YKRQVSGDEA YLEAAPLAEL HAPAGMILPV TSGDYAIPV TNGSGAVGKA LDIRPPAQPL ALVSGARTQF TGDTASLLVE NGRGNTLWPQ V VSVLQAKN YTITQRDDAG QTLTTDWVQW NRLDEDEQYR GRYQISVKPQ ...文字列: MAYSVQKSRL AKVAGVSLVL LLAACSSDSR YKRQVSGDEA YLEAAPLAEL HAPAGMILPV TSGDYAIPV TNGSGAVGKA LDIRPPAQPL ALVSGARTQF TGDTASLLVE NGRGNTLWPQ V VSVLQAKN YTITQRDDAG QTLTTDWVQW NRLDEDEQYR GRYQISVKPQ GYQQAVTVKL LN LEQAGKP VADAASMQRY STEMMNVISA GLDKSATDAA NAAQNRASTT MDVQSAADDT GLP MLVVRG PFNVVWQRLP AALEKVGMKV TDSTRSQGNM AVTYKPLSDS DWQELGASDP GLAS GDYKL QVGDLDNRSS LQFIDPKGHT LTQSQNDALV AVFQAAFSK |
-分子 #4: BamD
| 分子 | 名称: BamD / タイプ: protein_or_peptide / ID: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTRMKYLVAA ATLSLFLAGC SGSKEEVPDN PPNEIYATAQ QKLQDGNWRQ AITQLEALDN RYPFGPYSQ QVQLDLIYAY YKNADLPLAQ AAIDRFIRLN PTHPNIDYVM YMRGLTNMAL D DSALQGFF GVDRSDRDPQ HARAAFSDFS KLVRGYPNSQ YTTDATKRLV ...文字列: MTRMKYLVAA ATLSLFLAGC SGSKEEVPDN PPNEIYATAQ QKLQDGNWRQ AITQLEALDN RYPFGPYSQ QVQLDLIYAY YKNADLPLAQ AAIDRFIRLN PTHPNIDYVM YMRGLTNMAL D DSALQGFF GVDRSDRDPQ HARAAFSDFS KLVRGYPNSQ YTTDATKRLV FLKDRLAKYE YS VAEYYTE RGAWVAVVNR VEGMLRDYPD TQATRDALPL MENAYRQMQM NAQAEKVAKI IAA NSSNT |
-分子 #5: BamE
| 分子 | 名称: BamE / タイプ: protein_or_peptide / ID: 5 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MRCKTLTAAA AVLLMLTAGC STLERVVYRP DINQGNYLTA NDVSKIRVGM TQQQVAYALG TPLMSDPFG TNTWFYVFRQ QPGHEGVTQQ TLTLTFNSSG VLTNIDNKPA LSGN |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 6.5 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R3.5/1 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.039 kPa / 詳細: Pelco EasyGlo | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 277 K / 装置: LEICA EM GP 詳細: Double Blotted: 3ul sample applied, hand blotted then additional 3ul sample applied and blotted and plunge frozen. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 BASE (4k x 4k) デジタル化 - サイズ - 横: 3840 pixel / デジタル化 - サイズ - 縦: 3712 pixel / デジタル化 - サンプリング間隔: 5.0 µm / 撮影したグリッド数: 1 / 実像数: 7204 / 平均露光時間: 10.0 sec. / 平均電子線量: 40.0 e/Å2 詳細: Collected in movie mode at 2 FPS Final data used frames 4-14 for total dose of 20 electrons per square Angstrom |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 最大 デフォーカス(補正後): 3.5 µm / 最小 デフォーカス(補正後): 1.5 µm / 倍率(補正後): 48076 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.6 mm / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 2.0 µm / 倍率(公称値): 48076 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
|---|---|
| 得られたモデル |  PDB-5ljo: |
 ムービー
ムービー コントローラー
コントローラー



















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)