+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | cytochrome bc1-cbb3 supercomplex from Pseudomonas aeruginosa | |||||||||
 マップデータ マップデータ | cytochrome bc1-cbb3 supercomplex from Pseudomonas aeruginosa (composite structure) | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Electron transport chain / Supercomplex / Pseudomonas aeruginosa / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cytochrome-c oxidase / cytochrome-c oxidase / respiratory chain complex III / oxidative phosphorylation / quinol-cytochrome-c reductase / quinol-cytochrome-c reductase activity / cytochrome-c oxidase activity / electron transport coupled proton transport / proton transmembrane transport / respiratory electron transport chain ...cytochrome-c oxidase / cytochrome-c oxidase / respiratory chain complex III / oxidative phosphorylation / quinol-cytochrome-c reductase / quinol-cytochrome-c reductase activity / cytochrome-c oxidase activity / electron transport coupled proton transport / proton transmembrane transport / respiratory electron transport chain / 2 iron, 2 sulfur cluster binding / periplasmic space / electron transfer activity / oxidoreductase activity / iron ion binding / heme binding / metal ion binding / membrane / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.7 Å | |||||||||
 データ登録者 データ登録者 | Di Trani JM / Rubinstein JL | |||||||||
| 資金援助 |  カナダ, 2件 カナダ, 2件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2023 ジャーナル: Proc Natl Acad Sci U S A / 年: 2023タイトル: Structure of the - respiratory supercomplex from . 著者: Justin M Di Trani / Andreea A Gheorghita / Madison Turner / Peter Brzezinski / Pia Ädelroth / Siavash Vahidi / P Lynne Howell / John L Rubinstein /   要旨: Energy conversion by electron transport chains occurs through the sequential transfer of electrons between protein complexes and intermediate electron carriers, creating the proton motive force that ...Energy conversion by electron transport chains occurs through the sequential transfer of electrons between protein complexes and intermediate electron carriers, creating the proton motive force that enables ATP synthesis and membrane transport. These protein complexes can also form higher order assemblies known as respiratory supercomplexes (SCs). The electron transport chain of the opportunistic pathogen is closely linked with its ability to invade host tissue, tolerate harsh conditions, and resist antibiotics but is poorly characterized. Here, we determine the structure of a SC that forms between the quinol:cytochrome oxidoreductase (cytochrome ) and one of the organism's terminal oxidases, cytochrome , which is found only in some bacteria. Remarkably, the SC structure also includes two intermediate electron carriers: a diheme cytochrome and a single heme cytochrome . Together, these proteins allow electron transfer from ubiquinol in cytochrome to oxygen in cytochrome . We also present evidence that different isoforms of cytochrome can participate in formation of this SC without changing the overall SC architecture. Incorporating these different subunit isoforms into the SC would allow the bacterium to adapt to different environmental conditions. Bioinformatic analysis focusing on structural motifs in the SC suggests that cytochrome - SCs also exist in other bacterial pathogens. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_40601.map.gz emd_40601.map.gz | 106.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-40601-v30.xml emd-40601-v30.xml emd-40601.xml emd-40601.xml | 45.4 KB 45.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_40601.png emd_40601.png | 117.8 KB | ||
| Filedesc metadata |  emd-40601.cif.gz emd-40601.cif.gz | 8.5 KB | ||
| その他 |  emd_40601_additional_1.map.gz emd_40601_additional_1.map.gz emd_40601_additional_2.map.gz emd_40601_additional_2.map.gz emd_40601_additional_3.map.gz emd_40601_additional_3.map.gz emd_40601_additional_4.map.gz emd_40601_additional_4.map.gz emd_40601_additional_5.map.gz emd_40601_additional_5.map.gz emd_40601_additional_6.map.gz emd_40601_additional_6.map.gz emd_40601_additional_7.map.gz emd_40601_additional_7.map.gz emd_40601_additional_8.map.gz emd_40601_additional_8.map.gz emd_40601_half_map_1.map.gz emd_40601_half_map_1.map.gz emd_40601_half_map_2.map.gz emd_40601_half_map_2.map.gz | 70.3 MB 131.7 MB 106.9 MB 70.5 MB 1.9 MB 131.8 MB 66.4 MB 107.2 MB 129.7 MB 129.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-40601 http://ftp.pdbj.org/pub/emdb/structures/EMD-40601 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40601 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40601 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_40601_validation.pdf.gz emd_40601_validation.pdf.gz | 898.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_40601_full_validation.pdf.gz emd_40601_full_validation.pdf.gz | 898 KB | 表示 | |
| XML形式データ |  emd_40601_validation.xml.gz emd_40601_validation.xml.gz | 14.1 KB | 表示 | |
| CIF形式データ |  emd_40601_validation.cif.gz emd_40601_validation.cif.gz | 16.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40601 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40601 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40601 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40601 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8smrMC  8snhC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_40601.map.gz / 形式: CCP4 / 大きさ: 139.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_40601.map.gz / 形式: CCP4 / 大きさ: 139.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | cytochrome bc1-cbb3 supercomplex from Pseudomonas aeruginosa (composite structure) | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.03 Å | ||||||||||||||||||||||||||||||||||||
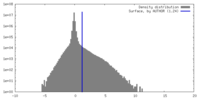
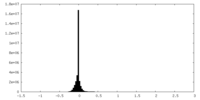
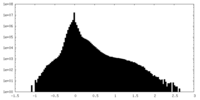
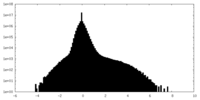
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
+追加マップ: cytochrome bc1-cbb3 supercomplex from Pseudomonas aeruginosa (Locally refined...
+追加マップ: cytochrome bc1-cbb3 supercomplex from Pseudomonas aeruginosa (Locally refined...
+追加マップ: cytochrome bc1 complex from Pseudomonas aeruginosa (Locally refined...
+追加マップ: cytochrome bc1 complex from Pseudomonas aeruginosa (Locally refined...
+追加マップ: cytochrome bc1 complex from Pseudomonas aeruginosa (Locally refined...
+追加マップ: cytochrome bc1-cbb3 supercomplex from Pseudomonas aeruginosa
+追加マップ: cytochrome bc1-cbb3 supercomplex from Pseudomonas aeruginosa (Locally refined...
+追加マップ: cytochrome bc1 complex from Pseudomonas aeruginosa
+ハーフマップ: cytochrome bc1-cbb3 supercomplex from Pseudomonas aeruginosa (Half map...
+ハーフマップ: cytochrome bc1-cbb3 supercomplex from Pseudomonas aeruginosa (Half map...
- 試料の構成要素
試料の構成要素
+全体 : cytochrome bc1-cbb3 supercomplex
+超分子 #1: cytochrome bc1-cbb3 supercomplex
+分子 #1: Ubiquinol-cytochrome c reductase iron-sulfur subunit
+分子 #2: Cytochrome b
+分子 #3: Cytochrome c1
+分子 #4: Cytochrome c4
+分子 #5: Cytochrome C5
+分子 #6: cytochrome-c oxidase
+分子 #7: Cbb3-type Cytochrome C oxidase subunit II
+分子 #8: Cbb3-type cytochrome c oxidase subunit
+分子 #9: FE2/S2 (INORGANIC) CLUSTER
+分子 #10: PROTOPORPHYRIN IX CONTAINING FE
+分子 #11: UBIQUINONE-10
+分子 #12: (2R)-2-(methoxymethyl)-4-{[(25R)-spirost-5-en-3beta-yl]oxy}butyl ...
+分子 #13: HEME C
+分子 #14: COPPER (II) ION
+分子 #15: CALCIUM ION
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE-PROPANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 51.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.9 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー


















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)