+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | E. coli 50S intermediate, srmB deletion strain, class: dead-B-a2 | |||||||||
 マップデータ マップデータ | dead-B-a2 class Particle stack from (https://doi.org/10.1101/2022.11.09.515851) was analyzed by CryoSPARC ab-initio subclassification. Aligned to 50S ref and resample to same grid. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Ribosome / 50S intermediate / RNP | |||||||||
| 生物種 |  | |||||||||
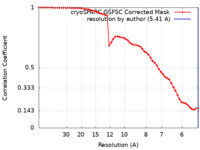
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 5.41 Å | |||||||||
 データ登録者 データ登録者 | Sheng K / Williamson JR | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Assembly landscape for the bacterial large ribosomal subunit. 著者: Kai Sheng / Ning Li / Jessica N Rabuck-Gibbons / Xiyu Dong / Dmitry Lyumkis / James R Williamson /  要旨: Assembly of ribosomes in bacteria is highly efficient, taking ~2-3 min, but this makes the abundance of assembly intermediates very low, which is a challenge for mechanistic understanding. Genetic ...Assembly of ribosomes in bacteria is highly efficient, taking ~2-3 min, but this makes the abundance of assembly intermediates very low, which is a challenge for mechanistic understanding. Genetic perturbations of the assembly process create bottlenecks where intermediates accumulate, facilitating structural characterization. We use cryo-electron microscopy, with iterative subclassification to identify intermediates in the assembly of the 50S ribosomal subunit from E. coli. The analysis of the ensemble of intermediates that spans the entire biogenesis pathway for the 50 S subunit was facilitated by a dimensionality reduction and cluster picking approach using PCA-UMAP-HDBSCAN. The identity of the cooperative folding units in the RNA with associated proteins is revealed, and the hierarchy of these units reveals a complete assembly map for all RNA and protein components. The assembly generally proceeds co-transcriptionally, with some flexibility in the landscape to ensure efficiency for this central cellular process under a variety of growth conditions. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_40519.map.gz emd_40519.map.gz | 12 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-40519-v30.xml emd-40519-v30.xml emd-40519.xml emd-40519.xml | 14.6 KB 14.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_40519_fsc.xml emd_40519_fsc.xml | 5.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_40519.png emd_40519.png | 73.7 KB | ||
| その他 |  emd_40519_half_map_1.map.gz emd_40519_half_map_1.map.gz emd_40519_half_map_2.map.gz emd_40519_half_map_2.map.gz | 14.5 MB 14.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-40519 http://ftp.pdbj.org/pub/emdb/structures/EMD-40519 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40519 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40519 | HTTPS FTP |
-関連構造データ
| 関連構造データ | C: 同じ文献を引用 ( |
|---|
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_40519.map.gz / 形式: CCP4 / 大きさ: 15.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_40519.map.gz / 形式: CCP4 / 大きさ: 15.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | dead-B-a2 class Particle stack from (https://doi.org/10.1101/2022.11.09.515851) was analyzed by CryoSPARC ab-initio subclassification. Aligned to 50S ref and resample to same grid. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.62 Å | ||||||||||||||||||||||||||||||||||||
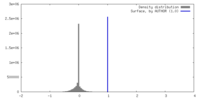
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: dead-B-a2 class Particle stack from (https://doi.org/10.1101/2022.11.09.515851) was analyzed...
| ファイル | emd_40519_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | dead-B-a2 class Particle stack from (https://doi.org/10.1101/2022.11.09.515851) was analyzed by CryoSPARC ab-initio subclassification. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
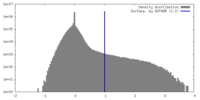
| 密度ヒストグラム |
-ハーフマップ: dead-B-a2 class Particle stack from (https://doi.org/10.1101/2022.11.09.515851) was analyzed...
| ファイル | emd_40519_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | dead-B-a2 class Particle stack from (https://doi.org/10.1101/2022.11.09.515851) was analyzed by CryoSPARC ab-initio subclassification. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : E. coli 50S assembly intermediate from deaD-deletion strain, grow...
| 全体 | 名称: E. coli 50S assembly intermediate from deaD-deletion strain, grow at 19 degree celsius |
|---|---|
| 要素 |
|
-超分子 #1: E. coli 50S assembly intermediate from deaD-deletion strain, grow...
| 超分子 | 名称: E. coli 50S assembly intermediate from deaD-deletion strain, grow at 19 degree celsius タイプ: complex / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 33.0 e/Å2 詳細: data were collected with tilts ranging from 0 to 60 degree at 10-degree increments |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 22500 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




































































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)