+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of a double loaded human UBA7-UBE2L6-ISG15 thioester mimetic complex (Form 2) | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Ligase / Signaling Protein | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報ISG15 activating enzyme activity / 合成酵素; C-S結合を形成; 酸とチオールを結合するもの / ISG15 transferase activity / positive regulation of protein oligomerization / ISG15-protein conjugation / regulation of type II interferon production / protein localization to mitochondrion / NS1 Mediated Effects on Host Pathways / response to type I interferon / Modulation of host responses by IFN-stimulated genes ...ISG15 activating enzyme activity / 合成酵素; C-S結合を形成; 酸とチオールを結合するもの / ISG15 transferase activity / positive regulation of protein oligomerization / ISG15-protein conjugation / regulation of type II interferon production / protein localization to mitochondrion / NS1 Mediated Effects on Host Pathways / response to type I interferon / Modulation of host responses by IFN-stimulated genes / negative regulation of type I interferon-mediated signaling pathway / E2 ubiquitin-conjugating enzyme / negative regulation of viral genome replication / RSV-host interactions / ubiquitin conjugating enzyme activity / positive regulation of interleukin-10 production / positive regulation of bone mineralization / negative regulation of protein ubiquitination / positive regulation of interferon-beta production / positive regulation of erythrocyte differentiation / ubiquitin binding / integrin-mediated signaling pathway / Negative regulators of DDX58/IFIH1 signaling / Termination of translesion DNA synthesis / PKR-mediated signaling / DDX58/IFIH1-mediated induction of interferon-alpha/beta / protein modification process / modification-dependent protein catabolic process / integrin binding / ISG15 antiviral mechanism / response to virus / positive regulation of type II interferon production / protein tag activity / protein polyubiquitination / ubiquitin-protein transferase activity / Interferon alpha/beta signaling / Antigen processing: Ubiquitination & Proteasome degradation / ubiquitin-dependent protein catabolic process / defense response to virus / defense response to bacterium / protein ubiquitination / Amyloid fiber formation / innate immune response / DNA damage response / ubiquitin protein ligase binding / SARS-CoV-2 activates/modulates innate and adaptive immune responses / extracellular region / nucleoplasm / ATP binding / nucleus / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
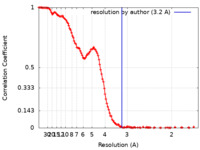
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.2 Å | |||||||||
 データ登録者 データ登録者 | Afsar M / Jia L / Ruben EA / Olsen SK | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Cryo-EM structures of Uba7 reveal the molecular basis for ISG15 activation and E1-E2 thioester transfer. 著者: Mohammad Afsar / GuanQun Liu / Lijia Jia / Eliza A Ruben / Digant Nayak / Zuberwasim Sayyad / Priscila Dos Santos Bury / Kristin E Cano / Anindita Nayak / Xiang Ru Zhao / Ankita Shukla / ...著者: Mohammad Afsar / GuanQun Liu / Lijia Jia / Eliza A Ruben / Digant Nayak / Zuberwasim Sayyad / Priscila Dos Santos Bury / Kristin E Cano / Anindita Nayak / Xiang Ru Zhao / Ankita Shukla / Patrick Sung / Elizabeth V Wasmuth / Michaela U Gack / Shaun K Olsen /  要旨: ISG15 plays a crucial role in the innate immune response and has been well-studied due to its antiviral activity and regulation of signal transduction, apoptosis, and autophagy. ISG15 is a ubiquitin- ...ISG15 plays a crucial role in the innate immune response and has been well-studied due to its antiviral activity and regulation of signal transduction, apoptosis, and autophagy. ISG15 is a ubiquitin-like protein that is activated by an E1 enzyme (Uba7) and transferred to a cognate E2 enzyme (UBE2L6) to form a UBE2L6-ISG15 intermediate that functions with E3 ligases that catalyze conjugation of ISG15 to target proteins. Despite its biological importance, the molecular basis by which Uba7 catalyzes ISG15 activation and transfer to UBE2L6 is unknown as there is no available structure of Uba7. Here, we present cryo-EM structures of human Uba7 in complex with UBE2L6, ISG15 adenylate, and ISG15 thioester intermediate that are poised for catalysis of Uba7-UBE2L6-ISG15 thioester transfer. Our structures reveal a unique overall architecture of the complex compared to structures from the ubiquitin conjugation pathway, particularly with respect to the location of ISG15 thioester intermediate. Our structures also illuminate the molecular basis for Uba7 activities and for its exquisite specificity for ISG15 and UBE2L6. Altogether, our structural, biochemical, and human cell-based data provide significant insights into the functions of Uba7, UBE2L6, and ISG15 in cells. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_40407.map.gz emd_40407.map.gz | 166.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-40407-v30.xml emd-40407-v30.xml emd-40407.xml emd-40407.xml | 21.7 KB 21.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_40407_fsc.xml emd_40407_fsc.xml | 16.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_40407.png emd_40407.png | 21.7 KB | ||
| マスクデータ |  emd_40407_msk_1.map emd_40407_msk_1.map | 178 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-40407.cif.gz emd-40407.cif.gz | 7.1 KB | ||
| その他 |  emd_40407_half_map_1.map.gz emd_40407_half_map_1.map.gz emd_40407_half_map_2.map.gz emd_40407_half_map_2.map.gz | 163.2 MB 163.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-40407 http://ftp.pdbj.org/pub/emdb/structures/EMD-40407 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40407 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40407 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8se9MC  8seaC  8sebC  8sv8C  40410 C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_40407.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_40407.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8332 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_40407_msk_1.map emd_40407_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_40407_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_40407_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cryo-EM structure of a double loaded human UBA7-UBE2L6-ISG15 thio...
| 全体 | 名称: Cryo-EM structure of a double loaded human UBA7-UBE2L6-ISG15 thioester mimetic complex (Form 2) |
|---|---|
| 要素 |
|
-超分子 #1: Cryo-EM structure of a double loaded human UBA7-UBE2L6-ISG15 thio...
| 超分子 | 名称: Cryo-EM structure of a double loaded human UBA7-UBE2L6-ISG15 thioester mimetic complex (Form 2) タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 160 KDa |
-分子 #1: Ubiquitin-like modifier-activating enzyme 7
| 分子 | 名称: Ubiquitin-like modifier-activating enzyme 7 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 111.822102 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MDALDASKLL DEELYSRQLY VLGSPAMQRI QGARVLVSGL QGLGAEVAKN LVLMGVGSLT LHDPHPTCWS DLAAQFLLSE QDLERSRAE ASQELLAQLN RAVQVVVHTG DITEDLLLDF QVVVLTAAKL EEQLKVGTLC HKHGVCFLAA DTRGLVGQLF C DFGEDFTV ...文字列: MDALDASKLL DEELYSRQLY VLGSPAMQRI QGARVLVSGL QGLGAEVAKN LVLMGVGSLT LHDPHPTCWS DLAAQFLLSE QDLERSRAE ASQELLAQLN RAVQVVVHTG DITEDLLLDF QVVVLTAAKL EEQLKVGTLC HKHGVCFLAA DTRGLVGQLF C DFGEDFTV QDPTEAEPLT AAIQHISQGS PGILTLRKGA NTHYFRDGDL VTFSGIEGMV ELNDCDPRSI HVREDGSLEI GD TTTFSRY LRGGAITEVK RPKTVRHKSL DTALLQPHVV AQSSQEVHHA HCLHQAFCAL HKFQHLHGRP PQPWDPVDAE TVV GLARDL EPLKRTEEEP LEEPLDEALV RTVALSSAGV LSPMVAMLGA VAAQEVLKAI SRKFMPLDQW LYFDALDCLP EDGE LLPSP EDCALRGSRY DGQIAVFGAG FQEKLRRQHY LLVGAGAIGC ELLKVFALVG LGAGNSGGLT VVDMDHIERS NLSRQ FLFR SQDVGRPKAE VAAAAARGLN PDLQVIPLTY PLDPTTEHIY GDNFFSRVDG VAAALDSFQA RRYVAARCTH YLKPLL EAG TSGTWGSATV FMPHVTEAYR APASAAASED APYPVCTVRY FPSTAEHTLQ WARHEFEELF RLSAETINHH QQAHTSL AD MDEPQTLTLL KPVLGVLRVR PQNWQDCVAW ALGHWKLCFH YGIKQLLRHF PPNKVLEDGT PFWSGPKQCP QPLEFDTN Q DTHLLYVLAA ANLYAQMHGL PGSQDWTALR ELLKLLPQPD PQQMAPIFAS NLELASASAE FGPEQQKELN KALEVWSVG PPLKPLMFEK DDDSNFHVDF VVAAASLRCQ NYGIPPVNRA QSKRIVGQII PAIATTTAAV AGLLGLELYK VVSGPRPRSA FRHSYLHLA ENYLIRYMPF APAIQTFHHL KWTSWDRLKV PAGQPERTLE SLLAHLQEQH GLRVRILLHG SALLYAAGWS P EKQAQHLP LRVTELVQQL TGQAPAPGQR VLVLELSCEG DDEDTAFPPL HYEL UniProtKB: Ubiquitin-like modifier-activating enzyme 7 |
-分子 #2: Ubiquitin-like protein ISG15
| 分子 | 名称: Ubiquitin-like protein ISG15 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 17.163701 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGWDLTVKML AGNEFQVSLS SSMSVSELKA QITQKIGVHA FQQRLAVHPS GVALQDRVPL ASQGLGPGST VLLVVDKCDE PLSILVRNN KGRSSTYEVR LTQTVAHLKQ QVSGLEGVQD DLFWLTFEGK PLEDQLPLGE YGLKPLSTVF MNLRLRGG UniProtKB: Ubiquitin-like protein ISG15 |
-分子 #3: Ubiquitin/ISG15-conjugating enzyme E2 L6
| 分子 | 名称: Ubiquitin/ISG15-conjugating enzyme E2 L6 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO / EC番号: E2 ubiquitin-conjugating enzyme |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 17.643377 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MASMRVVKEL EDLQKKPPPY LRNLSSDDAN VLVWHALLLP DQPPYHLKAF NLRISFPPEY PFKPPMIKFT TKIYHPNVDE NGQICLPII SSENWKPSTK TSQVLEALNV LVNRPNIREP KRMDLADLLT QNPELFRKNA EEFTLRFGVD RPS UniProtKB: Ubiquitin/ISG15-conjugating enzyme E2 L6 |
-分子 #4: ADENOSINE MONOPHOSPHATE
| 分子 | 名称: ADENOSINE MONOPHOSPHATE / タイプ: ligand / ID: 4 / コピー数: 1 / 式: AMP |
|---|---|
| 分子量 | 理論値: 347.221 Da |
| Chemical component information |  ChemComp-AMP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 80.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.888 µm / 最小 デフォーカス(公称値): 0.1 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)