+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4033 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | In situ atomic-resolution structure of the baseplate antenna complex in Chlorobaculum tepidum obtained combining solid-state NMR spectroscopy, cryo electron microscopy and polarization spectroscopy | |||||||||||||||
 マップデータ マップデータ | 3D reconstruction of the carotenosome CsmA-Bchl a baseplate obtained by cryo electron microscopy | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | photosynthesis / light-harvesting protein / binds bacteriochlorophyll a / oligomeric complex / bacteriochlorophyll binding protein | |||||||||||||||
| 機能・相同性 | chlorosome envelope / Bacteriochlorophyll c-binding protein / Bacteriochlorophyll c-binding superfamily / Bacteriochlorophyll C binding protein / bacteriochlorophyll binding / photosynthesis / metal ion binding / Bacteriochlorophyll c-binding protein 機能・相同性情報 機能・相同性情報 | |||||||||||||||
| 生物種 |  Chlorobaculum tepidum (バクテリア) / Chlorobaculum tepidum (バクテリア) /  Chlorobium tepidum (strain ATCC 49652 / DSM 12025 / NBRC 103806 / TLS) (バクテリア) Chlorobium tepidum (strain ATCC 49652 / DSM 12025 / NBRC 103806 / TLS) (バクテリア) | |||||||||||||||
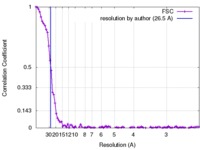
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 26.5 Å | |||||||||||||||
 データ登録者 データ登録者 | Nielsen JT / Kulminskaya NV | |||||||||||||||
| 資金援助 |  デンマーク, デンマーク,  Estonia, Estonia,  ハンガリー, 4件 ハンガリー, 4件
| |||||||||||||||
 引用 引用 | ジャーナル: Angew Chem Int Ed Engl / 年: 2012 タイトル: In situ solid-state NMR spectroscopy of protein in heterogeneous membranes: the baseplate antenna complex of Chlorobaculum tepidum. 著者: Natalia V Kulminskaya / Marie Ø Pedersen / Morten Bjerring / Jarl Underhaug / Mette Miller / Niels-Ulrik Frigaard / Jakob T Nielsen / Niels Chr Nielsen /  要旨: A clever combination: an in situ solid-state NMR analysis of CsmA proteins in the heterogeneous environment of the photoreceptor of Chlorobaculum tepidum is reported. Using different combinations of ...A clever combination: an in situ solid-state NMR analysis of CsmA proteins in the heterogeneous environment of the photoreceptor of Chlorobaculum tepidum is reported. Using different combinations of 2D and 3D solid-state NMR spectra, 90 % of the CsmA resonances are assigned and provide on the basis of chemical shift data information about the structure and conformation of CsmA in the CsmA-bacteriochlorophyll a complex. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4033.map.gz emd_4033.map.gz | 10.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4033-v30.xml emd-4033-v30.xml emd-4033.xml emd-4033.xml | 21.1 KB 21.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_4033_fsc.xml emd_4033_fsc.xml | 10.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4033.png emd_4033.png | 102.4 KB | ||
| Filedesc metadata |  emd-4033.cif.gz emd-4033.cif.gz | 6.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4033 http://ftp.pdbj.org/pub/emdb/structures/EMD-4033 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4033 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4033 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_4033_validation.pdf.gz emd_4033_validation.pdf.gz | 431 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_4033_full_validation.pdf.gz emd_4033_full_validation.pdf.gz | 430.6 KB | 表示 | |
| XML形式データ |  emd_4033_validation.xml.gz emd_4033_validation.xml.gz | 11.4 KB | 表示 | |
| CIF形式データ |  emd_4033_validation.cif.gz emd_4033_validation.cif.gz | 14.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4033 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4033 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4033 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4033 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4033.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4033.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3D reconstruction of the carotenosome CsmA-Bchl a baseplate obtained by cryo electron microscopy | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.2 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Carotenosome organelle consisting of csma proteins and bchla ante...
| 全体 | 名称: Carotenosome organelle consisting of csma proteins and bchla antenna molecules. |
|---|---|
| 要素 |
|
-超分子 #1: Carotenosome organelle consisting of csma proteins and bchla ante...
| 超分子 | 名称: Carotenosome organelle consisting of csma proteins and bchla antenna molecules. タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: #1 詳細: Cryo-EM data of the ice-embedded isolated carotenosomes reveals molecular complex structure. Captured images clearly show supramolecular organization of the protein rows, originating from the ...詳細: Cryo-EM data of the ice-embedded isolated carotenosomes reveals molecular complex structure. Captured images clearly show supramolecular organization of the protein rows, originating from the BChl a - CsmA baseplate |
|---|---|
| 由来(天然) | 生物種:  Chlorobaculum tepidum (バクテリア) / Organelle: carotenosome Chlorobaculum tepidum (バクテリア) / Organelle: carotenosome |
-分子 #1: Bacteriochlorophyll c-binding protein
| 分子 | 名称: Bacteriochlorophyll c-binding protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 14 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Chlorobium tepidum (strain ATCC 49652 / DSM 12025 / NBRC 103806 / TLS) (バクテリア) Chlorobium tepidum (strain ATCC 49652 / DSM 12025 / NBRC 103806 / TLS) (バクテリア) |
| 分子量 | 理論値: 6.160034 KDa |
| 組換発現 | 生物種:  Chlorobaculum tepidum (バクテリア) Chlorobaculum tepidum (バクテリア) |
| 配列 | 文字列: MSGGGVFTDI LAAAGRIFEV MVEGHWETVG MLFDSLGKGT MRINRNAYGS MGGGSLRGS UniProtKB: Bacteriochlorophyll c-binding protein |
-分子 #2: BACTERIOCHLOROPHYLL A
| 分子 | 名称: BACTERIOCHLOROPHYLL A / タイプ: ligand / ID: 2 / コピー数: 14 / 式: BCL |
|---|---|
| 分子量 | 理論値: 911.504 Da |
| Chemical component information |  ChemComp-BCL: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| グリッド | モデル: Quantifoil R2/4 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK II |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) 実像数: 270 / 平均電子線量: 8.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 最大 デフォーカス(補正後): 4.0 µm / 最小 デフォーカス(補正後): 1.7 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 倍率(公称値): 59000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)