+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Mycobacterium phage Adjutor | |||||||||
 マップデータ マップデータ | Sharpened map of ewald sphere corrected postprocess. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | T=7 / HK97 / Tailed bacteriophage / Capsid / VIRUS | |||||||||
| 機能・相同性 | Uncharacterized protein / Capsid decoration protein / Major capsid protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Mycobacterium phage Adjutor (ファージ) Mycobacterium phage Adjutor (ファージ) | |||||||||
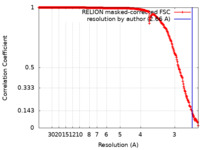
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.66 Å | |||||||||
 データ登録者 データ登録者 | Podgorski JM / White SJ | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2025 ジャーナル: Nat Commun / 年: 2025タイトル: Stabilization mechanism accommodating genome length variation in evolutionarily related viral capsids. 著者: Jennifer M Podgorski / Joshua Podgorski / Lawrence Abad / Deborah Jacobs-Sera / Krista G Freeman / Colin Brown / Graham F Hatfull / Antoni Luque / Simon J White /  要旨: Tailed bacteriophages are one of the most numerous and diverse group of viruses. They store their genome at quasi-crystalline densities in capsids built from multiple copies of proteins adopting the ...Tailed bacteriophages are one of the most numerous and diverse group of viruses. They store their genome at quasi-crystalline densities in capsids built from multiple copies of proteins adopting the HK97-fold. The high density of the genome exerts an internal pressure, requiring a maturation process that reinforces their capsids. However, it is unclear how capsid stabilization strategies have adapted to accommodate the evolution of larger genomes in this virus group. Here we characterize a capsid reinforcement mechanism in two evolutionary-related actinobacteriophages that modifies the length of a stabilization protein to accommodate a larger genome while maintaining the same capsid size. We use cryo-EM to reveal that capsids contain split hexamers of HK97-fold proteins with a stabilization protein in the chasm. The observation of split hexamers in mature capsids is unprecedented, so we rationalize this result mathematically, discovering that icosahedral capsids can be formed by all split or skewed hexamers as long as their T-number is not a multiple of three. Our results suggest that analogous stabilization mechanisms can be present in other icosahedral capsids, and they provide a strategy for engineering capsids accommodating larger DNA cargoes as gene delivery systems. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
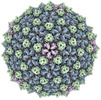
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_40271.map.gz emd_40271.map.gz | 1.7 GB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-40271-v30.xml emd-40271-v30.xml emd-40271.xml emd-40271.xml | 22.8 KB 22.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_40271_fsc.xml emd_40271_fsc.xml | 27.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_40271.png emd_40271.png | 293 KB | ||
| マスクデータ |  emd_40271_msk_1.map emd_40271_msk_1.map | 1.9 GB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-40271.cif.gz emd-40271.cif.gz | 6.4 KB | ||
| その他 |  emd_40271_additional_1.map.gz emd_40271_additional_1.map.gz emd_40271_half_map_1.map.gz emd_40271_half_map_1.map.gz emd_40271_half_map_2.map.gz emd_40271_half_map_2.map.gz | 1.8 GB 1.5 GB 1.5 GB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-40271 http://ftp.pdbj.org/pub/emdb/structures/EMD-40271 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40271 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40271 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_40271_validation.pdf.gz emd_40271_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_40271_full_validation.pdf.gz emd_40271_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_40271_validation.xml.gz emd_40271_validation.xml.gz | 35 KB | 表示 | |
| CIF形式データ |  emd_40271_validation.cif.gz emd_40271_validation.cif.gz | 47.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40271 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40271 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40271 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40271 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8sajMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_40271.map.gz / 形式: CCP4 / 大きさ: 1.9 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_40271.map.gz / 形式: CCP4 / 大きさ: 1.9 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map of ewald sphere corrected postprocess. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.2 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_40271_msk_1.map emd_40271_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Ewald sphere corrected map.
| ファイル | emd_40271_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Ewald sphere corrected map. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map of ewald sphere corrected map.
| ファイル | emd_40271_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map of ewald sphere corrected map. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map of ewald sphere corrected map.
| ファイル | emd_40271_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map of ewald sphere corrected map. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Mycobacterium phage Adjutor
| 全体 | 名称:  Mycobacterium phage Adjutor (ファージ) Mycobacterium phage Adjutor (ファージ) |
|---|---|
| 要素 |
|
-超分子 #1: Mycobacterium phage Adjutor
| 超分子 | 名称: Mycobacterium phage Adjutor / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 528321 / 生物種: Mycobacterium phage Adjutor / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア) |
| ウイルス殻 | Shell ID: 1 / 直径: 750.0 Å / T番号(三角分割数): 7 |
-分子 #1: Major capsid protein
| 分子 | 名称: Major capsid protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 7 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Mycobacterium phage Adjutor (ファージ) Mycobacterium phage Adjutor (ファージ) |
| 分子量 | 理論値: 45.525645 KDa |
| 配列 | 文字列: MKTATEGRVM RESFLEIIAV VGLSGHDNQG GYNTAGDIKY KTADGVSYDS LWNLFSNVTD EWNKHKSKMV QLMTFPVTNQ TEKVPRIGQ FGFEKASEFG VPESKRTELS FYQLAYDFED YDLAFRYTWK FLRDAPSSQI KAYHNQALQA DAKLIHRKVM E AIFDNRER ...文字列: MKTATEGRVM RESFLEIIAV VGLSGHDNQG GYNTAGDIKY KTADGVSYDS LWNLFSNVTD EWNKHKSKMV QLMTFPVTNQ TEKVPRIGQ FGFEKASEFG VPESKRTELS FYQLAYDFED YDLAFRYTWK FLRDAPSSQI KAYHNQALQA DAKLIHRKVM E AIFDNRER EADIEGLPYK VYPLYNGDNM IPPEYNGTTF STGHNHYLVS GGTKIDSADV EMAADHIREH GYTEENGTQL IA FAHKAEI QEVRRFRFGQ TNNNSAVANY DFVQSQGESP LYLPNADGLL GKQPQSMWKG LRVKGSYDDV LWIEEPTMPA GYV LFLATG GTLAQQNLVG LREHEDAAWR GLRQIPGNQT RYPLIDSFYQ RSFGTGIRQR GGAVVLQIKA SGTYDIPTKW TNGG GFE UniProtKB: Major capsid protein |
-分子 #2: gp_16 (Minor Capsid Protein)
| 分子 | 名称: gp_16 (Minor Capsid Protein) / タイプ: protein_or_peptide / ID: 2 / コピー数: 7 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Mycobacterium phage Adjutor (ファージ) Mycobacterium phage Adjutor (ファージ) |
| 分子量 | 理論値: 13.766352 KDa |
| 配列 | 文字列: MARYDKYNPY GGGFRAPLAA DWTDADAGKL YAVGINNVGA VVKGAGQSGV AGVLVLTKGA KAGSIVDVMK FGEVVEFGPT SGTPGTDFG AAGTAYYADT STGAINSTSG EAKVKVGHTV GAQRLIVAVA DGVVDPSPAA UniProtKB: Capsid decoration protein |
-分子 #3: HNH endonuclease
| 分子 | 名称: HNH endonuclease / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Mycobacterium phage Adjutor (ファージ) Mycobacterium phage Adjutor (ファージ) |
| 分子量 | 理論値: 6.198103 KDa |
| 配列 | 文字列: MAKGVKKLPK RKGTNPIPRD KWNSDDIARR QLEQDQKLHL TTKGPHTGTN DSFK UniProtKB: Uncharacterized protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 10 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 283 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 検出モード: COUNTING / 実像数: 6664 / 平均電子線量: 30.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 詳細 | Amino acid sequence built into the map for a single major capsid protein and refined with Phenix. Model then used for rest of asymmetric unit and refined with Phenix. Final step involved using Isolde. |
|---|---|
| 精密化 | プロトコル: AB INITIO MODEL |
| 得られたモデル |  PDB-8saj: |
 ムービー
ムービー コントローラー
コントローラー



 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)