+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-ET reconstruction of WT HSV-1 NEC coat | |||||||||
 マップデータ マップデータ | Cryo-ET reconstruction of HSV-1 WT NEC coat | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | HSV-1 / Nuclear egress / NEC lattice / UL31 / UL34 / suppressor mutation / cryoET / VIRAL PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報exit of virus from host cell nucleus by nuclear egress / viral budding from nuclear membrane / host cell nuclear inner membrane / zinc ion binding / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Human alphaherpesvirus 1 strain 17 (ヘルペスウイルス) Human alphaherpesvirus 1 strain 17 (ヘルペスウイルス) | |||||||||
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 5.9 Å | |||||||||
 データ登録者 データ登録者 | Wang H / Draganova EB | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: PLoS Pathog / 年: 2024 ジャーナル: PLoS Pathog / 年: 2024タイトル: The universal suppressor mutation restores membrane budding defects in the HSV-1 nuclear egress complex by stabilizing the oligomeric lattice. 著者: Elizabeth B Draganova / Hui Wang / Melanie Wu / Shiqing Liao / Amber Vu / Gonzalo L Gonzalez-Del Pino / Z Hong Zhou / Richard J Roller / Ekaterina E Heldwein /   要旨: Nuclear egress is an essential process in herpesvirus replication whereby nascent capsids translocate from the nucleus to the cytoplasm. This initial step of nuclear egress-budding at the inner ...Nuclear egress is an essential process in herpesvirus replication whereby nascent capsids translocate from the nucleus to the cytoplasm. This initial step of nuclear egress-budding at the inner nuclear membrane-is coordinated by the nuclear egress complex (NEC). Composed of the viral proteins UL31 and UL34, NEC deforms the membrane around the capsid as the latter buds into the perinuclear space. NEC oligomerization into a hexagonal membrane-bound lattice is essential for budding because NEC mutants designed to perturb lattice interfaces reduce its budding ability. Previously, we identified an NEC suppressor mutation capable of restoring budding to a mutant with a weakened hexagonal lattice. Using an established in-vitro budding assay and HSV-1 infected cell experiments, we show that the suppressor mutation can restore budding to a broad range of budding-deficient NEC mutants thereby acting as a universal suppressor. Cryogenic electron tomography of the suppressor NEC mutant lattice revealed a hexagonal lattice reminiscent of wild-type NEC lattice instead of an alternative lattice. Further investigation using x-ray crystallography showed that the suppressor mutation promoted the formation of new contacts between the NEC hexamers that, ostensibly, stabilized the hexagonal lattice. This stabilization strategy is powerful enough to override the otherwise deleterious effects of mutations that destabilize the NEC lattice by different mechanisms, resulting in a functional NEC hexagonal lattice and restoration of membrane budding. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_40223.map.gz emd_40223.map.gz | 20.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-40223-v30.xml emd-40223-v30.xml emd-40223.xml emd-40223.xml | 15.8 KB 15.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_40223.png emd_40223.png | 58.3 KB | ||
| マスクデータ |  emd_40223_msk_1.map emd_40223_msk_1.map | 28.7 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-40223.cif.gz emd-40223.cif.gz | 5.4 KB | ||
| その他 |  emd_40223_half_map_1.map.gz emd_40223_half_map_1.map.gz emd_40223_half_map_2.map.gz emd_40223_half_map_2.map.gz | 21.1 MB 21.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-40223 http://ftp.pdbj.org/pub/emdb/structures/EMD-40223 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40223 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40223 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_40223_validation.pdf.gz emd_40223_validation.pdf.gz | 963.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_40223_full_validation.pdf.gz emd_40223_full_validation.pdf.gz | 962.9 KB | 表示 | |
| XML形式データ |  emd_40223_validation.xml.gz emd_40223_validation.xml.gz | 10.4 KB | 表示 | |
| CIF形式データ |  emd_40223_validation.cif.gz emd_40223_validation.cif.gz | 12.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40223 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40223 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40223 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40223 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8g6dC C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_40223.map.gz / 形式: CCP4 / 大きさ: 28.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_40223.map.gz / 形式: CCP4 / 大きさ: 28.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-ET reconstruction of HSV-1 WT NEC coat | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.69 Å | ||||||||||||||||||||||||||||||||||||
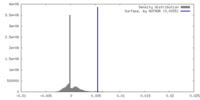
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
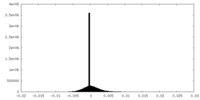
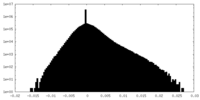
| ファイル |  emd_40223_msk_1.map emd_40223_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
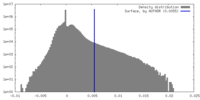
| 密度ヒストグラム |
-ハーフマップ: Cryo-ET reconstruction of HSV-1 WT NEC coat (half map 1)
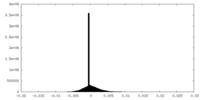
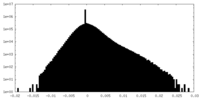
| ファイル | emd_40223_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-ET reconstruction of HSV-1 WT NEC coat (half map 1) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
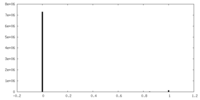
| 密度ヒストグラム |
-ハーフマップ: Cryo-ET reconstruction of HSV-1 WT NEC coat (half map 2)
| ファイル | emd_40223_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-ET reconstruction of HSV-1 WT NEC coat (half map 2) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
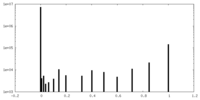
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Hexameric coat of the HSV-1 nuclear egress complex
| 全体 | 名称: Hexameric coat of the HSV-1 nuclear egress complex |
|---|---|
| 要素 |
|
-超分子 #1: Hexameric coat of the HSV-1 nuclear egress complex
| 超分子 | 名称: Hexameric coat of the HSV-1 nuclear egress complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Human alphaherpesvirus 1 strain 17 (ヘルペスウイルス) Human alphaherpesvirus 1 strain 17 (ヘルペスウイルス) |
-分子 #1: UL31
| 分子 | 名称: UL31 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Human alphaherpesvirus 1 strain 17 (ヘルペスウイルス) Human alphaherpesvirus 1 strain 17 (ヘルペスウイルス) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GPGSYDTDPH RRGSRPGPYH GKERRRSRSS AAGGTLGVVR RASRKSLPPH ARKQELCLHE RQRYRGLFAA LAQTPSEEIA IVRSLSVPLV KTTPVSLPFC LDQTVADNCL TLSGMGYYLG IGGCCPACNA GDGRFAATSR EALILAFVQQ INTIFEHRAF LASLVVLADR ...文字列: GPGSYDTDPH RRGSRPGPYH GKERRRSRSS AAGGTLGVVR RASRKSLPPH ARKQELCLHE RQRYRGLFAA LAQTPSEEIA IVRSLSVPLV KTTPVSLPFC LDQTVADNCL TLSGMGYYLG IGGCCPACNA GDGRFAATSR EALILAFVQQ INTIFEHRAF LASLVVLADR HNAPLQDLLA GILGQPELFF VHTILRGGGA CDPRLLFYPD PTYGGHMLYV IFPGTSAHLH YRLIDRMLTA CPGYRFVAHV WQSTFVLVVR RNAEKPTDAE IPTVSAADIY CKMRDISFDG GLMLEYQRLY ATFDEFPPP UniProtKB: Nuclear egress protein 1 |
-分子 #2: UL34
| 分子 | 名称: UL34 / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Human alphaherpesvirus 1 strain 17 (ヘルペスウイルス) Human alphaherpesvirus 1 strain 17 (ヘルペスウイルス) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GPLGSPEFPG RPMAGLGKPY TGHPGDAFEG LVQRIRLIVP STLRGGDGEA GPYSPSSLPS RCAFQFHGHD GSDESFPIEY VLRLMNDWAE VPCNPYLRIQ NTGVSVLFQG FFHRPHNAPG GAITPERTNV ILGSTETTGL SLGDLDTIKG RLGLDARPMM ASMWISCFVR ...文字列: GPLGSPEFPG RPMAGLGKPY TGHPGDAFEG LVQRIRLIVP STLRGGDGEA GPYSPSSLPS RCAFQFHGHD GSDESFPIEY VLRLMNDWAE VPCNPYLRIQ NTGVSVLFQG FFHRPHNAPG GAITPERTNV ILGSTETTGL SLGDLDTIKG RLGLDARPMM ASMWISCFVR MPRVQLAFRF MGPEDAGRTR RILCRAAEQA ITRRRRTRRS REAYGAEAGL GVAGTGFRAR GD UniProtKB: Nuclear egress protein 2 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | 2D array |
- 試料調製
試料調製
| 緩衝液 | pH: 7 構成要素:
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: EMS Lacey Carbon / 材質: GOLD | ||||||||||||
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 2.34 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 5.5 µm / 最小 デフォーカス(公称値): 2.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - 点群: C6 (6回回転対称) / 解像度のタイプ: BY AUTHOR / 解像度: 5.9 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用したサブトモグラム数: 34223 |
|---|---|
| 抽出 | トモグラム数: 43 / 使用した粒子像数: 83385 |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー



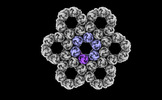


 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)