+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of HKU1B RBD with TMPRSS2 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | HKU1B / RBD / TMPRSS2 / VIRAL PROTEIN/HYDROLASE / VIRAL PROTEIN-HYDROLASE complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報transmembrane protease serine 2 / protein autoprocessing / Attachment and Entry / serine-type peptidase activity / viral translation / Induction of Cell-Cell Fusion / entry receptor-mediated virion attachment to host cell / Attachment and Entry / host cell endoplasmic reticulum-Golgi intermediate compartment membrane / positive regulation of viral entry into host cell ...transmembrane protease serine 2 / protein autoprocessing / Attachment and Entry / serine-type peptidase activity / viral translation / Induction of Cell-Cell Fusion / entry receptor-mediated virion attachment to host cell / Attachment and Entry / host cell endoplasmic reticulum-Golgi intermediate compartment membrane / positive regulation of viral entry into host cell / receptor-mediated virion attachment to host cell / endocytosis involved in viral entry into host cell / serine-type endopeptidase activity / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / host cell plasma membrane / virion membrane / proteolysis / extracellular exosome / extracellular region / nucleoplasm / identical protein binding / membrane / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  Candidatus Accumulibacter adiacens (バクテリア) Candidatus Accumulibacter adiacens (バクテリア) | |||||||||
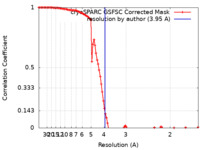
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.95 Å | |||||||||
 データ登録者 データ登録者 | Gao X / Cui S / Ding W / Zhu K / Shang K / Zhu H | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell Discov / 年: 2024 ジャーナル: Cell Discov / 年: 2024タイトル: Structural basis for the interaction between human coronavirus HKU1 spike receptor binding domain and its receptor TMPRSS2. 著者: Xiaopan Gao / Kaixiang Zhu / Lin Wang / Kun Shang / Lei Hua / Bo Qin / Hongtao Zhu / Wei Ding / Sheng Cui /  | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_39502.map.gz emd_39502.map.gz | 107.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-39502-v30.xml emd-39502-v30.xml emd-39502.xml emd-39502.xml | 18 KB 18 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_39502_fsc.xml emd_39502_fsc.xml | 10.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_39502.png emd_39502.png | 70.8 KB | ||
| Filedesc metadata |  emd-39502.cif.gz emd-39502.cif.gz | 6.5 KB | ||
| その他 |  emd_39502_half_map_1.map.gz emd_39502_half_map_1.map.gz emd_39502_half_map_2.map.gz emd_39502_half_map_2.map.gz | 107.6 MB 107.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-39502 http://ftp.pdbj.org/pub/emdb/structures/EMD-39502 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-39502 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-39502 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_39502_validation.pdf.gz emd_39502_validation.pdf.gz | 732.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_39502_full_validation.pdf.gz emd_39502_full_validation.pdf.gz | 731.8 KB | 表示 | |
| XML形式データ |  emd_39502_validation.xml.gz emd_39502_validation.xml.gz | 18.3 KB | 表示 | |
| CIF形式データ |  emd_39502_validation.cif.gz emd_39502_validation.cif.gz | 23.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-39502 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-39502 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-39502 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-39502 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8yqqMC  8yoyC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_39502.map.gz / 形式: CCP4 / 大きさ: 115.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_39502.map.gz / 形式: CCP4 / 大きさ: 115.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.81 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : HKU1B-RBD with TMPRSS2 receptor
| 全体 | 名称: HKU1B-RBD with TMPRSS2 receptor |
|---|---|
| 要素 |
|
-超分子 #1: HKU1B-RBD with TMPRSS2 receptor
| 超分子 | 名称: HKU1B-RBD with TMPRSS2 receptor / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Spike protein S1
| 分子 | 名称: Spike protein S1 / タイプ: protein_or_peptide / ID: 1 詳細: Sequence reference for Candidatus Accumulibacter adiacens (2954378) is not available in UniProt at the time of biocuration. Current sequence reference is from UniProt ID Q0ZME7. コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Candidatus Accumulibacter adiacens (バクテリア) Candidatus Accumulibacter adiacens (バクテリア) |
| 分子量 | 理論値: 31.703457 KDa |
| 組換発現 | 生物種:  Baculovirus expression vector pFastBac1-HM (ウイルス) Baculovirus expression vector pFastBac1-HM (ウイルス) |
| 配列 | 文字列: NLPDCDIDNW LNNVSVPSPL NWERRIFSNC NFNLSTLLRL VHVDSFSCNN LDKSKIFGSC FNSITVDKFA IPNRRRDDLQ LGSSGFLQS SNYKIDISSS SCQLYYSLPL VNVTINNFNP SSWNRRYGFG SFNLSSYDVV YSDHCFSVNS DFCPCADPSV V NSCAKSKP ...文字列: NLPDCDIDNW LNNVSVPSPL NWERRIFSNC NFNLSTLLRL VHVDSFSCNN LDKSKIFGSC FNSITVDKFA IPNRRRDDLQ LGSSGFLQS SNYKIDISSS SCQLYYSLPL VNVTINNFNP SSWNRRYGFG SFNLSSYDVV YSDHCFSVNS DFCPCADPSV V NSCAKSKP PSAICPAGTK YRHCDLDTTL YVKNWCRCSC LPDPISTYSP NTCPQKKVVV GIGEHCPGLG INEEKCGTQL NH SSCFCSP DAFLGWSFDS CISNNRCNIF SNFIFNGINS GTTCSNDL UniProtKB: Spike glycoprotein |
-分子 #2: Transmembrane protease serine 2
| 分子 | 名称: Transmembrane protease serine 2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO / EC番号: transmembrane protease serine 2 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 42.381848 KDa |
| 組換発現 | 生物種:  Baculovirus expression vector pFastBac1-HM (ウイルス) Baculovirus expression vector pFastBac1-HM (ウイルス) |
| 配列 | 文字列: MGSKCSNSGI ECDSSGTCIN PSNWCDGVSH CPGGEDENRC VRLYGPNFIL QVYSSQRKSW HPVCQDDWNE NYGRAACRDM GYKNNFYSS QGIVDDSGST SFMKLNTSAG NVDIYKKLYH SDACSSKAVV SLRCIACGVN LNSSRQSRIV GGESALPGAW P WQVSLHVQ ...文字列: MGSKCSNSGI ECDSSGTCIN PSNWCDGVSH CPGGEDENRC VRLYGPNFIL QVYSSQRKSW HPVCQDDWNE NYGRAACRDM GYKNNFYSS QGIVDDSGST SFMKLNTSAG NVDIYKKLYH SDACSSKAVV SLRCIACGVN LNSSRQSRIV GGESALPGAW P WQVSLHVQ NVHVCGGSII TPEWIVTAAH CVEKPLNNPW HWTAFAGILR QSFMFYGAGY QVEKVISHPN YDSKTKNNDI AL MKLQKPL TFNDLVKPVC LPNPGMMLQP EQLCWISGWG ATEEKGKTSE VLNAAKVLLI ETQRCNSRYV YDNLITPAMI CAG FLQGNV DSCQGDAGGP LVTSKNNIWW LIGDTSWGSG CAKAYRPGVY GNVMVFTDWI YRQMRADG UniProtKB: Transmembrane protease serine 2 |
-分子 #3: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 3 / コピー数: 6 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 55.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.8 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)