[日本語] English
 万見
万見- EMDB-39411: Cryo-EM structure of NRG1A(LRR) in complex with EDS1-SAG101-(ADPr-ATP) -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of NRG1A(LRR) in complex with EDS1-SAG101-(ADPr-ATP) | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Plant immunity / ETI / NRG1A / EDS1 / SAG101 / ADPr-ATP / PLANT PROTEIN / PLANT PROTEIN-HYDROLASE complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of defense response to oomycetes / positive regulation of leaf senescence / aerenchyma formation / EDS1 disease-resistance complex / leaf abscission / systemic acquired resistance, salicylic acid mediated signaling pathway / systemic acquired resistance / plant-type hypersensitive response / response to singlet oxygen / positive regulation of defense response to bacterium ...positive regulation of defense response to oomycetes / positive regulation of leaf senescence / aerenchyma formation / EDS1 disease-resistance complex / leaf abscission / systemic acquired resistance, salicylic acid mediated signaling pathway / systemic acquired resistance / plant-type hypersensitive response / response to singlet oxygen / positive regulation of defense response to bacterium / lipase activity / carboxylesterase / regulation of hydrogen peroxide metabolic process / carboxylesterase activity / carboxylic ester hydrolase activity / lipid catabolic process / positive regulation of defense response to virus by host / endomembrane system / chloroplast / defense response / ADP binding / lipid metabolic process / defense response to Gram-negative bacterium / response to hypoxia / endoplasmic reticulum / protein homodimerization activity / ATP binding / nucleus / membrane / cytoplasm / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |   | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.09 Å | |||||||||
 データ登録者 データ登録者 | Huang S / Xiao Y / Chai J | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2025 ジャーナル: Nature / 年: 2025タイトル: Balanced plant helper NLR activation by a modified host protein complex. 著者: Shijia Huang / Junli Wang / Ridan Song / Aolin Jia / Yu Xiao / Yue Sun / Lin Wang / Dennis Mahr / Zhongshou Wu / Zhifu Han / Xin Li / Jane E Parker / Jijie Chai /    要旨: Nucleotide-binding leucine-rich repeat (NLR) receptors play crucial roles in plant immunity by sensing pathogen effectors. In Arabidopsis, certain sensor NLRs function as NADases to catalyse the ...Nucleotide-binding leucine-rich repeat (NLR) receptors play crucial roles in plant immunity by sensing pathogen effectors. In Arabidopsis, certain sensor NLRs function as NADases to catalyse the production of second messengers, which can be recognized by enhanced disease susceptibility 1 (EDS1) with its partner senescence-associated gene 101 (SAG101), to activate helper NLR N requirement gene 1 (NRG1). A cryoelectron microscopy structure shows that second-messenger-activated EDS1-SAG101 mainly contacts the leucine-rich repeat domain of NRG1A to mediate the formation of an induced EDS1-SAG101-NRG1A complex. Structural comparisons show that binding of a second messenger induces conformational changes in EDS1-SAG101, which are recognized by NRG1A, leading to its allosteric activation. We further show that an inhibitory NRG1 family member, NRG1C, efficiently outcompetes NRG1A for binding to second-messenger-activated EDS1-SAG101. These findings uncover mechanisms for NRG1A activation through its recognition of a modified host EDS1-SAG101 complex, and NRG1A inhibition by NRG1C through sequestration of the activated EDS1-SAG101, thus shedding light on the activation and constraint of a central plant immune response system. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_39411.map.gz emd_39411.map.gz | 4.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-39411-v30.xml emd-39411-v30.xml emd-39411.xml emd-39411.xml | 21.2 KB 21.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_39411.png emd_39411.png | 119.6 KB | ||
| Filedesc metadata |  emd-39411.cif.gz emd-39411.cif.gz | 7.1 KB | ||
| その他 |  emd_39411_half_map_1.map.gz emd_39411_half_map_1.map.gz emd_39411_half_map_2.map.gz emd_39411_half_map_2.map.gz | 40.8 MB 40.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-39411 http://ftp.pdbj.org/pub/emdb/structures/EMD-39411 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-39411 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-39411 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_39411_validation.pdf.gz emd_39411_validation.pdf.gz | 723.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_39411_full_validation.pdf.gz emd_39411_full_validation.pdf.gz | 722.9 KB | 表示 | |
| XML形式データ |  emd_39411_validation.xml.gz emd_39411_validation.xml.gz | 11.6 KB | 表示 | |
| CIF形式データ |  emd_39411_validation.cif.gz emd_39411_validation.cif.gz | 13.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-39411 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-39411 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-39411 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-39411 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8yn1MC  8yn0C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_39411.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_39411.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.85 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_39411_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_39411_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Ternary complex of NRG1A(LRR) with EDS1-SAG101-(ADPr-ATP)
| 全体 | 名称: Ternary complex of NRG1A(LRR) with EDS1-SAG101-(ADPr-ATP) |
|---|---|
| 要素 |
|
-超分子 #1: Ternary complex of NRG1A(LRR) with EDS1-SAG101-(ADPr-ATP)
| 超分子 | 名称: Ternary complex of NRG1A(LRR) with EDS1-SAG101-(ADPr-ATP) タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Protein EDS1
| 分子 | 名称: Protein EDS1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 70.715953 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: AFEALTGING DLITRSWSAS KQAYLTERYH KEEAGAVVIF AFQPSFSEKD FFDPDNKSSF GEIKLNRVQF PCMRKIGKGD VATVNEAFL KNLEAIIDPR TSFQASVEMA VRSRKQIVFT GHSSGGATAI LATVWYLEKY FIRNPNVYLE PRCVTFGAPL V GDSIFSHA ...文字列: AFEALTGING DLITRSWSAS KQAYLTERYH KEEAGAVVIF AFQPSFSEKD FFDPDNKSSF GEIKLNRVQF PCMRKIGKGD VATVNEAFL KNLEAIIDPR TSFQASVEMA VRSRKQIVFT GHSSGGATAI LATVWYLEKY FIRNPNVYLE PRCVTFGAPL V GDSIFSHA LGREKWSRFF VNFVSRFDIV PRIMLARKAS VEETLPHVLA QLDPRKSSVQ ESEQRITEFY TRVMRDTSTV AN QAVCELT GSAEAFLETL SSFLELSPYR PAGTFVFSTE KRLVAVNNSD AILQMLFYTS QASDEQEWSL IPFRSIRDHH SYE ELVQSM GKKLFNHLDG ENSIESTLND LGVSTRGRQY VQAALEEEKK RVENQKKIIQ VIEQERFLKK LAWIEDEYKP KCQA HKNGY YDSFKVSNEE NDFKANVKRA ELAGVFDEVL GLMKKCQLPD EFEGDIDWIK LATRYRRLVE PLDIANYHRH LKNED TGPY MKRGRPTRYI YAQRGYEHYI LKPNGMIAED VFWNKVNGLN LGLQLEEIQE TLKNSGSECG SCFWAEVEEL KGKPYE EVE VRVKTLEGML GEWITDGEVD DKEIFLEGST FRKWWITLPK NHKSHSPLRD Y UniProtKB: Protein EDS1 |
-分子 #2: Senescence-associated carboxylesterase 101
| 分子 | 名称: Senescence-associated carboxylesterase 101 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO / EC番号: carboxylesterase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 61.677875 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SSSSLKGSAL GKLVVTSGLL HSSWSKILEI HNPPYSNHDP GLQVSKKKKD SGLEFQIHRE EKFTLVVFSA PPICRSSSSD STLLHVKDK ENPFPFLCSE NNPSFSLHTP AFNLFTSAST SLTYLKSELL QTLKSEKPVI ITGAALGGSV ASLYTLWLLE T IEPTLKRP ...文字列: SSSSLKGSAL GKLVVTSGLL HSSWSKILEI HNPPYSNHDP GLQVSKKKKD SGLEFQIHRE EKFTLVVFSA PPICRSSSSD STLLHVKDK ENPFPFLCSE NNPSFSLHTP AFNLFTSAST SLTYLKSELL QTLKSEKPVI ITGAALGGSV ASLYTLWLLE T IEPTLKRP LCITFGSPLI GDASLQQILE NSVRNSCFLH VVSAQTRIKM DFFKPFGTFL ICFDSGCVCI EDHVAVTELL NG VHDSGLV DYSQVLNRLD QSMLSLADSR LIPEDVIKGI EKRAEMKNLR FDMMFKKLND MKISMAYIEW YKKKCKEVKI GYY DRFKTQ LAFPSKEFDI NIKNHHKSEL NRFWKSVVEE VERRPQSDAS ILKRRFLFSG NNYRRMIEPL DIAEYYLEGR KEYR TTGRS HHYVMLEKWF GMESILIEKE RCKKRDLSDL LTFDSCFWAE VEDSLIVINQ LNTTVGMRDD VREVLTRKLV EFEGY VWEI ITKREVSPEI FLEESSFMKW WKEYKKIKGF NSSYLTEFMN TRKYESYGK UniProtKB: Senescence-associated carboxylesterase 101 |
-分子 #3: Probable disease resistance protein At5g66900
| 分子 | 名称: Probable disease resistance protein At5g66900 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 46.461461 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SFDALDPNLK ECFLDMGSFL EDQKIRASVI IDMWVELYGK GSSILYMYLE DLASQNLLKL VPLGTNEHED GFYNDFLVTQ HDILRELAI CQSEFKENLE RKRLNLEILE NTFPDWCLNT INASLLSIST DDLFSSKWLE MDCPNVEALV LNLSSSDYAL P SFISGMKK ...文字列: SFDALDPNLK ECFLDMGSFL EDQKIRASVI IDMWVELYGK GSSILYMYLE DLASQNLLKL VPLGTNEHED GFYNDFLVTQ HDILRELAI CQSEFKENLE RKRLNLEILE NTFPDWCLNT INASLLSIST DDLFSSKWLE MDCPNVEALV LNLSSSDYAL P SFISGMKK LKVLTITNHG FYPARLSNFS CLSSLPNLKR IRLEKVSITL LDIPQLQLSS LKKLSLVMCS FGEVFYDTED IV VSNALSK LQEIDIDYCY DLDELPYWIS EIVSLKTLSI TNCNKLSQLP EAIGNLSRLE VLRLCSSMNL SELPEATEGL SNL RFLDIS HCLGLRKLPQ EIGKLQNLKK ISMRKCSGCE LPESVTNLEN LEVKCDEETG LLWERLKPKM RNLRVQEEEI EHNL NLLQM F UniProtKB: Probable disease resistance protein At5g66900 |
-分子 #4: ADENOSINE-5-DIPHOSPHORIBOSE
| 分子 | 名称: ADENOSINE-5-DIPHOSPHORIBOSE / タイプ: ligand / ID: 4 / コピー数: 1 / 式: APR |
|---|---|
| 分子量 | 理論値: 559.316 Da |
| Chemical component information | 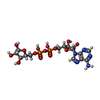 ChemComp-APR: |
-分子 #5: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 5 / コピー数: 1 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: PDB ENTRY PDBモデル - PDB ID: |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.09 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 275262 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





































