+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of artificial protein nanocage mTIP120-Ba | |||||||||||||||||||||||||||
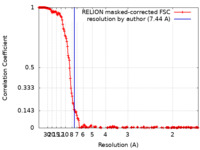
 マップデータ マップデータ | main map, job066, postprocess, C1, 7.44A resolution | |||||||||||||||||||||||||||
 試料 試料 |
| |||||||||||||||||||||||||||
 キーワード キーワード | Artificial designed protein complex / Protein cage / Protein nanoparticle / Metal-induced protein assembly / Protein metal complex / DE NOVO PROTEIN | |||||||||||||||||||||||||||
| 生物種 | synthetic construct (人工物) | |||||||||||||||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 7.44 Å | |||||||||||||||||||||||||||
 データ登録者 データ登録者 | Ohara N / Kawakami N / Arai R / Adachi N / Ikeda A / Senda T / Miyamoto K | |||||||||||||||||||||||||||
| 資金援助 |  日本, 8件 日本, 8件
| |||||||||||||||||||||||||||
 引用 引用 | ジャーナル: Angew Chem Int Ed Engl / 年: 2018 タイトル: Design of Hollow Protein Nanoparticles with Modifiable Interior and Exterior Surfaces. 著者: Norifumi Kawakami / Hiroki Kondo / Yuki Matsuzawa / Kaoru Hayasaka / Erika Nasu / Kenji Sasahara / Ryoichi Arai / Kenji Miyamoto /  要旨: Protein-based nanoparticles hold promise for a broad range of applications. Here, we report the production of a uniform anionic hollow protein nanoparticle, designated TIP60, which spontaneously ...Protein-based nanoparticles hold promise for a broad range of applications. Here, we report the production of a uniform anionic hollow protein nanoparticle, designated TIP60, which spontaneously assembles from a designed fusion protein subunit based on the geometric features of polyhedra. We show that TIP60 tolerates mutation and both its interior and exterior surfaces can be chemically modified. Moreover, TIP60 forms larger structures upon the addition of a cationic protein. Therefore, TIP60 can be used as a modifiable nano-building block for further molecular assembly. | |||||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_38931.map.gz emd_38931.map.gz | 561.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-38931-v30.xml emd-38931-v30.xml emd-38931.xml emd-38931.xml | 28.3 KB 28.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_38931_fsc.xml emd_38931_fsc.xml | 19.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_38931.png emd_38931.png | 109.7 KB | ||
| マスクデータ |  emd_38931_msk_1.map emd_38931_msk_1.map | 600.7 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-38931.cif.gz emd-38931.cif.gz | 6.7 KB | ||
| その他 |  emd_38931_additional_1.map.gz emd_38931_additional_1.map.gz emd_38931_half_map_1.map.gz emd_38931_half_map_1.map.gz emd_38931_half_map_2.map.gz emd_38931_half_map_2.map.gz | 485.1 MB 487.8 MB 487.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-38931 http://ftp.pdbj.org/pub/emdb/structures/EMD-38931 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-38931 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-38931 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_38931_validation.pdf.gz emd_38931_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_38931_full_validation.pdf.gz emd_38931_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_38931_validation.xml.gz emd_38931_validation.xml.gz | 26.5 KB | 表示 | |
| CIF形式データ |  emd_38931_validation.cif.gz emd_38931_validation.cif.gz | 35.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-38931 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-38931 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-38931 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-38931 | HTTPS FTP |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_38931.map.gz / 形式: CCP4 / 大きさ: 600.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_38931.map.gz / 形式: CCP4 / 大きさ: 600.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | main map, job066, postprocess, C1, 7.44A resolution | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.84 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_38931_msk_1.map emd_38931_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
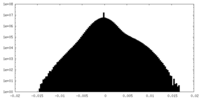
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: full map, job053, refine3d, C1, 8.40A resolution
| ファイル | emd_38931_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | full map, job053, refine3d, C1, 8.40A resolution | ||||||||||||
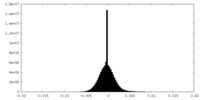
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 2, job053, refine3d, C1, 8.40A resolution
| ファイル | emd_38931_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 2, job053, refine3d, C1, 8.40A resolution | ||||||||||||
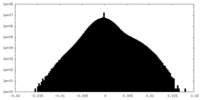
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 1, job053, refine3d, C1, 8.40A resolution
| ファイル | emd_38931_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 1, job053, refine3d, C1, 8.40A resolution | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : artificial protein cage, mTIP120-Ba
| 全体 | 名称: artificial protein cage, mTIP120-Ba |
|---|---|
| 要素 |
|
-超分子 #1: artificial protein cage, mTIP120-Ba
| 超分子 | 名称: artificial protein cage, mTIP120-Ba / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: metal-ion induced TIP120 (truncated icosahedral protein composed of 120-mer fusion proteins) complexed with barium ions |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 1.074 MDa |
-分子 #1: CoreN domain of artificial protein nanocage, mTIP120-Ba
| 分子 | 名称: CoreN domain of artificial protein nanocage, mTIP120-Ba タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSSHHHHHH SQDPKNIKIM RLVTGEDIIG NISESQGLIT IKKAFVIIPM QATP |
-分子 #2: CoreC domain of artificial protein nanocage, mTIP120-Ba
| 分子 | 名称: CoreC domain of artificial protein nanocage, mTIP120-Ba タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSSHHHHHH SQDPLEVLFQ GPGKPVQLVL SPWQPYTDDK EIVIDDSKVI TITSPKDDII KSYESHTRVL ENKQVEEILR LEKEIEDLQR MKEQQELSLT EASLQKLQER RDQELRRLEE E |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 5 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR / 詳細: The grid was washed by acetone prior to use. | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 291 K / 装置: FEI VITROBOT MARK IV / 詳細: Blotting time was 5 seconds (blot force 15). |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 1 / 実像数: 6191 / 平均露光時間: 4.66 sec. / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 120000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| 精密化 | 空間: REAL / プロトコル: OTHER / 温度因子: 582 / 当てはまり具合の基準: Correlation coefficient |
 ムービー
ムービー コントローラー
コントローラー




 Z
Z Y
Y X
X