+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Sialic acid bound form of Tripartite ATP-independent Periplasmic (TRAP) transporter from Fusobacterium nucleatum. | |||||||||
 マップデータ マップデータ | extracted map around pdb | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | sialic acid transporter / TRAP transporter / MEMBRANE PROTEIN / TRANSPORT PROTEIN. / MEMBRANE PROTEIN-IMMUNE SYSTEM complex | |||||||||
| 機能・相同性 | TRAP transporter large membrane protein DctM / TRAP C4-dicarboxylate transport system permease DctM subunit / : / Tripartite ATP-independent periplasmic transporters, DctQ component / Tripartite ATP-independent periplasmic transporter, DctM component / transmembrane transporter activity / plasma membrane / N-acetylneuraminate transporter small subunit 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Fusobacterium nucleatum (バクテリア) / Fusobacterium nucleatum (バクテリア) /  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.16 Å | |||||||||
 データ登録者 データ登録者 | Goyal P / Ramaswamy S / Vinothkumar KR | |||||||||
| 資金援助 |  インド, 2件 インド, 2件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2025 ジャーナル: Elife / 年: 2025タイトル: Molecular determinants of Neu5Ac binding to a tripartite ATP independent periplasmic (TRAP) transporter. 著者: Parveen Goyal / KanagaVijayan Dhanabalan / Mariafrancesca Scalise / Rosmarie Friemann / Cesare Indiveri / Renwick C J Dobson / Kutti R Vinothkumar / Subramanian Ramaswamy /       要旨: -Acetylneuraminic acid (Neu5Ac) is a negatively charged nine-carbon amino sugar that is often the peripheral sugar in human cell-surface glycoconjugates. Some bacteria scavenge, import, and ... -Acetylneuraminic acid (Neu5Ac) is a negatively charged nine-carbon amino sugar that is often the peripheral sugar in human cell-surface glycoconjugates. Some bacteria scavenge, import, and metabolize Neu5Ac or redeploy it on their cell surfaces for immune evasion. The import of Neu5Ac by many bacteria is mediated by tripartite ATP-independent periplasmic (TRAP) transporters. We have previously reported the structures of SiaQM, a membrane-embedded component of the TRAP transport system, (Currie et al., 2024). However, none of the published structures contain Neu5Ac bound to SiaQM. This information is critical for defining the transport mechanism and for further structure-activity relationship studies. Here, we report the structures of SiaQM with and without Neu5Ac. Both structures are in an inward (cytoplasmic side) facing conformation. The Neu5Ac-bound structure reveals the interactions of Neu5Ac with the transporter and its relationship with the Na binding sites. Two of the Na-binding sites are similar to those described previously. We identify a third metal-binding site that is further away and buried in the elevator domain. Ser300 and Ser345 interact with the C1-carboxylate group of Neu5Ac. Proteoliposome-based transport assays showed that Ser300-Neu5Ac interaction is critical for transport, whereas Ser345 is dispensable. Neu5Ac primarily interacts with residues in the elevator domain of the protein, thereby supporting the elevator with an operator mechanism. The residues interacting with Neu5Ac are conserved, providing fundamental information required to design inhibitors against this class of proteins. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_38925.map.gz emd_38925.map.gz | 3.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-38925-v30.xml emd-38925-v30.xml emd-38925.xml emd-38925.xml | 21.9 KB 21.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_38925_fsc.xml emd_38925_fsc.xml | 14.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_38925.png emd_38925.png | 70.8 KB | ||
| Filedesc metadata |  emd-38925.cif.gz emd-38925.cif.gz | 7.4 KB | ||
| その他 |  emd_38925_half_map_1.map.gz emd_38925_half_map_1.map.gz emd_38925_half_map_2.map.gz emd_38925_half_map_2.map.gz | 262.5 MB 262.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-38925 http://ftp.pdbj.org/pub/emdb/structures/EMD-38925 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-38925 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-38925 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_38925_validation.pdf.gz emd_38925_validation.pdf.gz | 752.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_38925_full_validation.pdf.gz emd_38925_full_validation.pdf.gz | 751.7 KB | 表示 | |
| XML形式データ |  emd_38925_validation.xml.gz emd_38925_validation.xml.gz | 20.2 KB | 表示 | |
| CIF形式データ |  emd_38925_validation.cif.gz emd_38925_validation.cif.gz | 25.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-38925 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-38925 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-38925 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-38925 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8y4wMC  8y4xC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_38925.map.gz / 形式: CCP4 / 大きさ: 3.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_38925.map.gz / 形式: CCP4 / 大きさ: 3.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | extracted map around pdb | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half Map B no mask applied
| ファイル | emd_38925_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map B no mask applied | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half Map A no mask applied
| ファイル | emd_38925_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map A no mask applied | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Sialic acid bound form of TRAP transporter in 1:1 complex with it...
| 全体 | 名称: Sialic acid bound form of TRAP transporter in 1:1 complex with its nanobody. |
|---|---|
| 要素 |
|
-超分子 #1: Sialic acid bound form of TRAP transporter in 1:1 complex with it...
| 超分子 | 名称: Sialic acid bound form of TRAP transporter in 1:1 complex with its nanobody. タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  Fusobacterium nucleatum (バクテリア) Fusobacterium nucleatum (バクテリア) |
-分子 #1: N-acetylneuraminate transporter small subunit
| 分子 | 名称: N-acetylneuraminate transporter small subunit / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Fusobacterium nucleatum (バクテリア) Fusobacterium nucleatum (バクテリア) |
| 分子量 | 理論値: 74.198938 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGGSHHHHHH GSWSHPQFEK ASMTGGQQMG RDLYDDDDKD RWGSELEMKV FNKLEEWLGG SLFIGMFVIL VMQIFSRQIF NSPLIWSEE LSRLIFVYVG LLGVSMGIRS QQHIMIDFLY AKFPKSMQKI IFTIIQILIL ACLIFFLYFG YDLFIKKEEI E IVSLGISM ...文字列: MGGSHHHHHH GSWSHPQFEK ASMTGGQQMG RDLYDDDDKD RWGSELEMKV FNKLEEWLGG SLFIGMFVIL VMQIFSRQIF NSPLIWSEE LSRLIFVYVG LLGVSMGIRS QQHIMIDFLY AKFPKSMQKI IFTIIQILIL ACLIFFLYFG YDLFIKKEEI E IVSLGISM KWMYLALPLI TLLMLVRFYQ AYSENYAQNK VYIKPIFILA LMIILVLIAF IKPELFKILK LSNYFDLGEM TI YYVLIAW LVMIFFGVPV GWSLLVACIL YFALTRWKVV YFAADKLVYS LDSFSLLSVP FFILTGILMN GAGITERIFN FAK AMLGHY TGGMGHVNVA ASLIFSGMSG SAIADAGGLG QLEIKAMRDE GYDDDICGGL TAASCIIGPL VPPSISMIIY GVIA NQSIA KLFLAGFVPG FLTTIALMIM NYFVCKKRGY KKTAKASPKE RWIAFKKSFW ALLTPILIIG GIFSGIFTPT EAAVI ATFY SIILGGFIYK ELTVKSFFKH CVEAVAISGV TVLMIMTVTF FGDIIAREQV AMRVAEIFIK YATSPMMVLV MINLLL LFL GMFIDALALQ FLVLPMLIPI AEQVGIDLVF FGVMTTLNMM IGILTPPMGM ALFVVAQVGK MSVSTVAKGV LPFLLPI FI TLVIITIFPQ IILFLPNLIV GG UniProtKB: N-acetylneuraminate transporter small subunit |
-分子 #2: FnTRAP specific nanobody
| 分子 | 名称: FnTRAP specific nanobody / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 14.408753 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: QVQLQESGGG LVQAGGSLRL SCTTSGFNFD DYAIGWFRQA PGKEREGVSC IHCTAYTPYY ARSVRDRFTI SSDNATNTVF LQMNNLRPE DTAVYYCVAD ATRYPYPEFY DYVGQGTQVT VSSHHHHHH |
-分子 #3: SODIUM ION
| 分子 | 名称: SODIUM ION / タイプ: ligand / ID: 3 / コピー数: 3 |
|---|---|
| 分子量 | 理論値: 22.99 Da |
-分子 #4: PHOSPHATIDYLETHANOLAMINE
| 分子 | 名称: PHOSPHATIDYLETHANOLAMINE / タイプ: ligand / ID: 4 / コピー数: 2 / 式: PTY |
|---|---|
| 分子量 | 理論値: 734.039 Da |
| Chemical component information |  ChemComp-PTY: |
-分子 #5: N-acetyl-beta-neuraminic acid
| 分子 | 名称: N-acetyl-beta-neuraminic acid / タイプ: ligand / ID: 5 / コピー数: 1 / 式: SLB |
|---|---|
| 分子量 | 理論値: 309.27 Da |
| Chemical component information | 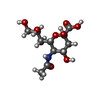 ChemComp-SLB: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.9 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| グリッド | モデル: Quantifoil / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 1 / 実像数: 2341 / 平均露光時間: 60.0 sec. / 平均電子線量: 24.67 e/Å2 / 詳細: Of the 2341 images 1752 were used. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 倍率(補正後): 130841 / 照射モード: OTHER / 撮影モード: OTHER / Cs: 2.7 mm 最大 デフォーカス(公称値): 2.8000000000000003 µm 最小 デフォーカス(公称値): 1.6 µm / 倍率(公称値): 75000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Source name: Other / Chain - Initial model type: experimental model / 詳細: from the apo strcuture of the same protein |
|---|---|
| 精密化 | 空間: REAL / プロトコル: OTHER / 温度因子: 43.2 |
| 得られたモデル |  PDB-8y4w: |
 ムービー
ムービー コントローラー
コントローラー





 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)






































