+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | SpCas9-pegRNA-target DNA complex (pre-initiation) | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | CRISPR-Cas / RNA BINDING PROTEIN-RNA-DNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報maintenance of CRISPR repeat elements / 3'-5' exonuclease activity / DNA endonuclease activity / defense response to virus / 加水分解酵素; エステル加水分解酵素 / DNA binding / RNA binding / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Streptococcus pyogenes (化膿レンサ球菌) / Streptococcus pyogenes (化膿レンサ球菌) /  Streptococcus pyogenes serotype M1 (化膿レンサ球菌) Streptococcus pyogenes serotype M1 (化膿レンサ球菌) | |||||||||
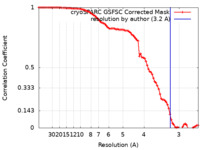
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.2 Å | |||||||||
 データ登録者 データ登録者 | Shuto Y / Nakagawa R / Hoki M / Omura SN / Hirano H / Itoh Y / Nureki O | |||||||||
| 資金援助 |  日本, 1件 日本, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2024 ジャーナル: Nature / 年: 2024タイトル: Structural basis for pegRNA-guided reverse transcription by a prime editor. 著者: Yutaro Shuto / Ryoya Nakagawa / Shiyou Zhu / Mizuki Hoki / Satoshi N Omura / Hisato Hirano / Yuzuru Itoh / Feng Zhang / Osamu Nureki /   要旨: The prime editor system composed of Streptococcus pyogenes Cas9 nickase (nSpCas9) and engineered Moloney murine leukaemia virus reverse transcriptase (M-MLV RT) collaborates with a prime editing ...The prime editor system composed of Streptococcus pyogenes Cas9 nickase (nSpCas9) and engineered Moloney murine leukaemia virus reverse transcriptase (M-MLV RT) collaborates with a prime editing guide RNA (pegRNA) to facilitate a wide variety of precise genome edits in living cells. However, owing to a lack of structural information, the molecular mechanism of pegRNA-guided reverse transcription by the prime editor remains poorly understood. Here we present cryo-electron microscopy structures of the SpCas9-M-MLV RTΔRNaseH-pegRNA-target DNA complex in multiple states. The termination structure, along with our functional analysis, reveals that M-MLV RT extends reverse transcription beyond the expected site, resulting in scaffold-derived incorporations that cause undesired edits at the target loci. Furthermore, structural comparisons among the pre-initiation, initiation and elongation states show that M-MLV RT remains in a consistent position relative to SpCas9 during reverse transcription, whereas the pegRNA-synthesized DNA heteroduplex builds up along the surface of SpCas9. On the basis of our structural insights, we rationally engineered pegRNA variants and prime-editor variants in which M-MLV RT is fused within SpCas9. Collectively, our findings provide structural insights into the stepwise mechanism of prime editing, and will pave the way for the development of a versatile prime editing toolbox. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_37860.map.gz emd_37860.map.gz | 9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-37860-v30.xml emd-37860-v30.xml emd-37860.xml emd-37860.xml | 18.2 KB 18.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_37860_fsc.xml emd_37860_fsc.xml | 8.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_37860.png emd_37860.png | 90.8 KB | ||
| マスクデータ |  emd_37860_msk_1.map emd_37860_msk_1.map | 10 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-37860.cif.gz emd-37860.cif.gz | 6.8 KB | ||
| その他 |  emd_37860_half_map_1.map.gz emd_37860_half_map_1.map.gz emd_37860_half_map_2.map.gz emd_37860_half_map_2.map.gz | 9.2 MB 9.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-37860 http://ftp.pdbj.org/pub/emdb/structures/EMD-37860 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37860 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37860 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_37860_validation.pdf.gz emd_37860_validation.pdf.gz | 769.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_37860_full_validation.pdf.gz emd_37860_full_validation.pdf.gz | 769.2 KB | 表示 | |
| XML形式データ |  emd_37860_validation.xml.gz emd_37860_validation.xml.gz | 13.3 KB | 表示 | |
| CIF形式データ |  emd_37860_validation.cif.gz emd_37860_validation.cif.gz | 17.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37860 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37860 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37860 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37860 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_37860.map.gz / 形式: CCP4 / 大きさ: 10 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_37860.map.gz / 形式: CCP4 / 大きさ: 10 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.2969 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_37860_msk_1.map emd_37860_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_37860_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_37860_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : SpCas9-pegRNA-target DNA complex
| 全体 | 名称: SpCas9-pegRNA-target DNA complex |
|---|---|
| 要素 |
|
-超分子 #1: SpCas9-pegRNA-target DNA complex
| 超分子 | 名称: SpCas9-pegRNA-target DNA complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#5 |
|---|---|
| 由来(天然) | 生物種:  Streptococcus pyogenes (化膿レンサ球菌) Streptococcus pyogenes (化膿レンサ球菌) |
-分子 #1: CRISPR-associated endonuclease Cas9/Csn1
| 分子 | 名称: CRISPR-associated endonuclease Cas9/Csn1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Streptococcus pyogenes serotype M1 (化膿レンサ球菌) Streptococcus pyogenes serotype M1 (化膿レンサ球菌) |
| 分子量 | 理論値: 158.457578 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: DKKYSIGLAI GTNSVGWAVI TDEYKVPSKK FKVLGNTDRH SIKKNLIGAL LFDSGETAEA TRLKRTARRR YTRRKNRICY LQEIFSNEM AKVDDSFFHR LEESFLVEED KKHERHPIFG NIVDEVAYHE KYPTIYHLRK KLVDSTDKAD LRLIYLALAH M IKFRGHFL ...文字列: DKKYSIGLAI GTNSVGWAVI TDEYKVPSKK FKVLGNTDRH SIKKNLIGAL LFDSGETAEA TRLKRTARRR YTRRKNRICY LQEIFSNEM AKVDDSFFHR LEESFLVEED KKHERHPIFG NIVDEVAYHE KYPTIYHLRK KLVDSTDKAD LRLIYLALAH M IKFRGHFL IEGDLNPDNS DVDKLFIQLV QTYNQLFEEN PINASGVDAK AILSARLSKS RRLENLIAQL PGEKKNGLFG NL IALSLGL TPNFKSNFDL AEDAKLQLSK DTYDDDLDNL LAQIGDQYAD LFLAAKNLSD AILLSDILRV NTEITKAPLS ASM IKRYDE HHQDLTLLKA LVRQQLPEKY KEIFFDQSKN GYAGYIDGGA SQEEFYKFIK PILEKMDGTE ELLVKLNRED LLRK QRTFD NGSIPHQIHL GELHAILRRQ EDFYPFLKDN REKIEKILTF RIPYYVGPLA RGNSRFAWMT RKSEETITPW NFEEV VDKG ASAQSFIERM TNFDKNLPNE KVLPKHSLLY EYFTVYNELT KVKYVTEGMR KPAFLSGEQK KAIVDLLFKT NRKVTV KQL KEDYFKKIEC FDSVEISGVE DRFNASLGTY HDLLKIIKDK DFLDNEENED ILEDIVLTLT LFEDREMIEE RLKTYAH LF DDKVMKQLKR RRYTGWGRLS RKLINGIRDK QSGKTILDFL KSDGFANRNF MQLIHDDSLT FKEDIQKAQV SGQGDSLH E HIANLAGSPA IKKGILQTVK VVDELVKVMG RHKPENIVIE MARENQTTQK GQKNSRERMK RIEEGIKELG SQILKEHPV ENTQLQNEKL YLYYLQNGRD MYVDQELDIN RLSDYDVDAI VPQSFLKDDS IDNKVLTRSD KNRGKSDNVP SEEVVKKMKN YWRQLLNAK LITQRKFDNL TKAERGGLSE LDKAGFIKRQ LVETRQITKH VAQILDSRMN TKYDENDKLI REVKVITLKS K LVSDFRKD FQFYKVREIN NYHHAHDAYL NAVVGTALIK KYPKLESEFV YGDYKVYDVR KMIAKSEQEI GKATAKYFFY SN IMNFFKT EITLANGEIR KRPLIETNGE TGEIVWDKGR DFATVRKVLS MPQVNIVKKT EVQTGGFSKE SILPKRNSDK LIA RKKDWD PKKYGGFDSP TVAYSVLVVA KVEKGKSKKL KSVKELLGIT IMERSSFEKN PIDFLEAKGY KEVKKDLIIK LPKY SLFEL ENGRKRMLAS AGELQKGNEL ALPSKYVNFL YLASHYEKLK GSPEDNEQKQ LFVEQHKHYL DEIIEQISEF SKRVI LADA NLDKVLSAYN KHRDKPIREQ AENIIHLFTL TNLGAPAAFK YFDTTIDRKR YTSTKEVLDA TLIHQSITGL YETRID LSQ LGGD UniProtKB: CRISPR-associated endonuclease Cas9/Csn1 |
-分子 #2: DNA (51-MER)
| 分子 | 名称: DNA (51-MER) / タイプ: dna / ID: 2 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Streptococcus pyogenes (化膿レンサ球菌) Streptococcus pyogenes (化膿レンサ球菌) |
| 分子量 | 理論値: 15.636033 KDa |
| 配列 | 文字列: (DC)(DT)(DA)(DG)(DT)(DA)(DC)(DT)(DC)(DT) (DG)(DC)(DC)(DA)(DT)(DC)(DA)(DG)(DA)(DG) (DC)(DA)(DA)(DG)(DC)(DA)(DC)(DT)(DA) (DC)(DG)(DG)(DC)(DC)(DG)(DA)(DT)(DT)(DG) (DC) (DT)(DC)(DT)(DA)(DA) ...文字列: (DC)(DT)(DA)(DG)(DT)(DA)(DC)(DT)(DC)(DT) (DG)(DC)(DC)(DA)(DT)(DC)(DA)(DG)(DA)(DG) (DC)(DA)(DA)(DG)(DC)(DA)(DC)(DT)(DA) (DC)(DG)(DG)(DC)(DC)(DG)(DA)(DT)(DT)(DG) (DC) (DT)(DC)(DT)(DA)(DA)(DG)(DT)(DG) (DA)(DT)(DC) |
-分子 #3: DNA (5'-D(*TP*GP*AP*TP*GP*GP*CP*AP*GP*AP*GP*TP*AP*CP*TP*AP*G)-3')
| 分子 | 名称: DNA (5'-D(*TP*GP*AP*TP*GP*GP*CP*AP*GP*AP*GP*TP*AP*CP*TP*AP*G)-3') タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Streptococcus pyogenes (化膿レンサ球菌) Streptococcus pyogenes (化膿レンサ球菌) |
| 分子量 | 理論値: 5.291446 KDa |
| 配列 | 文字列: (DT)(DG)(DA)(DT)(DG)(DG)(DC)(DA)(DG)(DA) (DG)(DT)(DA)(DC)(DT)(DA)(DG) |
-分子 #5: DNA (34-MER)
| 分子 | 名称: DNA (34-MER) / タイプ: dna / ID: 5 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Streptococcus pyogenes (化膿レンサ球菌) Streptococcus pyogenes (化膿レンサ球菌) |
| 分子量 | 理論値: 10.462743 KDa |
| 配列 | 文字列: (DG)(DA)(DT)(DC)(DA)(DC)(DT)(DT)(DA)(DG) (DA)(DG)(DC)(DA)(DA)(DT)(DC)(DG)(DG)(DC) (DC)(DC)(DA)(DG)(DA)(DC)(DT)(DG)(DA) (DG)(DC)(DA)(DC)(DG) |
-分子 #4: RNA (137-MER)
| 分子 | 名称: RNA (137-MER) / タイプ: rna / ID: 4 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  Streptococcus pyogenes (化膿レンサ球菌) Streptococcus pyogenes (化膿レンサ球菌) |
| 分子量 | 理論値: 44.300379 KDa |
| 配列 | 文字列: GGCCGUAGUG CUUGCUCUGA GUUUUAGAGC UAGAAAUAGC AAGUUAAAAU AAGGCUAGUC CGUUAUCAAC UUGAAAAAGU GGCACCGAG UCGGUGCAGG AGGAAGCAGU GCAACCAAAC CACAGCGUGC UCAGUCUG |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 297 K |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.6 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)