+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of a bacterial protein | |||||||||
 マップデータ マップデータ | main map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | bacterial immune proteins / IMMUNE SYSTEM | |||||||||
| 生物種 |  Paenibacillus sp. 453mf (バクテリア) Paenibacillus sp. 453mf (バクテリア) | |||||||||
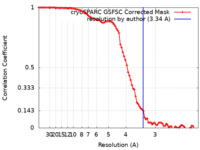
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.34 Å | |||||||||
 データ登録者 データ登録者 | Yu G / Liao F / Li X / Li Z / Zhang H | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Cryo-EM structure of a bacterial protein 著者: Yu G / Liao F / Li X / Li Z / Zhang H | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_37592.map.gz emd_37592.map.gz | 59.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-37592-v30.xml emd-37592-v30.xml emd-37592.xml emd-37592.xml | 16.9 KB 16.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_37592_fsc.xml emd_37592_fsc.xml | 8.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_37592.png emd_37592.png | 197.6 KB | ||
| Filedesc metadata |  emd-37592.cif.gz emd-37592.cif.gz | 6.1 KB | ||
| その他 |  emd_37592_half_map_1.map.gz emd_37592_half_map_1.map.gz emd_37592_half_map_2.map.gz emd_37592_half_map_2.map.gz | 59.4 MB 59.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-37592 http://ftp.pdbj.org/pub/emdb/structures/EMD-37592 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37592 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37592 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_37592_validation.pdf.gz emd_37592_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_37592_full_validation.pdf.gz emd_37592_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_37592_validation.xml.gz emd_37592_validation.xml.gz | 16.6 KB | 表示 | |
| CIF形式データ |  emd_37592_validation.cif.gz emd_37592_validation.cif.gz | 21.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37592 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37592 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37592 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37592 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_37592.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_37592.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | main map | ||||||||||||||||||||||||||||||||||||
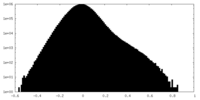
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.07578 Å | ||||||||||||||||||||||||||||||||||||
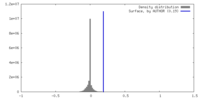
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: half map
| ファイル | emd_37592_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
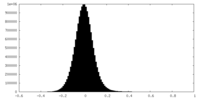
| 密度ヒストグラム |
-ハーフマップ: half map
| ファイル | emd_37592_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
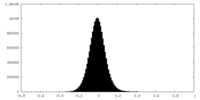
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cryo-EM structure of a bacterial protein
| 全体 | 名称: Cryo-EM structure of a bacterial protein |
|---|---|
| 要素 |
|
-超分子 #1: Cryo-EM structure of a bacterial protein
| 超分子 | 名称: Cryo-EM structure of a bacterial protein / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Paenibacillus sp. 453mf (バクテリア) Paenibacillus sp. 453mf (バクテリア) |
| 分子量 | 理論値: 400 KDa |
-分子 #1: Helicase HerA central domain-containing protein
| 分子 | 名称: Helicase HerA central domain-containing protein / タイプ: protein_or_peptide / ID: 1 / 詳細: WP_091160506.1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Paenibacillus sp. 453mf (バクテリア) Paenibacillus sp. 453mf (バクテリア) |
| 分子量 | 理論値: 77.626344 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MIGVNRMTEA STYIGTVQDV NGANIRVVLD INTISSLKFV DGQGYRIGQI GSFVRIPIGY INLFGIVSQV GAGAVPDKLL EVEPYGHRW ISVQLVGEEG IKKEFERGVS QYPTIGDKVH IVTEPDLKKI YGTQNKKYIS LGNIASVDSI PALVNIDTLV T RHSAVLGS ...文字列: MIGVNRMTEA STYIGTVQDV NGANIRVVLD INTISSLKFV DGQGYRIGQI GSFVRIPIGY INLFGIVSQV GAGAVPDKLL EVEPYGHRW ISVQLVGEEG IKKEFERGVS QYPTIGDKVH IVTEPDLKKI YGTQNKKYIS LGNIASVDSI PALVNIDTLV T RHSAVLGS TGSGKSTTVT SILQRISDMS QFPSARIIVF DIHGEYAAAF KGKAKVYKVT PSNNELKLSI PYWALTCDEF LS VAFGGLE GSGRNALIDK IYELKLQTLK RQEYEGINED SLTVDTPIPF SIHKLWFDLY RAEISTHYVQ GSHSEENEAL LLG EDGNPV QKGDSLKVVP PIYMPHTQAQ GATKIYLSNR GKNIRKPLEG LASLLKDPRY EFLFNADDWS VNLDGKTNKD LDAL LETWV GSEESISIFD LSGMPSSILD TLIGILIRIL YDSLFWSRNQ PEGGRERPLL VVLEEAHTYL GKDSRGIAID GVRKI VKEG RKYGIGMMLV SQRPSEIDST ILSQCGTLFA LRMNNSSDRN HVLGAVSDSF EGLMGMLPTL RTGEAIIIGE SVRLPM RTI ISPPPFGRRP DSLDPDVTAK WSNNRVQGDY KEVLTLWRQK KVRSQRIVEN IKRLPVVNEG EMTDMVREMV TSSNILS IG YEADSMTLEI EFNHGLVYQY YDVPETLHTE LLAAESHGKF FNSQIKNNYR FSRI |
-分子 #2: PHOSPHOTHIOPHOSPHORIC ACID-ADENYLATE ESTER
| 分子 | 名称: PHOSPHOTHIOPHOSPHORIC ACID-ADENYLATE ESTER / タイプ: ligand / ID: 2 / コピー数: 5 / 式: AGS |
|---|---|
| 分子量 | 理論値: 523.247 Da |
| Chemical component information |  ChemComp-AGS: |
-分子 #3: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 3 / コピー数: 5 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 検出モード: SUPER-RESOLUTION / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)