+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of CUL3-RBX1-KLHL22 complex without CUL3 NA motif | |||||||||||||||
 マップデータ マップデータ | ||||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | Cullin Ring E3 ubiquitin ligase / LIGASE | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of mitotic cell cycle phase transition / trophectodermal cellular morphogenesis / liver morphogenesis / POZ domain binding / nuclear protein quality control by the ubiquitin-proteasome system / polar microtubule / regulation protein catabolic process at postsynapse / COPII vesicle coating / anaphase-promoting complex-dependent catabolic process / cullin-RING-type E3 NEDD8 transferase ...positive regulation of mitotic cell cycle phase transition / trophectodermal cellular morphogenesis / liver morphogenesis / POZ domain binding / nuclear protein quality control by the ubiquitin-proteasome system / polar microtubule / regulation protein catabolic process at postsynapse / COPII vesicle coating / anaphase-promoting complex-dependent catabolic process / cullin-RING-type E3 NEDD8 transferase / NEDD8 transferase activity / cellular response to L-leucine / cullin-RING ubiquitin ligase complex / RHOBTB3 ATPase cycle / positive regulation of T cell mediated immune response to tumor cell / positive regulation of mitotic metaphase/anaphase transition / embryonic cleavage / cellular response to chemical stress / Cul7-RING ubiquitin ligase complex / ubiquitin-dependent protein catabolic process via the C-end degron rule pathway / cell projection organization / Loss of Function of FBXW7 in Cancer and NOTCH1 Signaling / positive regulation of protein autoubiquitination / RNA polymerase II transcription initiation surveillance / protein neddylation / Notch binding / NEDD8 ligase activity / fibroblast apoptotic process / VCB complex / RHOBTB1 GTPase cycle / negative regulation of response to oxidative stress / Cul5-RING ubiquitin ligase complex / SCF ubiquitin ligase complex / Cul2-RING ubiquitin ligase complex / negative regulation of type I interferon production / stem cell division / ubiquitin-ubiquitin ligase activity / SCF-dependent proteasomal ubiquitin-dependent protein catabolic process / Cul4A-RING E3 ubiquitin ligase complex / Cul4-RING E3 ubiquitin ligase complex / mitotic metaphase chromosome alignment / Cul3-RING ubiquitin ligase complex / Cul4B-RING E3 ubiquitin ligase complex / stress fiber assembly / mitotic spindle assembly checkpoint signaling / ubiquitin ligase complex scaffold activity / negative regulation of Rho protein signal transduction / negative regulation of mitophagy / Prolactin receptor signaling / positive regulation of cytokinesis / cullin family protein binding / mitotic sister chromatid segregation / protein monoubiquitination / endoplasmic reticulum to Golgi vesicle-mediated transport / sperm flagellum / RHOBTB2 GTPase cycle / ubiquitin-like ligase-substrate adaptor activity / protein K48-linked ubiquitination / protein autoubiquitination / Nuclear events stimulated by ALK signaling in cancer / intercellular bridge / gastrulation / 14-3-3 protein binding / transcription-coupled nucleotide-excision repair / positive regulation of TORC1 signaling / regulation of cellular response to insulin stimulus / intrinsic apoptotic signaling pathway / negative regulation of insulin receptor signaling pathway / post-translational protein modification / negative regulation of autophagy / cyclin binding / Regulation of BACH1 activity / T cell activation / positive regulation of protein ubiquitination / integrin-mediated signaling pathway / cellular response to amino acid stimulus / Degradation of DVL / kidney development / Degradation of GLI1 by the proteasome / GSK3B and BTRC:CUL1-mediated-degradation of NFE2L2 / Recognition of DNA damage by PCNA-containing replication complex / Negative regulation of NOTCH4 signaling / Vif-mediated degradation of APOBEC3G / Hedgehog 'on' state / G1/S transition of mitotic cell cycle / negative regulation of canonical Wnt signaling pathway / Degradation of GLI2 by the proteasome / GLI3 is processed to GLI3R by the proteasome / FBXL7 down-regulates AURKA during mitotic entry and in early mitosis / DNA Damage Recognition in GG-NER / protein destabilization / Degradation of beta-catenin by the destruction complex / RING-type E3 ubiquitin transferase / Oxygen-dependent proline hydroxylation of Hypoxia-inducible Factor Alpha / positive regulation of T cell activation / Evasion by RSV of host interferon responses / NOTCH1 Intracellular Domain Regulates Transcription / Dual Incision in GG-NER / Transcription-Coupled Nucleotide Excision Repair (TC-NER) / Formation of TC-NER Pre-Incision Complex 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||
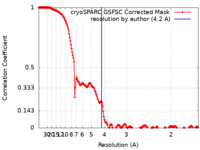
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.2 Å | |||||||||||||||
 データ登録者 データ登録者 | Wang W / Ling L / Dai Z / Zuo P / Yin Y | |||||||||||||||
| 資金援助 |  中国, 4件 中国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: A conserved N-terminal motif of CUL3 contributes to assembly and E3 ligase activity of CRL3. 著者: Weize Wang / Ling Liang / Zonglin Dai / Peng Zuo / Shang Yu / Yishuo Lu / Dian Ding / Hongyi Chen / Hui Shan / Yan Jin / Youdong Mao / Yuxin Yin /  要旨: The CUL3-RING E3 ubiquitin ligases (CRL3s) play an essential role in response to extracellular nutrition and stress stimuli. The ubiquitin ligase function of CRL3s is activated through dimerization. ...The CUL3-RING E3 ubiquitin ligases (CRL3s) play an essential role in response to extracellular nutrition and stress stimuli. The ubiquitin ligase function of CRL3s is activated through dimerization. However, how and why such a dimeric assembly is required for its ligase activity remains elusive. Here, we report the cryo-EM structure of the dimeric CRL3 complex and reveal a conserved N-terminal motif in CUL3 that contributes to the dimerization assembly and the E3 ligase activity of CRL3. We show that deletion of the CUL3 N-terminal motif impairs dimeric assembly and the E3 ligase activity of both CRL3 and several other CRL3s. In addition, we found that the dynamics of dimeric assembly of CRL3 generates a variable ubiquitination zone, potentially facilitating substrate recognition and ubiquitination. These findings demonstrate that a CUL3 N-terminal motif participates in the assembly process and provide insights into the assembly and activation of CRL3s. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_36987.map.gz emd_36987.map.gz | 453.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-36987-v30.xml emd-36987-v30.xml emd-36987.xml emd-36987.xml | 22 KB 22 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_36987_fsc.xml emd_36987_fsc.xml | 17 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_36987.png emd_36987.png | 42.6 KB | ||
| Filedesc metadata |  emd-36987.cif.gz emd-36987.cif.gz | 6.4 KB | ||
| その他 |  emd_36987_additional_1.map.gz emd_36987_additional_1.map.gz emd_36987_half_map_1.map.gz emd_36987_half_map_1.map.gz emd_36987_half_map_2.map.gz emd_36987_half_map_2.map.gz | 483.7 MB 474.8 MB 474.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-36987 http://ftp.pdbj.org/pub/emdb/structures/EMD-36987 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36987 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36987 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_36987_validation.pdf.gz emd_36987_validation.pdf.gz | 995 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_36987_full_validation.pdf.gz emd_36987_full_validation.pdf.gz | 994.6 KB | 表示 | |
| XML形式データ |  emd_36987_validation.xml.gz emd_36987_validation.xml.gz | 26.1 KB | 表示 | |
| CIF形式データ |  emd_36987_validation.cif.gz emd_36987_validation.cif.gz | 34.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36987 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36987 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36987 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36987 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8k9iMC  8k8tC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_36987.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_36987.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.821 Å | ||||||||||||||||||||||||||||||||||||
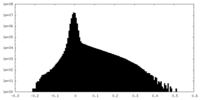
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Raw map of CUL3(NA motif deletion)-RBX1-KLHL22 complex
| ファイル | emd_36987_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Raw map of CUL3(NA motif deletion)-RBX1-KLHL22 complex | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
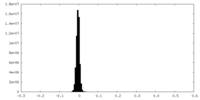
| 密度ヒストグラム |
-ハーフマップ: half map B
| ファイル | emd_36987_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
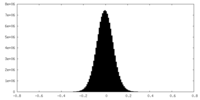
| 密度ヒストグラム |
-ハーフマップ: half map A
| ファイル | emd_36987_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
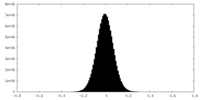
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : CUL3-RBX1-KLHL22 complex without CUL3 NA motif
| 全体 | 名称: CUL3-RBX1-KLHL22 complex without CUL3 NA motif |
|---|---|
| 要素 |
|
-超分子 #1: CUL3-RBX1-KLHL22 complex without CUL3 NA motif
| 超分子 | 名称: CUL3-RBX1-KLHL22 complex without CUL3 NA motif / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Cullin-3
| 分子 | 名称: Cullin-3 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 87.432312 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MDEKYVNSIW DLLKNAIQEI QRKNNSGLSF EELYRNAYTM VLHKHGEKLY TGLREVVTEH LINKVREDVL NSLNNNFLQT LNQAWNDHQ TAMVMIRDIL MYMDRVYVQQ NNVENVYNLG LIIFRDQVVR YGCIRDHLRQ TLLDMIARER KGEVVDRGAI R NACQMLMI ...文字列: MDEKYVNSIW DLLKNAIQEI QRKNNSGLSF EELYRNAYTM VLHKHGEKLY TGLREVVTEH LINKVREDVL NSLNNNFLQT LNQAWNDHQ TAMVMIRDIL MYMDRVYVQQ NNVENVYNLG LIIFRDQVVR YGCIRDHLRQ TLLDMIARER KGEVVDRGAI R NACQMLMI LGLEGRSVYE EDFEAPFLEM SAEFFQMESQ KFLAENSASV YIKKVEARIN EEIERVMHCL DKSTEEPIVK VV ERELISK HMKTIVEMEN SGLVHMLKNG KTEDLGCMYK LFSRVPNGLK TMCECMSSYL REQGKALVSE EGEGKNPVDY IQG LLDLKS RFDRFLLESF NNDRLFKQTI AGDFEYFLNL NSRSPEYLSL FIDDKLKKGV KGLTEQEVET ILDKAMVLFR FMQE KDVFE RYYKQHLARR LLTNKSVSDD SEKNMISKLK TECGCQFTSK LEGMFRDMSI SNTTMDEFRQ HLQATGVSLG GVDLT VRVL TTGYWPTQSA TPKCNIPPAP RHAFEIFRRF YLAKHSGRQL TLQHHMGSAD LNATFYGPVK KEDGSEVGVG GAQVTG SNT RKHILQVSTF QMTILMLFNN REKYTFEEIQ QETDIPEREL VRALQSLACG KPTQRVLTKE PKSKEIENGH IFTVNDQ FT SKLHRVKIQT VAAKQGESDP ERKETRQKVD DDRKHEIEAA IVRIMKSRKK MQHNVLVAEV TQQLKARFLP SPVVIKKR I EGLIEREYLA RTPEDRKVYT YVAKLHHHHH H UniProtKB: Cullin-3 |
-分子 #2: Kelch-like protein 22
| 分子 | 名称: Kelch-like protein 22 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 20.36534 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAEEQEFTQL CKLPAQPSHP HCVNNTYRSA QHSQALLRGL LALRDSGILF DVVLVVEGRH IEAHRILLAA SCDYFRGMFA GGLKEMEQE EVLIHGVSYN AMCQILHFIY TSELELSLSN VQETLVAACQ LQIPEIIHFC CDFLMSWVDE ENILDVYRLA E LFDLSRLT EQLDTYILKN UniProtKB: Kelch-like protein 22 |
-分子 #3: E3 ubiquitin-protein ligase RBX1, N-terminally processed
| 分子 | 名称: E3 ubiquitin-protein ligase RBX1, N-terminally processed タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 12.289977 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAAAMDVDTP SGTNSGAGKK RFEVKKWNAV ALWAWDIVVD NCAICRNHIM DLCIECQANQ ASATSEECTV AWGVCNHAFH FHCISRWLK TRQVCPLDNR EWEFQKYGH UniProtKB: E3 ubiquitin-protein ligase RBX1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.00 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: DIFFRACTION / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)