+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of a 2,3-hydroxycinnamic acid 1,2-dioxygenase MhpB in apo form | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | dioxygenase / BIOSYNTHETIC PROTEIN | |||||||||
| 機能・相同性 | 3-carboxyethylcatechol 2,3-dioxygenase / 3-carboxyethylcatechol 2,3-dioxygenase activity / 2,3-dihydroxyphenylpropionate/2,3-dihydroxicinnamic acid 1,2-dioxygenase / Extradiol ring-cleavage dioxygenase, class III enzyme, subunit B / Catalytic LigB subunit of aromatic ring-opening dioxygenase / 3-phenylpropionate catabolic process / ferrous iron binding / 2,3-dihydroxyphenylpropionate/2,3-dihydroxicinnamic acid 1,2-dioxygenase 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.72 Å | |||||||||
 データ登録者 データ登録者 | Jiang WX / Cheng XQ / Ma LX / Xing Q | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: J Hazard Mater / 年: 2025 ジャーナル: J Hazard Mater / 年: 2025タイトル: Structural and catalytic insights into MhpB: A dioxygenase enzyme for degrading catecholic pollutants. 著者: Xu Dong / Manli Xu / Miao Wu / Ying Wang / Xiaoqi Cheng / Wenxue Jiang / Dule Zheng / Ahmed Habiba Omar / Yibin Cheng / Aitao Li / Lixin Ma / Qiong Xing /  要旨: The increasing environmental pollution from persistent aromatic compounds requires effective biodegradation strategies. In this study, we focused on MhpB, an extradiol dioxygenase (EDO) from ...The increasing environmental pollution from persistent aromatic compounds requires effective biodegradation strategies. In this study, we focused on MhpB, an extradiol dioxygenase (EDO) from Escherichia coli. It is known for its role in the degradation of catechols, key intermediates in the degradation of aromatic compounds. We report the high-resolution structure of MhpB determined by cryo-electron microscopy, revealing a decameric conformation with the catalytic chamber at the side. The structure-based analysis allowed us to investigate the substrate-enzyme interaction and the substrate selectivity, which are crucial for its catalytic function. Site-directed mutagenesis was used to modulate the in vitro and in vivo substrate preference of MhpB, enhancing its potential for industrial applications in pollutant degradation. The study provides insight into the mechanism of the enzyme and paves the way for the development of engineered EDOs for environmental remediation of aromatic pollutants. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_36757.map.gz emd_36757.map.gz | 116.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-36757-v30.xml emd-36757-v30.xml emd-36757.xml emd-36757.xml | 13.5 KB 13.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_36757.png emd_36757.png | 57 KB | ||
| Filedesc metadata |  emd-36757.cif.gz emd-36757.cif.gz | 6.5 KB | ||
| その他 |  emd_36757_half_map_1.map.gz emd_36757_half_map_1.map.gz emd_36757_half_map_2.map.gz emd_36757_half_map_2.map.gz | 226.2 MB 226.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-36757 http://ftp.pdbj.org/pub/emdb/structures/EMD-36757 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36757 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36757 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_36757_validation.pdf.gz emd_36757_validation.pdf.gz | 840.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_36757_full_validation.pdf.gz emd_36757_full_validation.pdf.gz | 840.4 KB | 表示 | |
| XML形式データ |  emd_36757_validation.xml.gz emd_36757_validation.xml.gz | 16 KB | 表示 | |
| CIF形式データ |  emd_36757_validation.cif.gz emd_36757_validation.cif.gz | 19 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36757 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36757 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36757 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36757 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8k04MC  9ktiC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_36757.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_36757.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.56667 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_36757_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
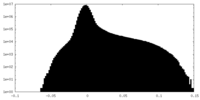
| 投影像・断面図 |
| ||||||||||||
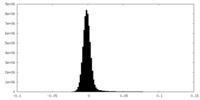
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_36757_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
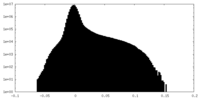
| 投影像・断面図 |
| ||||||||||||
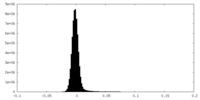
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Homohexamer of inositol phosphate phosphatase SopB
| 全体 | 名称: Homohexamer of inositol phosphate phosphatase SopB |
|---|---|
| 要素 |
|
-超分子 #1: Homohexamer of inositol phosphate phosphatase SopB
| 超分子 | 名称: Homohexamer of inositol phosphate phosphatase SopB / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: 2,3-dihydroxyphenylpropionate/2,3-dihydroxicinnamic acid 1,2-diox...
| 分子 | 名称: 2,3-dihydroxyphenylpropionate/2,3-dihydroxicinnamic acid 1,2-dioxygenase タイプ: protein_or_peptide / ID: 1 / コピー数: 10 / 光学異性体: LEVO / EC番号: 3-carboxyethylcatechol 2,3-dioxygenase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 34.230961 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MHAYLHCLSH SPLVGYVDPA QEVLDEVNGV IASARERIAA FSPELVVLFA PDHYNGFFYD VMPPFCLGVG ATAIGDFGSA AGELPVPVE LAEACAHAVM KSGIDLAVSY CMQVDHGFAQ PLEFLLGGLD KVPVLPVFIN GVATPLPGFQ RTRMLGEAIG R FTSTLNKR ...文字列: MHAYLHCLSH SPLVGYVDPA QEVLDEVNGV IASARERIAA FSPELVVLFA PDHYNGFFYD VMPPFCLGVG ATAIGDFGSA AGELPVPVE LAEACAHAVM KSGIDLAVSY CMQVDHGFAQ PLEFLLGGLD KVPVLPVFIN GVATPLPGFQ RTRMLGEAIG R FTSTLNKR VLFLGSGGLS HQPPVPELAK ADAHMRDRLL GSGKDLPASE RELRQQRVIS AAEKFVEDQR TLHPLNPIWD NQ FMTLLEQ GRIQELDAVS NEELSAIAGK STHEIKTWVA AFAAISAFGN WRSEGRYYRP IPEWIAGFGS LSARTEN UniProtKB: 2,3-dihydroxyphenylpropionate/2,3-dihydroxicinnamic acid 1,2-dioxygenase |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.2 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 39.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: DIFFRACTION / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 2.72 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 364814 |
| 初期 角度割当 | タイプ: NOT APPLICABLE |
| 最終 角度割当 | タイプ: NOT APPLICABLE |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)